Kurs
Code aus diesem Tutorial online ausführen und bearbeiten
Code ausführenDie Hauptkomponentenanalyse (PCA) ist ein Verfahren zur linearen Dimensionalitätsreduktion, mit dem man Informationen aus einem hochdimensionalen Raum extrahieren kann, indem man sie in einen niedrigdimensionalen Unterraum projiziert. Wenn du mit der Sprache der linearen Algebra vertraut bist, könntest du auch sagen, dass die Hauptkomponentenanalyse darin besteht, die Eigenvektoren der Kovarianzmatrix zu finden, um die Richtungen der maximalen Varianz in den Daten zu identifizieren.
Ein wichtiger Punkt bei der PCA ist, dass es sich um eine unüberwachte Dimensionalitätsreduktion handelt, d.h. du kannst ähnliche Datenpunkte auf der Grundlage ihrer Korrelation clustern, ohne dass sie überwacht werden (oder beschriftet sind).
Hinweis: Merkmale, Dimensionen und Variablen beziehen sich alle auf dieselbe Sache. Du wirst feststellen, dass sie austauschbar verwendet werden.
Wo kannst du PCA anwenden?
-
Datenvisualisierung: Bei der Arbeit an einem datenbezogenen Problem besteht die Herausforderung in der heutigen Welt in der schieren Menge an Daten und den Variablen/Merkmalen, die diese Daten definieren. Um ein Problem zu lösen, bei dem Daten der Schlüssel sind, brauchst du eine umfangreiche Datenexploration, z. B. um herauszufinden, wie die Variablen korreliert sind oder um die Verteilung einiger Variablen zu verstehen. Wenn man bedenkt, dass es eine große Anzahl von Variablen oder Dimensionen gibt, entlang derer die Daten verteilt sind, kann die Visualisierung eine Herausforderung darstellen und fast unmöglich sein.
Das kann die PCA für dich tun, denn sie projiziert die Daten in eine niedrigere Dimension und ermöglicht es dir so, die Daten mit bloßem Auge in einem 2D- oder 3D-Raum zu visualisieren.
-
Einen Machine Learning (ML) Algorithmus beschleunigen: Da die Hauptidee der PCA die Dimensionalitätsreduktion ist, kannst du sie nutzen, um die Trainings- und Testzeit deines Algorithmus für maschinelles Lernen zu verkürzen, wenn deine Daten viele Merkmale haben und der ML-Algorithmus zu langsam lernt.
Auf einer abstrakten Ebene nimmst du einen Datensatz mit vielen Merkmalen und vereinfachst diesen Datensatz, indem du einige Principal Components aus den ursprünglichen Merkmalen auswählst.
Was ist eine Hauptkomponente?
Hauptkomponenten sind der Schlüssel zur PCA; sie stellen dar, was sich unter der Haube deiner Daten befindet. Laienhaft ausgedrückt: Wenn die Daten von einem höheren Raum in eine niedrigere Dimension (nehmen wir drei Dimensionen an) projiziert werden, sind die drei Dimensionen nichts anderes als die drei Hauptkomponenten, die den größten Teil der Varianz (Information) deiner Daten einfangen (oder enthalten).
Hauptkomponenten haben sowohl eine Richtung als auch eine Größe. Die Richtung gibt an, über welche Hauptachsen die Daten am stärksten verteilt sind oder die größte Varianz aufweisen, und die Größe gibt an, wie viel Varianz die Hauptkomponente der Daten erfasst, wenn sie auf diese Achse projiziert wird. Die Hauptkomponenten sind eine gerade Linie, und die erste Hauptkomponente enthält die größte Varianz in den Daten. Jede weitere Hauptkomponente ist orthogonal zur letzten und hat eine geringere Varianz. Auf diese Weise erhältst du bei einer Menge von x korrelierten Variablen über y Stichproben eine Menge von u unkorrelierten Hauptkomponenten über die gleichen y Stichproben.
Der Grund dafür, dass du aus den ursprünglichen Merkmalen unkorrelierte Hauptkomponenten erhältst, liegt darin, dass die korrelierten Merkmale zur gleichen Hauptkomponente beitragen und dadurch die ursprünglichen Datenmerkmale in unkorrelierte Hauptkomponenten zerlegt werden, die jeweils einen anderen Satz korrelierter Merkmale mit unterschiedlichem Ausmaß an Variabilität darstellen. Jede Hauptkomponente steht für einen bestimmten Prozentsatz der Gesamtvariabilität, die von den Daten erfasst wird.
In der heutigen Schulung werden wir PCA anwenden, um durch Datenvisualisierung Erkenntnisse zu gewinnen, und wir werden PCA auch anwenden, um unseren Algorithmus für maschinelles Lernen zu beschleunigen. Um die beiden oben genannten Aufgaben zu erfüllen, wirst du zwei bekannte Datensätze verwenden: Brustkrebs und CIFAR - 10. Der erste ist ein numerischer Datensatz, der zweite ist ein Bilddatensatz.
Die Daten verstehen
Bevor du loslegst und die Daten lädst, ist es gut, wenn du die Daten, mit denen du arbeiten wirst, verstehst und dir ansiehst!
Brustkrebs
Der Brustkrebs-Datensatz ist ein reellwertiger multivariater Datensatz, der aus zwei Klassen besteht, wobei jede Klasse angibt, ob eine Patientin Brustkrebs hat oder nicht. Die beiden Kategorien sind: bösartig und gutartig.
Die bösartige Klasse umfasst 212 Proben, während die gutartige Klasse 357 Proben umfasst.
Es hat 30 Merkmale, die für alle Klassen gleich sind: Radius, Textur, Umfang, Fläche, Glätte, fraktale Dimension usw.
Du kannst den Brustkrebs-Datensatz hier herunterladen, oder du lädst ihn einfach mit Hilfe der sklearn Bibliothek.
CIFAR - 10
Der CIFAR-10-Datensatz (Canadian Institute For Advanced Research) besteht aus 60000 Bildern mit jeweils 32x32x3 Farbbildern, die in zehn Klassen eingeteilt sind, mit 6000 Bildern pro Kategorie.
Der Datensatz besteht aus 50000 Trainingsbildern und 10000 Testbildern.
Die Klassen im Datensatz sind Flugzeug, Auto, Vogel, Katze, Hirsch, Hund, Frosch, Pferd, Schiff und LKW.
Du kannst den CIFAR-Datensatz von hier herunterladen oder ihn mit Hilfe einer Deep-Learning-Bibliothek wie Keras auch direkt laden.
Datenexploration mit PCA
Jetzt wirst du die Datensätze Breast Cancer und CIFAR-10 laden und analysieren. Inzwischen hast du eine Vorstellung von der Dimensionalität der beiden Datensätze.
Lass uns also schnell beide Datensätze untersuchen.
Erforschung von Brustkrebsdaten
Schauen wir uns zunächst den Datensatz Breast Cancer an.
Du verwendest das Modul sklearn's datasets und importierst daraus den Datensatz Breast Cancer.
from sklearn.datasets import load_breast_cancer
load_breast_cancer erhältst du sowohl die Beschriftungen als auch die Daten. Um die Daten zu holen, rufst du .data auf und zum Holen der Labels .target.
Die Daten enthalten 569 Stichproben mit dreißig Merkmalen, und jede Stichprobe ist mit einem Label versehen. In diesem Datensatz gibt es zwei Labels.
breast = load_breast_cancer()
breast_data = breast.data
Lass uns die Form der Daten überprüfen.
breast_data.shape
(569, 30)
Auch wenn du die Beschriftungen für diesen Lehrgang nicht brauchst, lass uns zum besseren Verständnis die Beschriftungen laden und die Form überprüfen.
breast_labels = breast.target
breast_labels.shape
(569,)
Jetzt importierst du numpy, da du die breast_labels umgestalten wirst, um sie mit der breast_data zu verketten, damit du schließlich eine DataFrame erstellen kannst, die sowohl die Daten als auch die Beschriftungen enthält.
import numpy as np
labels = np.reshape(breast_labels,(569,1))
Nach reshaping werden die Beschriftungen concatenate die Daten und Beschriftungen entlang der zweiten Achse, was bedeutet, dass die endgültige Form des Arrays 569 x 31 sein wird.
final_breast_data = np.concatenate([breast_data,labels],axis=1)
final_breast_data.shape
(569, 31)
Jetzt importierst du pandas, um die DataFrame der endgültigen Daten zu erstellen und die Daten in Tabellenform darzustellen.
import pandas as pd
breast_dataset = pd.DataFrame(final_breast_data)
Lass uns schnell die Merkmale ausdrucken, die es im Brustkrebs-Datensatz gibt!
features = breast.feature_names
features
array(['mean radius', 'mean texture', 'mean perimeter', 'mean area',
'mean smoothness', 'mean compactness', 'mean concavity',
'mean concave points', 'mean symmetry', 'mean fractal dimension',
'radius error', 'texture error', 'perimeter error', 'area error',
'smoothness error', 'compactness error', 'concavity error',
'concave points error', 'symmetry error',
'fractal dimension error', 'worst radius', 'worst texture',
'worst perimeter', 'worst area', 'worst smoothness',
'worst compactness', 'worst concavity', 'worst concave points',
'worst symmetry', 'worst fractal dimension'], dtype='<U23')
Wie du im features Array siehst, fehlt das Feld label. Daher musst du sie manuell zum Array features hinzufügen, da du dieses Array mit den Spaltennamen deines DataFrames breast_dataset gleichsetzen wirst.
features_labels = np.append(features,'label')
Toll! Jetzt fügst du die Spaltennamen in den DataFrame breast_dataset ein.
breast_dataset.columns = features_labels
Lass uns die ersten Zeilen des DataFrames ausdrucken.
breast_dataset.head()
| mittlerer Radius | mittlere Textur | mittlerer Umfang | mittlere Fläche | mittlere Glattheit | mittlere Kompaktheit | mittlere Konkavität | mittlere konkave Punkte | mittlere Symmetrie | mittlere fraktale Dimension | ... | schlechteste Beschaffenheit | schlimmster Umfang | schlimmste Gegend | schlechteste Glätte | schlechteste Kompaktheit | schlechteste Konkavität | schlechteste konkave Punkte | schlechteste Symmetrie | schlechteste fraktale Dimension | Etikett | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 17.99 | 10.38 | 122.80 | 1001.0 | 0.11840 | 0.27760 | 0.3001 | 0.14710 | 0.2419 | 0.07871 | ... | 17.33 | 184.60 | 2019.0 | 0.1622 | 0.6656 | 0.7119 | 0.2654 | 0.4601 | 0.11890 | 0.0 |
| 1 | 20.57 | 17.77 | 132.90 | 1326.0 | 0.08474 | 0.07864 | 0.0869 | 0.07017 | 0.1812 | 0.05667 | ... | 23.41 | 158.80 | 1956.0 | 0.1238 | 0.1866 | 0.2416 | 0.1860 | 0.2750 | 0.08902 | 0.0 |
| 2 | 19.69 | 21.25 | 130.00 | 1203.0 | 0.10960 | 0.15990 | 0.1974 | 0.12790 | 0.2069 | 0.05999 | ... | 25.53 | 152.50 | 1709.0 | 0.1444 | 0.4245 | 0.4504 | 0.2430 | 0.3613 | 0.08758 | 0.0 |
| 3 | 11.42 | 20.38 | 77.58 | 386.1 | 0.14250 | 0.28390 | 0.2414 | 0.10520 | 0.2597 | 0.09744 | ... | 26.50 | 98.87 | 567.7 | 0.2098 | 0.8663 | 0.6869 | 0.2575 | 0.6638 | 0.17300 | 0.0 |
| 4 | 20.29 | 14.34 | 135.10 | 1297.0 | 0.10030 | 0.13280 | 0.1980 | 0.10430 | 0.1809 | 0.05883 | ... | 16.67 | 152.20 | 1575.0 | 0.1374 | 0.2050 | 0.4000 | 0.1625 | 0.2364 | 0.07678 | 0.0 |
5 Zeilen × 31 Spalten
Da die ursprünglichen Beschriftungen im Format 0,1 vorliegen, änderst du die Beschriftungen mit der Funktion .replace in benign und malignant. Du verwendest inplace=True, um den DataFrame breast_dataset zu ändern.
breast_dataset['label'].replace(0, 'Benign',inplace=True)
breast_dataset['label'].replace(1, 'Malignant',inplace=True)
Lass uns die letzten Zeilen der breast_dataset ausdrucken.
breast_dataset.tail()
| mittlerer Radius | mittlere Textur | mittlerer Umfang | mittlere Fläche | mittlere Glattheit | mittlere Kompaktheit | mittlere Konkavität | mittlere konkave Punkte | mittlere Symmetrie | mittlere fraktale Dimension | ... | schlechteste Beschaffenheit | schlimmster Umfang | schlimmste Gegend | schlechteste Glätte | schlechteste Kompaktheit | schlechteste Konkavität | schlechteste konkave Punkte | schlechteste Symmetrie | schlechteste fraktale Dimension | Etikett | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 564 | 21.56 | 22.39 | 142.00 | 1479.0 | 0.11100 | 0.11590 | 0.24390 | 0.13890 | 0.1726 | 0.05623 | ... | 26.40 | 166.10 | 2027.0 | 0.14100 | 0.21130 | 0.4107 | 0.2216 | 0.2060 | 0.07115 | Gutartig |
| 565 | 20.13 | 28.25 | 131.20 | 1261.0 | 0.09780 | 0.10340 | 0.14400 | 0.09791 | 0.1752 | 0.05533 | ... | 38.25 | 155.00 | 1731.0 | 0.11660 | 0.19220 | 0.3215 | 0.1628 | 0.2572 | 0.06637 | Gutartig |
| 566 | 16.60 | 28.08 | 108.30 | 858.1 | 0.08455 | 0.10230 | 0.09251 | 0.05302 | 0.1590 | 0.05648 | ... | 34.12 | 126.70 | 1124.0 | 0.11390 | 0.30940 | 0.3403 | 0.1418 | 0.2218 | 0.07820 | Gutartig |
| 567 | 20.60 | 29.33 | 140.10 | 1265.0 | 0.11780 | 0.27700 | 0.35140 | 0.15200 | 0.2397 | 0.07016 | ... | 39.42 | 184.60 | 1821.0 | 0.16500 | 0.86810 | 0.9387 | 0.2650 | 0.4087 | 0.12400 | Gutartig |
| 568 | 7.76 | 24.54 | 47.92 | 181.0 | 0.05263 | 0.04362 | 0.00000 | 0.00000 | 0.1587 | 0.05884 | ... | 30.37 | 59.16 | 268.6 | 0.08996 | 0.06444 | 0.0000 | 0.0000 | 0.2871 | 0.07039 | Bösartig |
5 Zeilen × 31 Spalten
CIFAR - 10 Datenexploration
Als Nächstes wirst du den CIFAR - 10 Bilddatensatz erkunden.
Du kannst den CIFAR - 10 Datensatz mit der Deep Learning Bibliothek Keras laden.
from keras.datasets import cifar10
Nach dem Import verwendest du die Methode .load_data(), um die Daten herunterzuladen. Sie lädt die Daten herunter und speichert sie in deinem Verzeichnis Keras. Je nach deiner Internetgeschwindigkeit kann dies einige Zeit dauern.
(x_train, y_train), (x_test, y_test) = cifar10.load_data()
Die obige Codezeile gibt Trainings- und Testbilder zusammen mit den Beschriftungen zurück.
Lass uns schnell die Form der Trainings- und Testbilder ausdrucken.
print('Traning data shape:', x_train.shape)
print('Testing data shape:', x_test.shape)
Traning data shape: (50000, 32, 32, 3)
Testing data shape: (10000, 32, 32, 3)
Lass uns auch die Form der Etiketten drucken.
y_train.shape,y_test.shape
((50000, 1), (10000, 1))
Wir wollen auch die Gesamtzahl der Etiketten und die verschiedenen Arten von Klassen herausfinden, die die Daten haben.
# Find the unique numbers from the train labels
classes = np.unique(y_train)
nClasses = len(classes)
print('Total number of outputs : ', nClasses)
print('Output classes : ', classes)
Total number of outputs : 10
Output classes : [0 1 2 3 4 5 6 7 8 9]
Um die Bilder von CIFAR-10 zu plotten, importierst du matplotlib und verwendest außerdem den Befehl magic (%) %matplotlib inline , um dem Jupyter-Notebook mitzuteilen, dass es die Ausgabe im Notebook selbst anzeigen soll!
import matplotlib.pyplot as plt
%matplotlib inline
Zum besseren Verständnis erstellen wir ein Wörterbuch mit Klassennamen und den dazugehörigen kategorischen Klassenbezeichnungen.
label_dict = {
0: 'airplane',
1: 'automobile',
2: 'bird',
3: 'cat',
4: 'deer',
5: 'dog',
6: 'frog',
7: 'horse',
8: 'ship',
9: 'truck',
}
plt.figure(figsize=[5,5])
# Display the first image in training data
plt.subplot(121)
curr_img = np.reshape(x_train[0], (32,32,3))
plt.imshow(curr_img)
print(plt.title("(Label: " + str(label_dict[y_train[0][0]]) + ")"))
# Display the first image in testing data
plt.subplot(122)
curr_img = np.reshape(x_test[0],(32,32,3))
plt.imshow(curr_img)
print(plt.title("(Label: " + str(label_dict[y_test[0][0]]) + ")"))
Text(0.5, 1.0, '(Label: frog)')
Text(0.5, 1.0, '(Label: cat)')

Auch wenn die beiden obigen Bilder unscharf sind, kannst du dennoch irgendwie erkennen, dass das erste Bild ein Frosch mit der Bezeichnung frog ist, während das zweite Bild eine Katze mit der Bezeichnung cat zeigt.
Datenvisualisierung mit PCA
Jetzt kommt der spannendste Teil des Tutorials. Da du bereits gelernt hast, dass PCA-Projekte hochdimensionale Daten in eine niedrigdimensionale Hauptkomponente umwandeln, ist es nun an der Zeit, dies mit Hilfe von Python zu visualisieren!
Visualisierung der Brustkrebsdaten
-
Du beginnst mit
Standardizingden Daten, da das Ergebnis der PCA von der Skala der Merkmale der Daten abhängt.Es ist eine gängige Praxis, deine Daten zu normalisieren, bevor du sie in einen Algorithmus für maschinelles Lernen einspeist.
Um die Normalisierung anzuwenden, importierst du das Modul
StandardScaleraus der Sklearn-Bibliothek und wählst nur die Merkmale aus derbreast_datasetaus, die du im Schritt Datenexploration erstellt hast. Sobald du die Merkmale hast, wendest du die Skalierung an, indem dufit_transformauf die Merkmalsdaten anwendest.Wenn du StandardScaler anwendest, sollte jedes Merkmal deiner Daten normalverteilt sein, so dass es die Verteilung auf einen Mittelwert von Null und eine Standardabweichung von Eins skaliert.
from sklearn.preprocessing import StandardScaler
x = breast_dataset.loc[:, features].values
x = StandardScaler().fit_transform(x) # normalizing the features
x.shape
(569, 30)
Prüfen wir, ob die normalisierten Daten einen Mittelwert von Null und eine Standardabweichung von Eins haben.
np.mean(x),np.std(x)
(-6.826538293184326e-17, 1.0)
Lass uns die normalisierten Merkmale mit Hilfe von DataFrame in ein Tabellenformat umwandeln.
feat_cols = ['feature'+str(i) for i in range(x.shape[1])]
normalised_breast = pd.DataFrame(x,columns=feat_cols)
normalised_breast.tail()
| feature0 | feature1 | feature2 | feature3 | feature4 | feature5 | feature6 | feature7 | feature8 | feature9 | ... | feature20 | feature21 | feature22 | feature23 | feature24 | feature25 | feature26 | feature27 | feature28 | feature29 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 564 | 2.110995 | 0.721473 | 2.060786 | 2.343856 | 1.041842 | 0.219060 | 1.947285 | 2.320965 | -0.312589 | -0.931027 | ... | 1.901185 | 0.117700 | 1.752563 | 2.015301 | 0.378365 | -0.273318 | 0.664512 | 1.629151 | -1.360158 | -0.709091 |
| 565 | 1.704854 | 2.085134 | 1.615931 | 1.723842 | 0.102458 | -0.017833 | 0.693043 | 1.263669 | -0.217664 | -1.058611 | ... | 1.536720 | 2.047399 | 1.421940 | 1.494959 | -0.691230 | -0.394820 | 0.236573 | 0.733827 | -0.531855 | -0.973978 |
| 566 | 0.702284 | 2.045574 | 0.672676 | 0.577953 | -0.840484 | -0.038680 | 0.046588 | 0.105777 | -0.809117 | -0.895587 | ... | 0.561361 | 1.374854 | 0.579001 | 0.427906 | -0.809587 | 0.350735 | 0.326767 | 0.414069 | -1.104549 | -0.318409 |
| 567 | 1.838341 | 2.336457 | 1.982524 | 1.735218 | 1.525767 | 3.272144 | 3.296944 | 2.658866 | 2.137194 | 1.043695 | ... | 1.961239 | 2.237926 | 2.303601 | 1.653171 | 1.430427 | 3.904848 | 3.197605 | 2.289985 | 1.919083 | 2.219635 |
| 568 | -1.808401 | 1.221792 | -1.814389 | -1.347789 | -3.112085 | -1.150752 | -1.114873 | -1.261820 | -0.820070 | -0.561032 | ... | -1.410893 | 0.764190 | -1.432735 | -1.075813 | -1.859019 | -1.207552 | -1.305831 | -1.745063 | -0.048138 | -0.751207 |
5 Zeilen × 30 Spalten
-
Jetzt kommt der kritische Teil: In den nächsten Zeilen des Codes werden die dreißigdimensionalen Brustkrebsdaten auf zweidimensionale Daten projiziert
principal components.Du verwendest die Sklearn-Bibliothek, um das Modul
PCAzu importieren. In der PCA-Methode übergibst du die Anzahl der Komponenten (n_components=2) und rufst schließlich fit_transform für die aggregierten Daten auf. Hier stellen mehrere Komponenten die untere Dimension dar, in die du deine höherdimensionalen Daten projizieren wirst.
from sklearn.decomposition import PCA
pca_breast = PCA(n_components=2)
principalComponents_breast = pca_breast.fit_transform(x)
Als Nächstes erstellen wir einen DataFrame, der die Hauptkomponentenwerte für alle 569 Stichproben enthält.
principal_breast_Df = pd.DataFrame(data = principalComponents_breast
, columns = ['principal component 1', 'principal component 2'])
principal_breast_Df.tail()
| Hauptkomponente 1 | Hauptkomponente 2 | |
|---|---|---|
| 564 | 6.439315 | -3.576817 |
| 565 | 3.793382 | -3.584048 |
| 566 | 1.256179 | -1.902297 |
| 567 | 10.374794 | 1.672010 |
| 568 | -5.475243 | -0.670637 |
- Sobald du die Hauptkomponenten hast, kannst du die
explained_variance_ratio. Sie zeigt dir, wie viel Information bzw. Varianz jede Hauptkomponente enthält, nachdem du die Daten auf einen niedrigdimensionalen Unterraum projiziert hast.
print('Explained variability per principal component: {}'.format(pca_breast.explained_variance_ratio_))
Explained variability per principal component: [0.44272026 0.18971182]
Aus der obigen Ausgabe kannst du ersehen, dass die principal component 1 44,2% der Informationen enthält, während die principal component 2 nur 19% der Informationen enthält. Ein weiterer wichtiger Punkt ist, dass bei der Projektion von dreißigdimensionalen Daten auf zweidimensionale Daten 36,8 % der Informationen verloren gingen.
Lass uns die Visualisierung der 569 Proben entlang der Achsen principal component - 1 und principal component - 2 aufzeichnen. Das sollte dir einen guten Einblick geben, wie deine Proben auf die beiden Klassen verteilt sind.
plt.figure()
plt.figure(figsize=(10,10))
plt.xticks(fontsize=12)
plt.yticks(fontsize=14)
plt.xlabel('Principal Component - 1',fontsize=20)
plt.ylabel('Principal Component - 2',fontsize=20)
plt.title("Principal Component Analysis of Breast Cancer Dataset",fontsize=20)
targets = ['Benign', 'Malignant']
colors = ['r', 'g']
for target, color in zip(targets,colors):
indicesToKeep = breast_dataset['label'] == target
plt.scatter(principal_breast_Df.loc[indicesToKeep, 'principal component 1']
, principal_breast_Df.loc[indicesToKeep, 'principal component 2'], c = color, s = 50)
plt.legend(targets,prop={'size': 15})
<matplotlib.legend.Legend at 0x14552a630>
<Figure size 432x288 with 0 Axes>
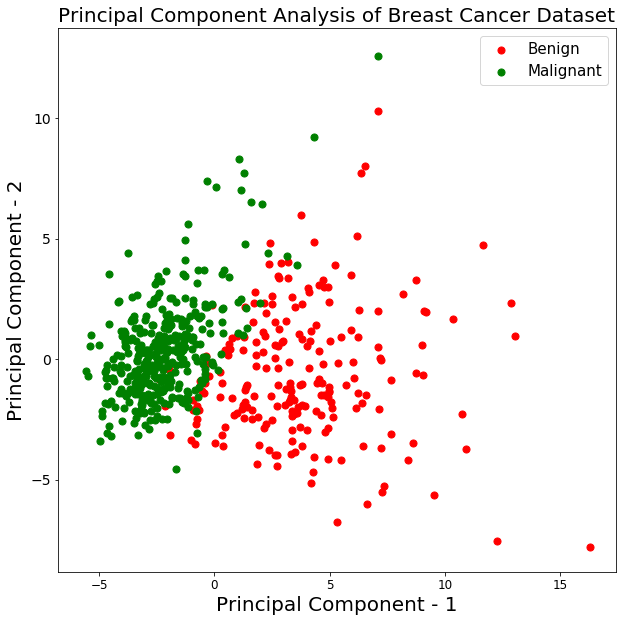
Aus der obigen Grafik kannst du ersehen, dass die beiden Klassen benign und malignant, wenn sie auf einen zweidimensionalen Raum projiziert werden, bis zu einem gewissen Grad linear trennbar sind. Andere Beobachtungen können sein, dass die Klasse benign im Vergleich zur Klasse malignant verteilt ist.
Visualisierung der CIFAR - 10 Daten
Die folgenden Codezeilen zur Visualisierung der CIFAR-10-Daten sind der PCA-Visualisierung der Brustkrebsdaten sehr ähnlich.
- Überprüfen wir schnell die Maximal- und Minimalwerte der CIFAR-10 Trainingsbilder und
normalizedie Pixel zwischen 0 und 1 einschließlich.
np.min(x_train),np.max(x_train)
(0.0, 1.0)
x_train = x_train/255.0
np.min(x_train),np.max(x_train)
(0.0, 0.00392156862745098)
x_train.shape
(50000, 32, 32, 3)
Als Nächstes erstellst du einen DataFrame, der die Pixelwerte der Bilder zusammen mit ihren jeweiligen Beschriftungen in einem Zeilen-Spalten-Format enthält.
Aber vorher müssen wir die Bildmaße von drei auf eins reduzieren (die Bilder abflachen).
x_train_flat = x_train.reshape(-1,3072)
feat_cols = ['pixel'+str(i) for i in range(x_train_flat.shape[1])]
df_cifar = pd.DataFrame(x_train_flat,columns=feat_cols)
df_cifar['label'] = y_train
print('Size of the dataframe: {}'.format(df_cifar.shape))
Size of the dataframe: (50000, 3073)
Perfekt! Die Größe des DataFrames ist korrekt, denn es gibt 50.000 Trainingsbilder mit jeweils 3072 Pixeln und einer zusätzlichen Spalte für Beschriftungen, also insgesamt 3073.
Die PCA wird auf alle Spalten angewandt, außer auf die letzte Spalte, die die Beschriftung für jedes Bild ist.
df_cifar.head()
| pixel0 | pixel1 | pixel2 | pixel3 | pixel4 | pixel5 | pixel6 | pixel7 | pixel8 | pixel9 | ... | pixel3063 | pixel3064 | pixel3065 | pixel3066 | pixel3067 | pixel3068 | pixel3069 | pixel3070 | pixel3071 | Etikett | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 0.231373 | 0.243137 | 0.247059 | 0.168627 | 0.180392 | 0.176471 | 0.196078 | 0.188235 | 0.168627 | 0.266667 | ... | 0.847059 | 0.721569 | 0.549020 | 0.592157 | 0.462745 | 0.329412 | 0.482353 | 0.360784 | 0.282353 | 6 |
| 1 | 0.603922 | 0.694118 | 0.733333 | 0.494118 | 0.537255 | 0.533333 | 0.411765 | 0.407843 | 0.372549 | 0.400000 | ... | 0.560784 | 0.521569 | 0.545098 | 0.560784 | 0.525490 | 0.556863 | 0.560784 | 0.521569 | 0.564706 | 9 |
| 2 | 1.000000 | 1.000000 | 1.000000 | 0.992157 | 0.992157 | 0.992157 | 0.992157 | 0.992157 | 0.992157 | 0.992157 | ... | 0.305882 | 0.333333 | 0.325490 | 0.309804 | 0.333333 | 0.325490 | 0.313725 | 0.337255 | 0.329412 | 9 |
| 3 | 0.109804 | 0.098039 | 0.039216 | 0.145098 | 0.133333 | 0.074510 | 0.149020 | 0.137255 | 0.078431 | 0.164706 | ... | 0.211765 | 0.184314 | 0.109804 | 0.247059 | 0.219608 | 0.145098 | 0.282353 | 0.254902 | 0.180392 | 4 |
| 4 | 0.666667 | 0.705882 | 0.776471 | 0.658824 | 0.698039 | 0.768627 | 0.694118 | 0.725490 | 0.796078 | 0.717647 | ... | 0.294118 | 0.309804 | 0.321569 | 0.278431 | 0.294118 | 0.305882 | 0.286275 | 0.301961 | 0.313725 | 1 |
5 Zeilen × 3073 Spalten
- Als Nächstes erstellst du die PCA-Methode, gibst die Anzahl der Komponenten mit zwei an und wendest
fit_transformauf die Trainingsdaten an. Dies kann einige Sekunden dauern, da es 50.000 Proben gibt.
pca_cifar = PCA(n_components=2)
principalComponents_cifar = pca_cifar.fit_transform(df_cifar.iloc[:,:-1])
Dann konvertierst du die Hauptkomponenten für jedes der 50.000 Bilder von einem Numpy-Array in einen Pandas DataFrame.
principal_cifar_Df = pd.DataFrame(data = principalComponents_cifar
, columns = ['principal component 1', 'principal component 2'])
principal_cifar_Df['y'] = y_train
principal_cifar_Df.head()
| Hauptkomponente 1 | Hauptkomponente 2 | y | |
|---|---|---|---|
| 0 | -6.401018 | 2.729039 | 6 |
| 1 | 0.829783 | -0.949943 | 9 |
| 2 | 7.730200 | -11.522102 | 9 |
| 3 | -10.347817 | 0.010738 | 4 |
| 4 | -2.625651 | -4.969240 | 1 |
- Lass uns schnell herausfinden, wie viele Informationen oder
variancedie Hauptkomponenten enthalten.
print('Explained variability per principal component: {}'.format(pca_cifar.explained_variance_ratio_))
Explained variability per principal component: [0.2907663 0.11253144]
Nun, es sieht so aus, als ob die Hauptkomponenten 1 und 2 eine anständige Menge an Informationen behalten haben, wenn man bedenkt, dass die Daten von 3072 Dimensionen auf nur zwei Hauptkomponenten projiziert wurden.
Es ist an der Zeit, die CIFAR-10-Daten in einem zweidimensionalen Raum zu visualisieren. Denke daran, dass es in diesem Datensatz einige semantische Klassenüberschneidungen gibt, was bedeutet, dass ein Frosch eine leicht ähnliche Form wie eine Katze oder ein Reh wie ein Hund haben kann; vor allem, wenn er in einen zweidimensionalen Raum projiziert wird. Die Unterschiede zwischen ihnen sind vielleicht nicht so gut zu erkennen.
import seaborn as sns
plt.figure(figsize=(16,10))
sns.scatterplot(
x="principal component 1", y="principal component 2",
hue="y",
palette=sns.color_palette("hls", 10),
data=principal_cifar_Df,
legend="full",
alpha=0.3
)
<matplotlib.axes._subplots.AxesSubplot at 0x12a5ba8d0>
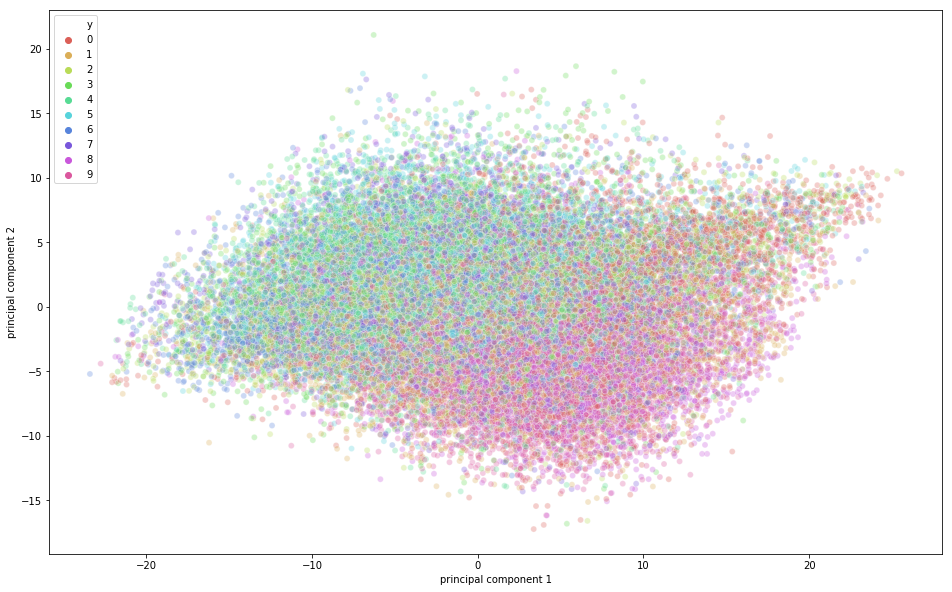
Aus der obigen Abbildung kannst du ersehen, dass eine gewisse Variation durch die Hauptkomponenten erfasst wurde, da es eine gewisse Struktur in den Punkten gibt, wenn sie entlang der zwei Hauptkomponentenachsen projiziert werden. Die Punkte, die zur gleichen Klasse gehören, liegen nahe beieinander, und die Punkte oder Bilder, die sich semantisch stark unterscheiden, sind weiter voneinander entfernt.
Beschleunige das Deep Learning Training mit PCA
In diesem letzten Abschnitt des Tutorials lernst du, wie du den Trainingsprozess deines Deep Learning-Modells mit PCA beschleunigen kannst.
Hinweis: Um die grundlegenden Begriffe zu lernen, die in diesem Abschnitt verwendet werden, schau dir bitte dieses Tutorial an.
Zuerst normalisieren wir die Trainings- und Testbilder. Wenn du dich daran erinnerst, dass die Trainingsbilder im PCA-Visualisierungsteil normalisiert wurden, musst du nur noch die Testbilder normalisieren. Also, lass uns das schnell machen!
x_test = x_test/255.0
x_test = x_test.reshape(-1,32,32,3)
Die Testdaten findest du unter reshape.
x_test_flat = x_test.reshape(-1,3072)
Als Nächstes erstellst du die Instanz des PCA-Modells.
Hier kannst du auch angeben, wie viel Varianz du mit der PCA erfassen willst. Übergeben wir 0,9 als Parameter an das PCA-Modell. Das bedeutet, dass die PCA 90% der Varianz erfasst und die number of components verwendet wird, die erforderlich ist, um 90% der Varianz zu erfassen.
Beachte, dass du zuvor n_components als Parameter übergeben hast und dann herausfinden konntest, wie viel Varianz von diesen beiden Komponenten erfasst wurde. Aber hier geben wir explizit an, wie viel Varianz wir mit der PCA erfassen möchten, und daher wird die n_components je nach Varianzparameter variieren.
Wenn du keine Varianz angibst, ist die Anzahl der Komponenten gleich der ursprünglichen Dimension der Daten.
pca = PCA(0.9)
Dann passt du die Instanz PCA an die Trainingsbilder an.
pca.fit(x_train_flat)
PCA(copy=True, iterated_power='auto', n_components=0.9, random_state=None,
svd_solver='auto', tol=0.0, whiten=False)
Jetzt wollen wir herausfinden, wie viele n_components PCA benötigt, um 0,9 Varianz zu erfassen.
pca.n_components_
99
Aus der obigen Ausgabe kannst du ersehen, dass die Dimensionen von 3072 auf 99 Hauptkomponenten reduziert wurden, um eine Varianz von 90% zu erreichen.
Zum Schluss wendest du transform auf den Trainings- und Testdatensatz an, um aus den Parametern der fit Methode einen transformierten Datensatz zu erzeugen.
train_img_pca = pca.transform(x_train_flat)
test_img_pca = pca.transform(x_test_flat)
Als Nächstes importieren wir schnell die notwendigen Bibliotheken, um das Deep-Learning-Modell auszuführen.
from keras.models import Sequential
from keras.layers import Dense
from keras.utils import np_utils
from keras.optimizers import RMSprop
Jetzt konvertierst du deine Trainings- und Testkennungen in einen One-Hot-Codierungsvektor.
y_train = np_utils.to_categorical(y_train)
y_test = np_utils.to_categorical(y_test)
Legen wir die Anzahl der Epochen, die Anzahl der Klassen und die Stapelgröße für dein Modell fest.
batch_size = 128
num_classes = 10
epochs = 20
Als Nächstes wirst du dein Sequential Modell definieren!
model = Sequential()
model.add(Dense(1024, activation='relu', input_shape=(99,)))
model.add(Dense(1024, activation='relu'))
model.add(Dense(512, activation='relu'))
model.add(Dense(256, activation='relu'))
model.add(Dense(num_classes, activation='softmax'))
Lass uns die Modellzusammenfassung drucken.
model.summary()
_________________________________________________________________
Layer (type) Output Shape Param #
=================================================================
dense_1 (Dense) (None, 1024) 102400
_________________________________________________________________
dense_2 (Dense) (None, 1024) 1049600
_________________________________________________________________
dense_3 (Dense) (None, 512) 524800
_________________________________________________________________
dense_4 (Dense) (None, 256) 131328
_________________________________________________________________
dense_5 (Dense) (None, 10) 2570
=================================================================
Total params: 1,810,698
Trainable params: 1,810,698
Non-trainable params: 0
_________________________________________________________________
Endlich ist es an der Zeit, das Modell zu erstellen und zu trainieren!
model.compile(loss='categorical_crossentropy',
optimizer=RMSprop(),
metrics=['accuracy'])
history = model.fit(train_img_pca, y_train,batch_size=batch_size,epochs=epochs,verbose=1,
validation_data=(test_img_pca, y_test))
WARNING:tensorflow:From /Users/adityasharma/blog/lib/python3.7/site-packages/keras/backend/tensorflow_backend.py:2704: calling reduce_sum (from tensorflow.python.ops.math_ops) with keep_dims is deprecated and will be removed in a future version.
Instructions for updating:
keep_dims is deprecated, use keepdims instead
WARNING:tensorflow:From /Users/adityasharma/blog/lib/python3.7/site-packages/keras/backend/tensorflow_backend.py:1257: calling reduce_mean (from tensorflow.python.ops.math_ops) with keep_dims is deprecated and will be removed in a future version.
Instructions for updating:
keep_dims is deprecated, use keepdims instead
Train on 50000 samples, validate on 10000 samples
Epoch 1/20
50000/50000 [==============================] - 7s - loss: 1.9032 - acc: 0.2962 - val_loss: 1.6925 - val_acc: 0.3875
Epoch 2/20
50000/50000 [==============================] - 7s - loss: 1.6480 - acc: 0.4055 - val_loss: 1.5313 - val_acc: 0.4412
Epoch 3/20
50000/50000 [==============================] - 7s - loss: 1.5205 - acc: 0.4534 - val_loss: 1.4609 - val_acc: 0.4695
Epoch 4/20
50000/50000 [==============================] - 7s - loss: 1.4322 - acc: 0.4849 - val_loss: 1.6164 - val_acc: 0.4503
Epoch 5/20
50000/50000 [==============================] - 7s - loss: 1.3621 - acc: 0.5120 - val_loss: 1.3626 - val_acc: 0.5081
Epoch 6/20
50000/50000 [==============================] - 7s - loss: 1.2995 - acc: 0.5330 - val_loss: 1.4100 - val_acc: 0.4940
Epoch 7/20
50000/50000 [==============================] - 7s - loss: 1.2473 - acc: 0.5529 - val_loss: 1.3589 - val_acc: 0.5251
Epoch 8/20
50000/50000 [==============================] - 7s - loss: 1.2010 - acc: 0.5669 - val_loss: 1.3315 - val_acc: 0.5232
Epoch 9/20
50000/50000 [==============================] - 7s - loss: 1.1524 - acc: 0.5868 - val_loss: 1.3903 - val_acc: 0.5197
Epoch 10/20
50000/50000 [==============================] - 7s - loss: 1.1134 - acc: 0.6013 - val_loss: 1.2722 - val_acc: 0.5499
Epoch 11/20
50000/50000 [==============================] - 7s - loss: 1.0691 - acc: 0.6160 - val_loss: 1.5911 - val_acc: 0.4768
Epoch 12/20
50000/50000 [==============================] - 7s - loss: 1.0325 - acc: 0.6289 - val_loss: 1.2515 - val_acc: 0.5602
Epoch 13/20
50000/50000 [==============================] - 7s - loss: 0.9977 - acc: 0.6420 - val_loss: 1.5678 - val_acc: 0.4914
Epoch 14/20
50000/50000 [==============================] - 8s - loss: 0.9567 - acc: 0.6567 - val_loss: 1.3525 - val_acc: 0.5418
Epoch 15/20
50000/50000 [==============================] - 9s - loss: 0.9158 - acc: 0.6713 - val_loss: 1.3525 - val_acc: 0.5540
Epoch 16/20
50000/50000 [==============================] - 10s - loss: 0.8948 - acc: 0.6816 - val_loss: 1.5633 - val_acc: 0.5156
Epoch 17/20
50000/50000 [==============================] - 9s - loss: 0.8690 - acc: 0.6903 - val_loss: 1.6980 - val_acc: 0.5084
Epoch 18/20
50000/50000 [==============================] - 9s - loss: 0.8586 - acc: 0.7002 - val_loss: 1.6325 - val_acc: 0.5247
Epoch 19/20
50000/50000 [==============================] - 8s - loss: 0.9367 - acc: 0.6853 - val_loss: 1.8253 - val_acc: 0.5165
Epoch 20/20
50000/50000 [==============================] - 8s - loss: 2.3761 - acc: 0.5971 - val_loss: 6.0192 - val_acc: 0.4409
Aus der obigen Ausgabe kannst du ersehen, dass die Zeit, die für das Training jeder Epoche benötigt wurde, nur 7 seconds auf einer CPU betrug. Das Modell leistete gute Arbeit bei den Trainingsdaten und erreichte eine Genauigkeit von 70%, während es bei den Testdaten nur eine Genauigkeit von 56% erreichte. Das bedeutet, dass er die Trainingsdaten übererfüllt hat. Bedenke jedoch, dass die Daten von 3072 Dimensionen auf 99 Dimensionen projiziert wurden, und trotzdem hat sie gute Arbeit geleistet!
Abschließend wollen wir sehen, wie viel Zeit das Modell zum Trainieren auf dem Originaldatensatz benötigt und wie viel Genauigkeit es mit demselben Deep Learning-Modell erreichen kann.
model = Sequential()
model.add(Dense(1024, activation='relu', input_shape=(3072,)))
model.add(Dense(1024, activation='relu'))
model.add(Dense(512, activation='relu'))
model.add(Dense(256, activation='relu'))
model.add(Dense(num_classes, activation='softmax'))
model.compile(loss='categorical_crossentropy',
optimizer=RMSprop(),
metrics=['accuracy'])
history = model.fit(x_train_flat, y_train,batch_size=batch_size,epochs=epochs,verbose=1,
validation_data=(x_test_flat, y_test))
Train on 50000 samples, validate on 10000 samples
Epoch 1/20
50000/50000 [==============================] - 23s - loss: 2.0657 - acc: 0.2200 - val_loss: 2.0277 - val_acc: 0.2485
Epoch 2/20
50000/50000 [==============================] - 22s - loss: 1.8727 - acc: 0.3166 - val_loss: 1.8428 - val_acc: 0.3215
Epoch 3/20
50000/50000 [==============================] - 22s - loss: 1.7801 - acc: 0.3526 - val_loss: 1.7657 - val_acc: 0.3605
Epoch 4/20
50000/50000 [==============================] - 22s - loss: 1.7141 - acc: 0.3796 - val_loss: 1.6345 - val_acc: 0.4132
Epoch 5/20
50000/50000 [==============================] - 22s - loss: 1.6566 - acc: 0.4001 - val_loss: 1.6384 - val_acc: 0.4076
Epoch 6/20
50000/50000 [==============================] - 22s - loss: 1.6083 - acc: 0.4209 - val_loss: 1.7507 - val_acc: 0.3574
Epoch 7/20
50000/50000 [==============================] - 22s - loss: 1.5626 - acc: 0.4374 - val_loss: 1.7125 - val_acc: 0.4010
Epoch 8/20
50000/50000 [==============================] - 22s - loss: 1.5252 - acc: 0.4486 - val_loss: 1.5914 - val_acc: 0.4321
Epoch 9/20
50000/50000 [==============================] - 24s - loss: 1.4924 - acc: 0.4620 - val_loss: 1.5352 - val_acc: 0.4616
Epoch 10/20
50000/50000 [==============================] - 25s - loss: 1.4627 - acc: 0.4728 - val_loss: 1.4561 - val_acc: 0.4798
Epoch 11/20
50000/50000 [==============================] - 24s - loss: 1.4349 - acc: 0.4820 - val_loss: 1.5044 - val_acc: 0.4723
Epoch 12/20
50000/50000 [==============================] - 24s - loss: 1.4120 - acc: 0.4919 - val_loss: 1.4740 - val_acc: 0.4790
Epoch 13/20
50000/50000 [==============================] - 23s - loss: 1.3913 - acc: 0.4981 - val_loss: 1.4430 - val_acc: 0.4891
Epoch 14/20
50000/50000 [==============================] - 27s - loss: 1.3678 - acc: 0.5098 - val_loss: 1.4323 - val_acc: 0.4888
Epoch 15/20
50000/50000 [==============================] - 27s - loss: 1.3508 - acc: 0.5148 - val_loss: 1.6179 - val_acc: 0.4372
Epoch 16/20
50000/50000 [==============================] - 25s - loss: 1.3443 - acc: 0.5167 - val_loss: 1.5868 - val_acc: 0.4656
Epoch 17/20
50000/50000 [==============================] - 25s - loss: 1.3734 - acc: 0.5101 - val_loss: 1.4756 - val_acc: 0.4913
Epoch 18/20
50000/50000 [==============================] - 26s - loss: 5.5126 - acc: 0.3591 - val_loss: 5.7580 - val_acc: 0.3084
Epoch 19/20
50000/50000 [==============================] - 27s - loss: 5.6346 - acc: 0.3395 - val_loss: 3.7362 - val_acc: 0.3402
Epoch 20/20
50000/50000 [==============================] - 26s - loss: 6.4199 - acc: 0.3030 - val_loss: 13.9429 - val_acc: 0.1326
Voila! Aus dem obigen Ergebnis ist ersichtlich, dass der Zeitaufwand für das Training jeder Epoche etwa 23 seconds auf einer CPU betrug und damit fast dreimal so hoch war wie bei dem Modell, das anhand der PCA-Ausgabe trainiert wurde.
Außerdem ist sowohl die Trainings- als auch die Testgenauigkeit geringer als die Genauigkeit, die du mit den 99 Hauptkomponenten als Input für das Modell erreicht hast.
Durch die Anwendung von PCA auf die Trainingsdaten konntest du deinen Deep-Learning-Algorithmus also nicht nur fast trainieren, sondern er erzielte auch bessere accuracy bei den Testdaten im Vergleich zu dem Deep-Learning-Algorithmus, der mit den ursprünglichen Trainingsdaten trainiert wurde.
Geh weiter!
Herzlichen Glückwunsch zum Abschluss des Tutorials.
Dieses Tutorium war eine hervorragende und umfassende Einführung in die PCA in Python, die sowohl die theoretischen als auch die praktischen Konzepte der PCA abdeckte.
Wenn du tiefer in die Techniken zur Dimensionalitätsreduzierung eintauchen möchtest, dann solltest du über t-distributed Stochastic Neighbor Embedding ( tSNE) lesen, eine nichtlineare probabilistische Technik zur Dimensionalitätsreduzierung.
Wenn du mehr über unüberwachte Lerntechniken wie PCA erfahren möchtest, besuche den Kurs Unüberwachtes Lernen in Python von DataCamp.
Referenzen für weiteres Lernen:
Häufig gestellte Fragen
Was ist der Unterschied zwischen Faktorenanalyse und Hauptkomponentenanalyse?
Die Faktorenanalyse (FA) und die Hauptkomponentenanalyse (PCA) sind beides Techniken zur Dimensionalitätsreduzierung, die jedoch unterschiedliche Ziele verfolgen. Die PCA konzentriert sich darauf, die Gesamtvariabilität der Daten zu erhalten, indem sie in einen neuen Satz unkorrelierter Variablen (Hauptkomponenten) umgewandelt wird, die nach dem Anteil der Varianz, den sie erklären, geordnet sind. Im Gegensatz dazu zielt die FA darauf ab, die zugrunde liegenden Beziehungen zwischen den beobachteten Variablen zu identifizieren, indem sie die Daten mit einigen latenten Faktoren modelliert, die die Korrelationen zwischen den Variablen erklären.
Was ist die Hauptkomponentenanalyse?
Bei der Hauptkomponentenanalyse werden die Eigenvektoren der Kovarianzmatrix der Daten ermittelt, um sie auf einen niedrigdimensionalen Raum zu projizieren, der durch die Hauptkomponenten (die mit den größten Eigenwerten) definiert ist.