Curso
Imagina que intentas optimizar las rutas de reparto de los camiones. Cada camión tiene muchas rutas posibles, y tú tienes muchos camiones con muchas paradas. El número de combinaciones posibles puede ser abrumador, y encontrar la mejor solución puede ser como buscar una aguja en un pajar.
Para resolver problemas como éstos, podemos tomar prestado un concepto de la biología: la evolución. En biología, la selección natural es una fuerza que impulsa la evolución. El problema de la naturaleza es encontrar las combinaciones óptimas de rasgos que permitan a un organismo sobrevivir y reproducirse. Utiliza la selección natural para poner diferentes conjuntos de rasgos en competencia entre sí y elegir las mejores combinaciones.
Del mismo modo, podemos utilizar este concepto para crear un algoritmo que enfrente diferentes soluciones entre sí y utilice la selección para "hacerlas evolucionar" hacia la mejor solución. Los algoritmos que hacen esto se llaman algoritmos genéticos (AG).
Desarrollar aplicaciones de IA
Inspirados en la evolución natural, los AG exploran eficazmente el espacio de soluciones para descubrir soluciones óptimas o casi óptimas, incluso para problemas complejos con múltiples partes móviles. Al imitar el proceso de selección natural, los AG pueden hacer evolucionar las soluciones a lo largo del tiempo, mejorando su rendimiento de forma iterativa.
Antecedentes biológicos de los algoritmos genéticos
Los algoritmos genéticos se inspiraron en el proceso de la selección natural y la genética. Imitan el modo en que los organismos vivos evolucionan a lo largo de generaciones para adaptarse y sobrevivir en su entorno. Comprender los principios biológicos básicos de los AG puede ayudarnos a entender cómo funcionan estos algoritmos y a utilizarlos de forma más estratégica.
La selección natural
La selección natural es el proceso que hace que los individuos con mejores combinaciones de rasgos favorables tengan más probabilidades de sobrevivir y reproducirse. Con el tiempo, estos rasgos ventajosos se hacen más comunes en la población.
En el contexto de los AG, este concepto se refleja en la selección de individuos con puntuaciones de aptitud más altas para la reproducción. De este modo, transmiten sus rasgos favorables, o "genes", a la siguiente generación.
Genes, cruce y mutación
Los rasgos de un organismo biológico están codificados por sus genes. Los genes forman parte del ADN, que está organizado en cromosomas.
Los AG toman prestados muchos de los mismos conceptos y terminología. Las posibles soluciones a un problema se representan como "cromosomas", que suelen codificarse como cadenas o matrices. Cada elemento del cromosoma corresponde a un gen que determina un rasgo específico de la solución.
El cruce, o recombinación, es una operación genética que combina partes del material genético de dos cromosomas parentales en un nuevo cromosoma que tiene partes de cada progenitor. Esta nueva mezcla de cromosomas se transmite al niño. Es una de las razones por las que puedes tener el color de ojos de tu madre y el color de pelo de tu padre.
En los AG, el cruce consiste en intercambiar segmentos de los cromosomas padres para crear nuevos individuos. Esta operación introduce variabilidad y permite que la descendencia herede rasgos beneficiosos de ambos progenitores.
La mutación introduce cambios aleatorios en los genes de un organismo, que pueden dar lugar a nuevos rasgos.
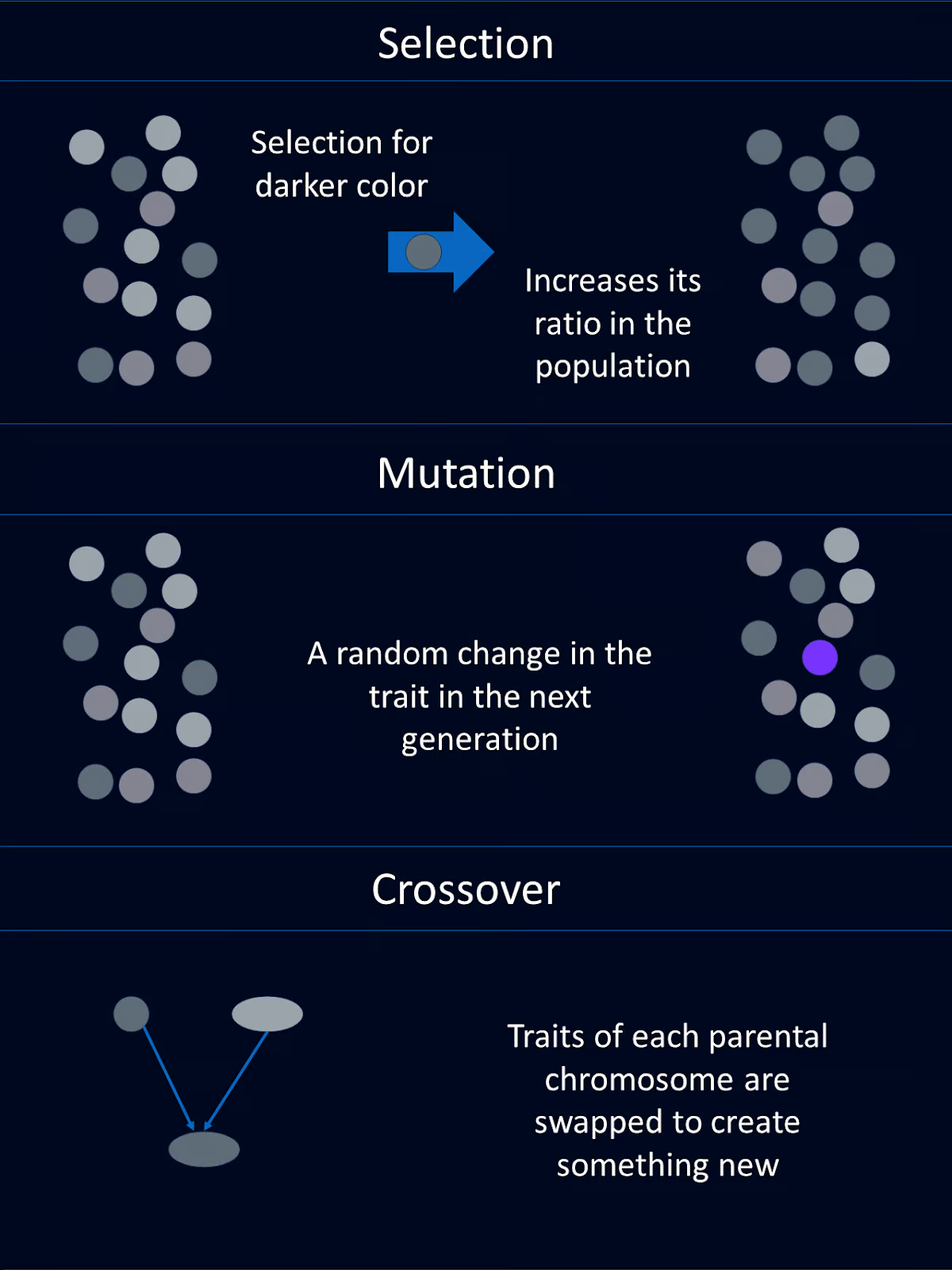
En los algoritmos genéticos, la mutación consiste en alterar aleatoriamente uno o varios genes de un cromosoma. Esto ayuda a mantener la diversidad genética dentro de la población y permite al algoritmo explorar nuevas áreas del espacio de soluciones.
Evaluación de la aptitud física
En la naturaleza, la aptitud de un individuo viene determinada por su capacidad para sobrevivir y reproducirse. Si mueres antes de reproducirte, tienes una aptitud de cero. Si tienes 12 nietos, tu forma física será mayor que la de alguien con 2 nietos.
En los AG, la aptitud es similar, pero un poco diferente. Una función de adecuación evalúa lo bien que una solución resuelve el problema planteado. Las soluciones con puntuaciones de aptitud más altas tienen más probabilidades de ser seleccionadas para la reproducción, lo que garantiza que las mejores soluciones se propaguen a través de las generaciones.
Componentes de un Algoritmo Genético
Los algoritmos genéticos se inspiraron en la evolución. En consecuencia, los componentes de un AG comparten nombres y funciones similares a los de sus homólogos biológicos. Cada uno de estos componentes suele codificarse como una función propia.
Población
La población en un algoritmo genético es un conjunto de soluciones candidatas, a menudo llamadas individuos. Cada individuo se representa como un cromosoma, que puede codificarse como cadenas binarias, matricesu otras estructuras de datos.
Por ejemplo, cada cromosoma podría representar un conjunto de valores de entrada a una función que tenemos que optimizar. O cada cromosoma podría representar una ruta de camión para los conductores de reparto.
Función de aptitud
La función de adecuación evalúa la capacidad de cada individuo para resolver el problema que nos interesa. Asigna un valor de aptitud a cada individuo, con valores más altos para las mejores soluciones. La función de adecuación guía al algoritmo hacia mejores soluciones.
Por ejemplo, en nuestro ejemplo de optimización de una función matemática, la función de adecuación podría devolver el valor de la función para las entradas dadas. Para nuestro ejemplo de rutas de camiones, la función de adecuación podría devolver la velocidad de finalización de la entrega.
Función de selección
La función de selección elige a los individuos de la población que se van a reproducir en función de su aptitud. Los individuos con mayor aptitud tienen más probabilidades de ser elegidos para la reproducción. Esto imita la selección natural, en la que los individuos mejor adaptados tienen más probabilidades de sobrevivir y reproducirse.
Existen varios métodos de selección. Los métodos de selección habituales son la selección de ruleta, la selección de torneo y la selección por rango.
La selección mediante ruleta consiste en seleccionar a los individuos en función de una probabilidad proporcional a su aptitud, de forma similar a como se hace girar una ruleta.
En la selección por torneo se elige al azar un subconjunto de individuos y se selecciona al individuo más apto de ese subconjunto.
La selección por rango clasifica a los individuos en función de su aptitud. A continuación, se asignan probabilidades de selección basadas en estos rangos en lugar de en los valores brutos de aptitud.
Cada método tiene sus propias ventajas y debe elegirse en función de los requisitos específicos del problema en cuestión.
Función cruzada
El cruce combina la información de dos individuos para crear descendencia. El objetivo es heredar rasgos beneficiosos de ambos progenitores. Las técnicas habituales de cruce son el cruce de un punto, el cruce de varios puntos, el cruce uniforme y el cruce mixto.
El cruce de un solo punto consiste en elegir un punto al azar e intercambiar el material genético de dos progenitores en ese punto para crear dos descendientes. El cruce multipunto utiliza varios puntos para intercambiar segmentos entre los progenitores, lo que permite combinaciones genéticas más complejas.
El cruce uniforme decide aleatoriamente qué progenitor aportará cada gen, proporcionando un alto nivel de diversidad genética. El cruce de mezcla genera descendencia mezclando los genes de los padres mediante un factor de mezcla aleatorio. La decisión de qué técnica utilizar debe basarse en la complejidad del problema y en el nivel de diversidad deseado en la descendencia.
Función de mutación
La mutación introduce cambios aleatorios en el material genético de la descendencia. Esto ayuda a mantener la diversidad en la población y explora nuevas áreas del espacio de soluciones. Las mutaciones pueden ser tan sencillas como voltear un bit en una cadena binaria o pueden implicar cambios más complejos según el esquema de codificación.
El proceso del algoritmo genético
Un algoritmo genético sigue una serie de pasos que imitan los procesos evolutivos naturales para encontrar soluciones óptimas. Estos pasos permiten que la población evolucione a lo largo de las generaciones, mejorando la calidad de las soluciones. He aquí una pauta general de cómo procede un algoritmo genético:
Paso 1: Inicialización
En primer lugar, generamos una población inicial de individuos aleatorios. Este paso crea un conjunto diverso de soluciones potenciales para iniciar el algoritmo.
Paso 2: Evaluación
A continuación, tenemos que calcular la aptitud de cada individuo de la población. Aquí utilizamos la función de adecuación para evaluar lo buena que es cada solución.
Paso 3: Selección
Utilizando los criterios de selección, seleccionamos a los individuos para la reproducción en función de su aptitud. Este paso determina qué individuos serán padres.
Paso 4: Cruce
El cruce viene a continuación. Combinando el material genético de los padres seleccionados, aplicamos técnicas de cruce para generar nuevas soluciones o descendientes.
Paso 5: Mutación
Para mantener la diversidad en nuestra población, necesitamos introducir mutaciones aleatorias en la descendencia.
Paso 6: Sustitución
A continuación, sustituimos parte o toda la población antigua por la nueva descendencia, determinando qué individuos pasan a la siguiente generación.
Paso 7: Repite
Los pasos anteriores 2-6 se repiten en bucle durante un número determinado de generaciones o hasta que se cumpla una condición de finalización. Este bucle permite que la población evolucione con el tiempo, con la esperanza de que dé lugar a una buena solución.
Ejemplo de Python
Ahora que ya sabemos qué son los algoritmos genéticos y, en general, cómo funcionan, vamos a construir nuestro propio algoritmo genético para resolver un sencillo problema de optimización.
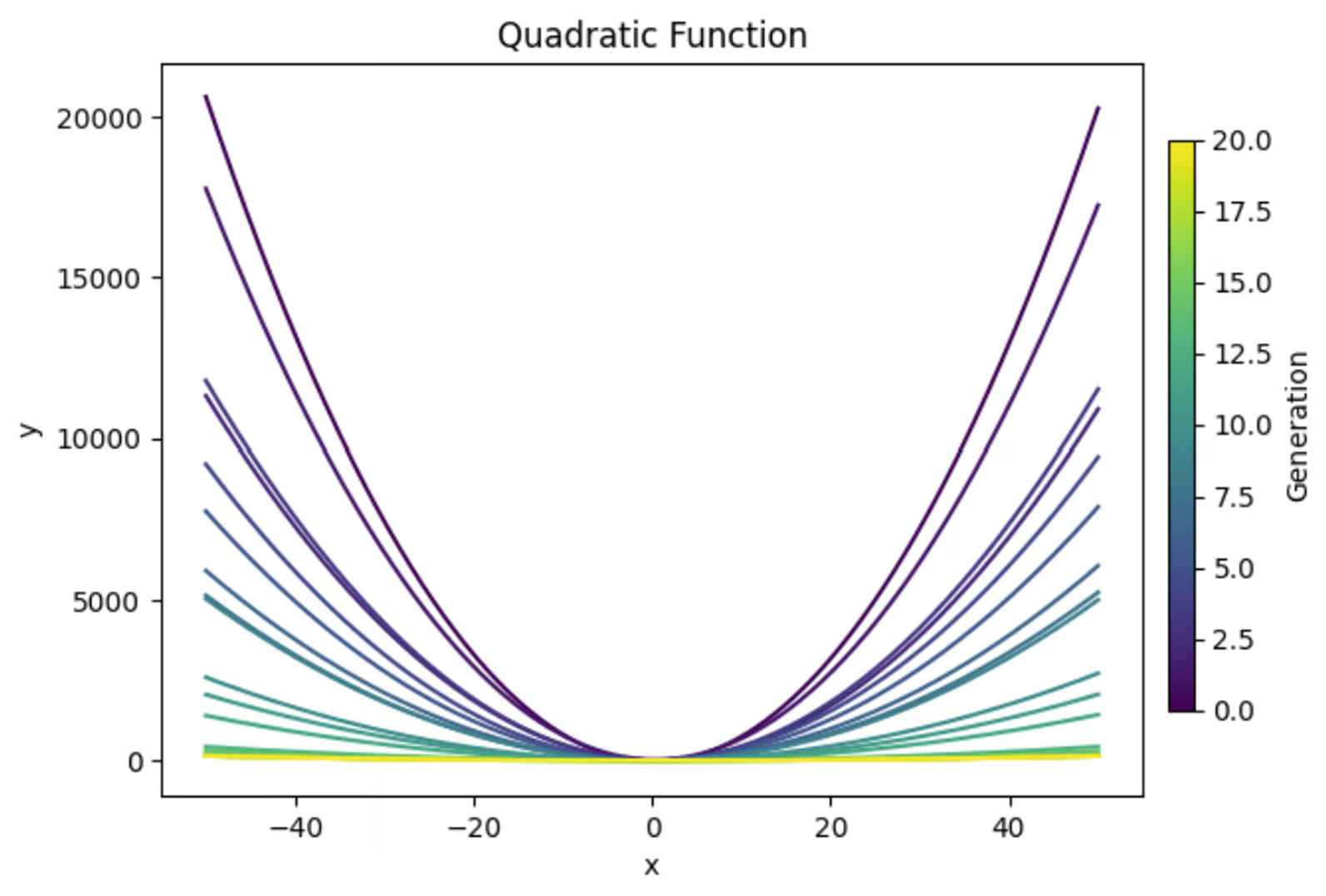
La ecuación y=ax2+bx+c, cuando se representa gráficamente, crea una parábola. Utilizaremos un algoritmo genético para encontrar la combinación de valores de a, b y c que dé como resultado la parábola más plana. Aquí tienes un avance de lo que conseguiremos:
Importar bibliotecas
En la parte superior de nuestra función, importaremos las bibliotecas necesarias. Utilizaremos "aleatorio" para generar valores aleatorios para nuestra población inicial. Numpy en general es útil para hacer cualquier cálculo, en mi opinión.
Y soy un firme creyente de que siempre debes hacer parcelas para asegurarte de que tu código está haciendo lo que crees que está haciendo. Así que importaremos 'matplotlib.pyplot' para hacer algunos gráficos.
También me gusta imprimir los resultados importantes de cada generación (suponiendo un número relativamente pequeño de generaciones) para comprender mejor lo que ocurrió durante la simulación. Para ello importaremos 'PrettyTable'.
import random
import matplotlib.pyplot as plt
import numpy as np
from prettytable import PrettyTableDefinir la función de adecuación
A continuación tenemos que definir la función de aptitud que evaluará la aptitud de cada solución. En este caso, queremos encontrar la forma en U graficada más plana. Así que calculé el valor de y en el vértice y en x=-1 y x=1.
Luego calculé la curvatura de la gráfica como la diferencia del valor dey en estos tres puntos. Como queremos obtener la curva más plana, he negado la curvatura para terminar la función de adecuación.
# Define the fitness function (objective is to create the flattest U-shape)
def fitness_function(params):
a, b, c = params
if a <= 0:
return -float('inf') # Penalize downward facing u-shapes heavily
vertex_x = -b / (2 * a) #x value at vertex
vertex_y = a * (vertex_x ** 2) + b * vertex_x + c #y value at vertex
y_left = a * (-1) ** 2 + b * (-1) + c #y-coordinate at x = -1
y_right = a * (1) ** 2 + b * (1) + c #y-coordinate at x = 1
curviness = abs(y_left - vertex_y) + abs(y_right - vertex_y)
return -curviness # Negate to minimize curvinessCrear población inicial
Utilizaremos la biblioteca "random" para generar una población de soluciones aleatorias. Cada individuo de esta población es un conjunto de valores para a, b y c.
# Create the initial population
def create_initial_population(size, lower_bound, upper_bound):
population = []
for _ in range(size):
individual = (random.uniform(lower_bound, upper_bound),
random.uniform(lower_bound, upper_bound),
random.uniform(lower_bound, upper_bound))
population.append(individual)
return populationCrear una función de selección
La función de selección determinará qué individuos se reproducen para crear la siguiente población. En este ejemplo, utilizaremos un proceso de selección de torneo, en el que se tomará un subconjunto aleatorio de individuos de la población, y se seleccionarán para la reproducción los individuos con la mayor aptitud dentro de ese subconjunto.
# Selection function using tournament selection
def selection(population, fitnesses, tournament_size=3):
selected = []
for _ in range(len(population)):
tournament = random.sample(list(zip(population, fitnesses)), tournament_size)
winner = max(tournament, key=lambda x: x[1])[0]
selected.append(winner)
return selectedHaz una función de cruce
A continuación, escribiremos una función rápida para utilizar el cruce para crear nuevas soluciones basadas en soluciones existentes.
# Crossover function
def crossover(parent1, parent2):
alpha = random.random()
child1 = tuple(alpha * p1 + (1 - alpha) * p2 for p1, p2 in zip(parent1, parent2))
child2 = tuple(alpha * p2 + (1 - alpha) * p1 for p1, p2 in zip(parent1, parent2))
return child1, child2Diseña una función de mutación
También necesitamos una función de mutación para introducir aleatoriedad en las generaciones posteriores. Esto es importante para asegurarse de que hay suficiente diversidad en cada generación para seleccionar.
# Mutation function
def mutation(individual, mutation_rate, lower_bound, upper_bound):
individual = list(individual)
for i in range(len(individual)):
if random.random() < mutation_rate:
mutation_amount = random.uniform(-1, 1)
individual[i] += mutation_amount
# Ensure the individual stays within bounds
individual[i] = max(min(individual[i], upper_bound), lower_bound)
return tuple(individual)Bucle principal
A continuación tenemos que crear el bucle principal que unirá todas estas piezas para ejecutar nuestro algoritmo. Como también necesitamos trazar nuestros resultados, añadiré el código de trazado en esta sección, así como el código necesario para crear nuestra tabla final.
# Main genetic algorithm function
def genetic_algorithm(population_size, lower_bound, upper_bound, generations, mutation_rate):
population = create_initial_population(population_size, lower_bound, upper_bound)
# Prepare for plotting
fig, axs = plt.subplots(3, 1, figsize=(12, 18)) # 3 rows, 1 column for subplots
best_performers = []
all_populations = []
# Prepare for table
table = PrettyTable()
table.field_names = ["Generation", "a", "b", "c", "Fitness"]
for generation in range(generations):
fitnesses = [fitness_function(ind) for ind in population]
# Store the best performer of the current generation
best_individual = max(population, key=fitness_function)
best_fitness = fitness_function(best_individual)
best_performers.append((best_individual, best_fitness))
all_populations.append(population[:])
table.add_row([generation + 1, best_individual[0], best_individual[1], best_individual[2], best_fitness])
population = selection(population, fitnesses)
next_population = []
for i in range(0, len(population), 2):
parent1 = population[i]
parent2 = population[i + 1]
child1, child2 = crossover(parent1, parent2)
next_population.append(mutation(child1, mutation_rate, lower_bound, upper_bound))
next_population.append(mutation(child2, mutation_rate, lower_bound, upper_bound))
# Replace the old population with the new one, preserving the best individual
next_population[0] = best_individual
population = next_population
# Print the table
print(table)
# Plot the population of one generation (last generation)
final_population = all_populations[-1]
final_fitnesses = [fitness_function(ind) for ind in final_population]
axs[0].scatter(range(len(final_population)), [ind[0] for ind in final_population], color='blue', label='a')
axs[0].scatter([final_population.index(best_individual)], [best_individual[0]], color='cyan', s=100, label='Best Individual a')
axs[0].set_ylabel('a', color='blue')
axs[0].legend(loc='upper left')
axs[1].scatter(range(len(final_population)), [ind[1] for ind in final_population], color='green', label='b')
axs[1].scatter([final_population.index(best_individual)], [best_individual[1]], color='magenta', s=100, label='Best Individual b')
axs[1].set_ylabel('b', color='green')
axs[1].legend(loc='upper left')
axs[2].scatter(range(len(final_population)), [ind[2] for ind in final_population], color='red', label='c')
axs[2].scatter([final_population.index(best_individual)], [best_individual[2]], color='yellow', s=100, label='Best Individual c')
axs[2].set_ylabel('c', color='red')
axs[2].set_xlabel('Individual Index')
axs[2].legend(loc='upper left')
axs[0].set_title(f'Final Generation ({generations}) Population Solutions')
# Plot the values of a, b, and c over generations
generations_list = range(1, len(best_performers) + 1)
a_values = [ind[0][0] for ind in best_performers]
b_values = [ind[0][1] for ind in best_performers]
c_values = [ind[0][2] for ind in best_performers]
fig, ax = plt.subplots()
ax.plot(generations_list, a_values, label='a', color='blue')
ax.plot(generations_list, b_values, label='b', color='green')
ax.plot(generations_list, c_values, label='c', color='red')
ax.set_xlabel('Generation')
ax.set_ylabel('Parameter Values')
ax.set_title('Parameter Values Over Generations')
ax.legend()
# Plot the fitness values over generations
best_fitness_values = [fit[1] for fit in best_performers]
min_fitness_values = [min([fitness_function(ind) for ind in population]) for population in all_populations]
max_fitness_values = [max([fitness_function(ind) for ind in population]) for population in all_populations]
fig, ax = plt.subplots()
ax.plot(generations_list, best_fitness_values, label='Best Fitness', color='black')
ax.fill_between(generations_list, min_fitness_values, max_fitness_values, color='gray', alpha=0.5, label='Fitness Range')
ax.set_xlabel('Generation')
ax.set_ylabel('Fitness')
ax.set_title('Fitness Over Generations')
ax.legend()
# Plot the quadratic function for each generation
fig, ax = plt.subplots()
colors = plt.cm.viridis(np.linspace(0, 1, generations))
for i, (best_ind, best_fit) in enumerate(best_performers):
color = colors[i]
a, b, c = best_ind
x_range = np.linspace(lower_bound, upper_bound, 400)
y_values = a * (x_range ** 2) + b * x_range + c
ax.plot(x_range, y_values, color=color)
ax.set_xlabel('x')
ax.set_ylabel('y')
ax.set_title('Quadratic Function')
# Create a subplot for the colorbar
cax = fig.add_axes([0.92, 0.2, 0.02, 0.6]) # [left, bottom, width, height]
norm = plt.cm.colors.Normalize(vmin=0, vmax=generations)
sm = plt.cm.ScalarMappable(cmap='viridis', norm=norm)
sm.set_array([])
fig.colorbar(sm, ax=ax, cax=cax, orientation='vertical', label='Generation')
plt.show()
return max(population, key=fitness_function)Hacer que todo funcione
Por último, tenemos que configurar los parámetros de nuestro algoritmo y ejecutarlo.
# Parameters for the genetic algorithm
population_size = 100
lower_bound = -50
upper_bound = 50
generations = 20
mutation_rate = 1
# Run the genetic algorithm
best_solution = genetic_algorithm(population_size, lower_bound, upper_bound, generations, mutation_rate)
print(f"Best solution found: a = {best_solution[0]}, b = {best_solution[1]}, c = {best_solution[2]}")Condición de finalización (opcional)
Ahora, en este ejemplo, fijamos un número concreto de generaciones para que se ejecute nuestro algoritmo. Sin embargo, también podríamos haber establecido una condición de finalización que detuviera el programa cuando alcanzáramos un valor objetivo de aptitud. Podría ser algo así:
def termination_condition(fitnesses, target_fitness):
return max(fitnesses) >= target_fitnessPonerlo todo junto
Aquí tienes el código completo
import random
import matplotlib.pyplot as plt
import numpy as np
from prettytable import PrettyTable
# Define the fitness function (objective is to create the flattest U-shape)
def fitness_function(params):
a, b, c = params
if a <= 0:
return -float('inf') # Penalize downward facing u-shapes heavily
vertex_x = -b / (2 * a) #x value at vertex
vertex_y = a * (vertex_x ** 2) + b * vertex_x + c #y value at vertex
y_left = a * (-1) ** 2 + b * (-1) + c #y-coordinate at x = -1
y_right = a * (1) ** 2 + b * (1) + c #y-coordinate at x = 1
curviness = abs(y_left - vertex_y) + abs(y_right - vertex_y)
return -curviness # Negate to minimize curviness
# Create the initial population
def create_initial_population(size, lower_bound, upper_bound):
population = []
for _ in range(size):
individual = (random.uniform(lower_bound, upper_bound),
random.uniform(lower_bound, upper_bound),
random.uniform(lower_bound, upper_bound))
population.append(individual)
return population
# Selection function using tournament selection
def selection(population, fitnesses, tournament_size=3):
selected = []
for _ in range(len(population)):
tournament = random.sample(list(zip(population, fitnesses)), tournament_size)
winner = max(tournament, key=lambda x: x[1])[0]
selected.append(winner)
return selected
# Crossover function
def crossover(parent1, parent2):
alpha = random.random()
child1 = tuple(alpha * p1 + (1 - alpha) * p2 for p1, p2 in zip(parent1, parent2))
child2 = tuple(alpha * p2 + (1 - alpha) * p1 for p1, p2 in zip(parent1, parent2))
return child1, child2
# Mutation function
def mutation(individual, mutation_rate, lower_bound, upper_bound):
individual = list(individual)
for i in range(len(individual)):
if random.random() < mutation_rate:
mutation_amount = random.uniform(-1, 1)
individual[i] += mutation_amount
# Ensure the individual stays within bounds
individual[i] = max(min(individual[i], upper_bound), lower_bound)
return tuple(individual)
# Main genetic algorithm function
def genetic_algorithm(population_size, lower_bound, upper_bound, generations, mutation_rate):
population = create_initial_population(population_size, lower_bound, upper_bound)
# Prepare for plotting
fig, axs = plt.subplots(3, 1, figsize=(12, 18)) # 3 rows, 1 column for subplots
best_performers = []
all_populations = []
# Prepare for table
table = PrettyTable()
table.field_names = ["Generation", "a", "b", "c", "Fitness"]
for generation in range(generations):
fitnesses = [fitness_function(ind) for ind in population]
# Store the best performer of the current generation
best_individual = max(population, key=fitness_function)
best_fitness = fitness_function(best_individual)
best_performers.append((best_individual, best_fitness))
all_populations.append(population[:])
table.add_row([generation + 1, best_individual[0], best_individual[1], best_individual[2], best_fitness])
population = selection(population, fitnesses)
next_population = []
for i in range(0, len(population), 2):
parent1 = population[i]
parent2 = population[i + 1]
child1, child2 = crossover(parent1, parent2)
next_population.append(mutation(child1, mutation_rate, lower_bound, upper_bound))
next_population.append(mutation(child2, mutation_rate, lower_bound, upper_bound))
# Replace the old population with the new one, preserving the best individual
next_population[0] = best_individual
population = next_population
# Print the table
print(table)
# Plot the population of one generation (last generation)
final_population = all_populations[-1]
final_fitnesses = [fitness_function(ind) for ind in final_population]
axs[0].scatter(range(len(final_population)), [ind[0] for ind in final_population], color='blue', label='a')
axs[0].scatter([final_population.index(best_individual)], [best_individual[0]], color='cyan', s=100, label='Best Individual a')
axs[0].set_ylabel('a', color='blue')
axs[0].legend(loc='upper left')
axs[1].scatter(range(len(final_population)), [ind[1] for ind in final_population], color='green', label='b')
axs[1].scatter([final_population.index(best_individual)], [best_individual[1]], color='magenta', s=100, label='Best Individual b')
axs[1].set_ylabel('b', color='green')
axs[1].legend(loc='upper left')
axs[2].scatter(range(len(final_population)), [ind[2] for ind in final_population], color='red', label='c')
axs[2].scatter([final_population.index(best_individual)], [best_individual[2]], color='yellow', s=100, label='Best Individual c')
axs[2].set_ylabel('c', color='red')
axs[2].set_xlabel('Individual Index')
axs[2].legend(loc='upper left')
axs[0].set_title(f'Final Generation ({generations}) Population Solutions')
# Plot the values of a, b, and c over generations
generations_list = range(1, len(best_performers) + 1)
a_values = [ind[0][0] for ind in best_performers]
b_values = [ind[0][1] for ind in best_performers]
c_values = [ind[0][2] for ind in best_performers]
fig, ax = plt.subplots()
ax.plot(generations_list, a_values, label='a', color='blue')
ax.plot(generations_list, b_values, label='b', color='green')
ax.plot(generations_list, c_values, label='c', color='red')
ax.set_xlabel('Generation')
ax.set_ylabel('Parameter Values')
ax.set_title('Parameter Values Over Generations')
ax.legend()
# Plot the fitness values over generations
best_fitness_values = [fit[1] for fit in best_performers]
min_fitness_values = [min([fitness_function(ind) for ind in population]) for population in all_populations]
max_fitness_values = [max([fitness_function(ind) for ind in population]) for population in all_populations]
fig, ax = plt.subplots()
ax.plot(generations_list, best_fitness_values, label='Best Fitness', color='black')
ax.fill_between(generations_list, min_fitness_values, max_fitness_values, color='gray', alpha=0.5, label='Fitness Range')
ax.set_xlabel('Generation')
ax.set_ylabel('Fitness')
ax.set_title('Fitness Over Generations')
ax.legend()
# Plot the quadratic function for each generation
fig, ax = plt.subplots()
colors = plt.cm.viridis(np.linspace(0, 1, generations))
for i, (best_ind, best_fit) in enumerate(best_performers):
color = colors[i]
a, b, c = best_ind
x_range = np.linspace(lower_bound, upper_bound, 400)
y_values = a * (x_range ** 2) + b * x_range + c
ax.plot(x_range, y_values, color=color)
ax.set_xlabel('x')
ax.set_ylabel('y')
ax.set_title('Quadratic Function')
# Create a subplot for the colorbar
cax = fig.add_axes([0.92, 0.2, 0.02, 0.6]) # [left, bottom, width, height]
norm = plt.cm.colors.Normalize(vmin=0, vmax=generations)
sm = plt.cm.ScalarMappable(cmap='viridis', norm=norm)
sm.set_array([])
fig.colorbar(sm, ax=ax, cax=cax, orientation='vertical', label='Generation')
plt.show()
return max(population, key=fitness_function)
# Parameters for the genetic algorithm
population_size = 100
lower_bound = -50
upper_bound = 50
generations = 20
mutation_rate = 1
# Run the genetic algorithm
best_solution = genetic_algorithm(population_size, lower_bound, upper_bound, generations, mutation_rate)
print(f"Best solution found: a = {best_solution[0]}, b = {best_solution[1]}, c = {best_solution[2]}")Evaluación de los resultados
Utilicemos nuestros resultados para ver cómo ha funcionado nuestra función.
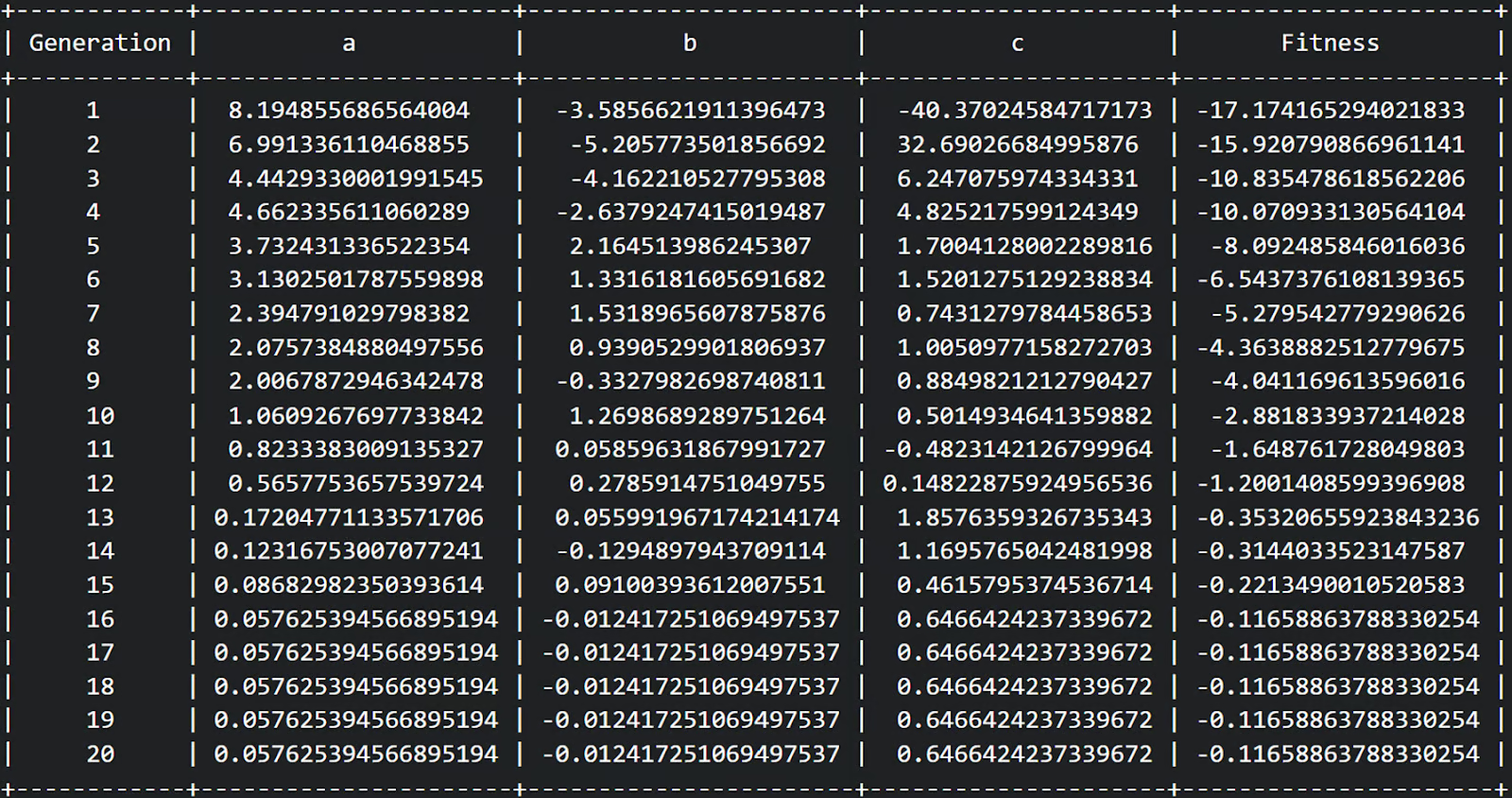
En primer lugar, podemos ver en nuestra tabla que completamos 20 generaciones y que la aptitud parecía aumentar de una aptitud relativamente negativa a una aptitud menos negativa a lo largo de cada generación. ¡Estupendo!
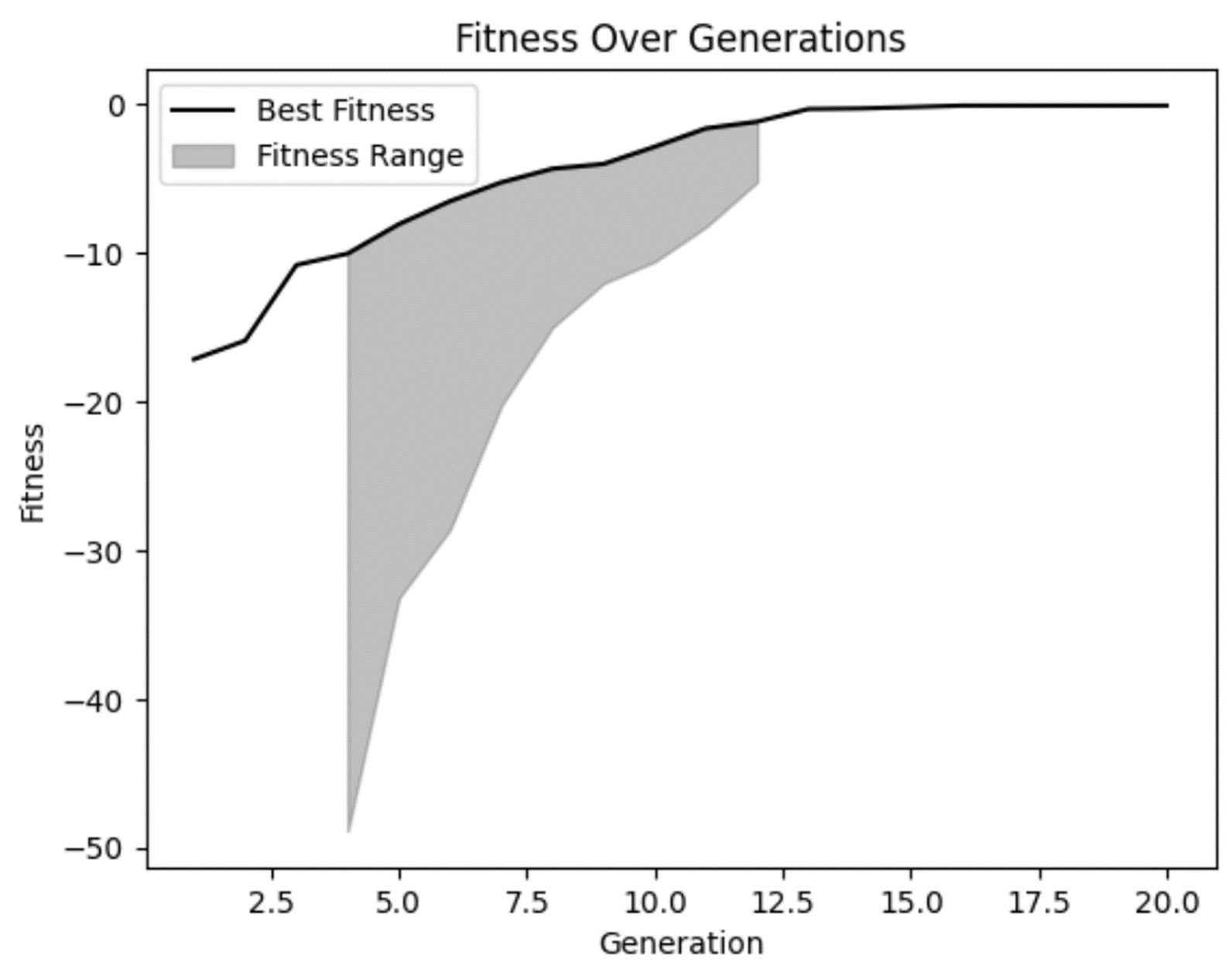
A partir de nuestro gráfico de aptitud, podemos confirmar nuestras sospechas de que la aptitud mejoró con el tiempo y parece haberse aplanado un poco en las generaciones posteriores. Pero también podemos ver que en algunas de las primeras generaciones había algunas soluciones realmente malas que nuestro modelo rechazó acertadamente.
También podemos ver que en varias generaciones no había mucha diversidad en el rango de aptitud. Aunque eso no es necesariamente malo, es algo que hay que vigilar. En algunos casos, esto puede ser un indicador de convergencia prematura, que es un problema que se produce cuando no hay suficiente diversidad en tu población.
A continuación, tracemos nuestras ecuaciones de cada generación para ver si realmente las hemos aplanado.

Vemos que nuestras soluciones de primera generación son muy curvas, lo cual tiene sentido. Esta ecuación produce un gráfico en forma de U. Pero también podemos ver que las generaciones futuras se hicieron cada vez más planas. ¡Esto es exactamente lo que buscábamos!
Ahora bien, hay que señalar que sólo buscamos explícitamente dentro de nuestros límites establecidos de -50 - 50. Si ampliáramos este gráfico, se vería que incluso estas líneas aparentemente planas siguen teniendo forma de U, sólo que con bases más anchas.
Este ejemplo pone de relieve la importancia de ser cautelosos con nuestros supuestos en los análisis para asegurarnos de que apoyan nuestras predicciones. Nuestro modelo hizo exactamente lo que queríamos, pero sólo dentro de los límites que le dimos. Si quisiéramos encontrar una combinación de variables que creara una forma plana más alejada, tendríamos que volver a ejecutar el modelo con límites más amplios.
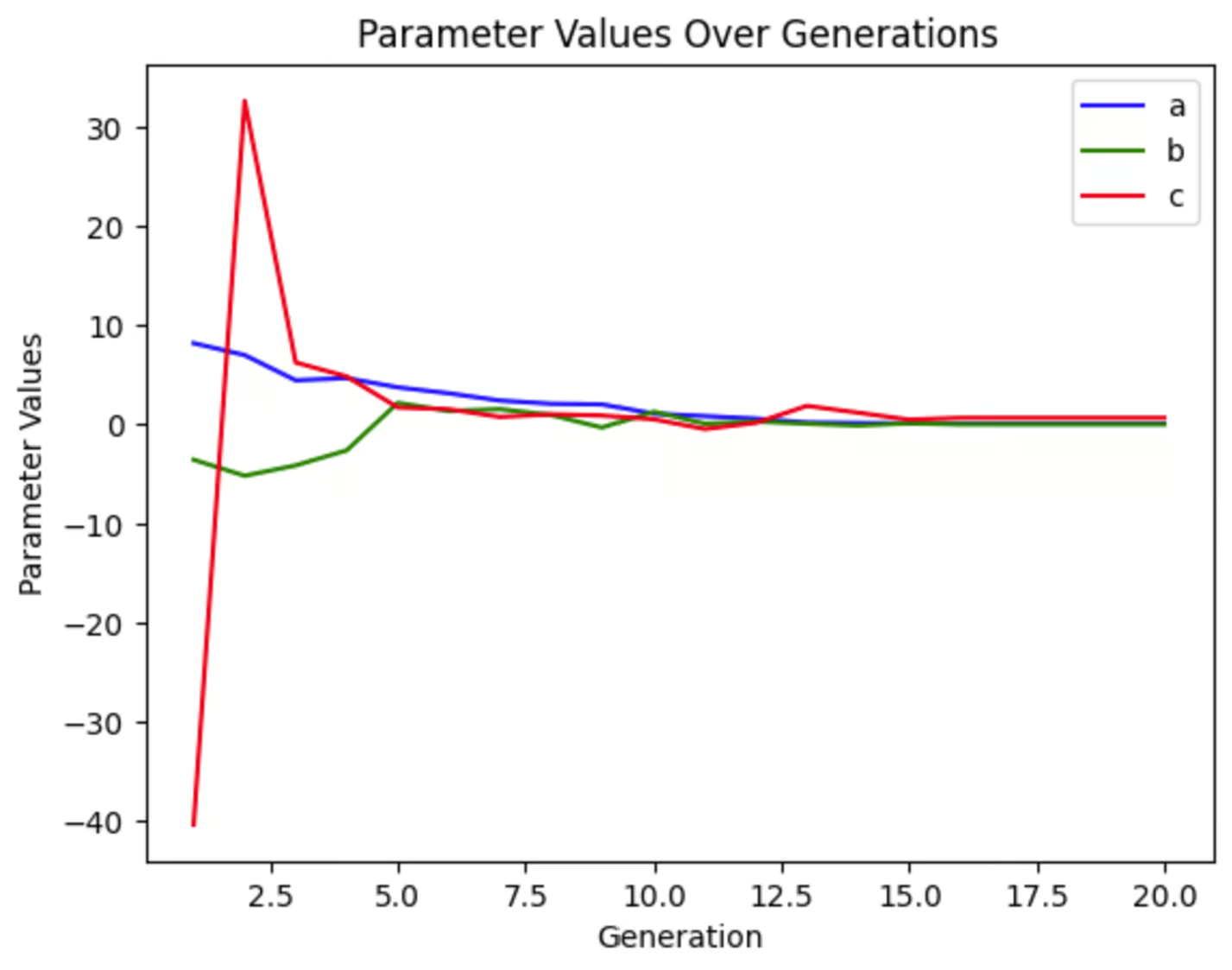
Podemos trazar cada una de las variables para ver cuánto han cambiado a lo largo de las generaciones. Podemos ver que en esta ejecución de la simulación, c varió mucho al principio, mientras que a y b cambiaron mucho menos drásticamente. Este gráfico tendrá un aspecto diferente cada vez que ejecutemos esta simulación, aunque no cambiemos ningún parámetro, porque nuestra población inicial es aleatoria.
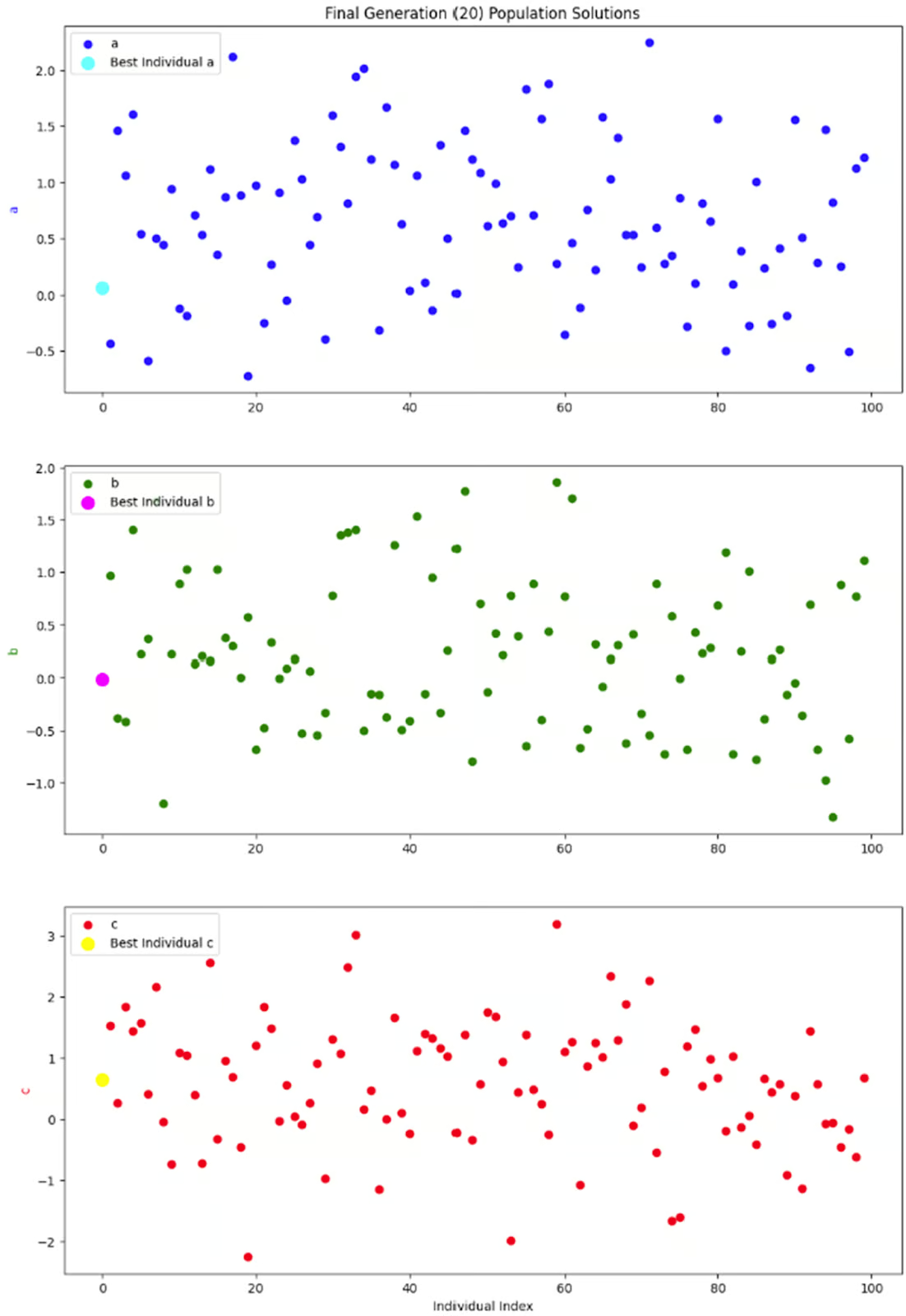
El último gráfico que quiero ver es una instantánea de toda la población en una generación: la generación final. Como cada individuo de la población tiene tres parámetros, decidí separarlos en tres subparcelas, en lugar de complicarme con gráficos tridimensionales.
Lo que realmente quiero ver en estos gráficos es si hay una buena diversidad dentro de esta generación. Sin una buena cantidad de diversidad, podríamos llegar a una situación en la que las únicas soluciones disponibles fueran aquellas con valores de aptitud bajos, lo que provocaría una convergencia prematura. Pero parece que este gráfico muestra una buena cantidad de diversidad, así que estoy contento con este resultado.
Consideraciones y consejos
Los algoritmos genéticos pueden utilizarse en una gran variedad de situaciones. Pero hay que tener en cuenta algunas consideraciones.
Ajuste de parámetros
Como ocurre con cualquier modelo, el rendimiento de un algoritmo genético depende de varios parámetros, sobre todo del tamaño de la población, la tasa de cruce, la tasa de mutación y los parámetros de delimitación. Si cambias estos parámetros, cambiará el rendimiento de tu modelo.
Como regla general, los tamaños de población mayores te ayudarán a encontrar la solución óptima más rápidamente, porque hay más opciones entre las que elegir. Sin embargo, los tamaños de población más grandes también requieren más tiempo y recursos para funcionar.
Una tasa de cruce más alta puede conducir a una convergencia más rápida al combinar rasgos beneficiosos de distintos individuos con mayor frecuencia. Sin embargo, una tasa de cruce demasiado alta podría alterar la estructura de la población, provocando una convergencia prematura.
Una tasa de mutación más alta ayuda a mantener la diversidad genética, evitando que el algoritmo se atasque en un óptimo local. Sin embargo, si la tasa de mutación es demasiado alta, puede perturbar el proceso de convergencia al introducir demasiada aleatoriedad, dificultando que el algoritmo perfeccione las soluciones.
Los parámetros de delimitación definen el rango dentro del cual el algoritmo busca soluciones. Es importante adaptarlos al problema concreto de tu empresa. Un área de delimitación demasiado estrecha puede pasar por alto soluciones óptimas a tu problema. Un área de delimitación demasiado amplia necesitará más tiempo y recursos para ejecutarse. Pero también hay otras consideraciones.
Por ejemplo, en nuestro ejemplo codificado anterior, esa ecuación teóricamente no tiene límites. Pero en la práctica, no podemos pedir al ordenador que encuentre el arco más plano en un grafo infinitamente grande. Por tanto, los límites son necesarios. Pero cambiar esos límites también cambiará la respuesta óptima. En mi opinión, establecer límites adecuados para tu caso de uso específico es imprescindible con cualquier modelo.
Puede haber otros parámetros importantes que afinar en tu modelo concreto. Experimenta con distintos valores para encontrar los mejores ajustes para tu problema. Para saber cómo afinar específicamente los AG, consulta Métodos Informados: Algoritmos genéticos.
Codificación
La codificación es un paso importante en la creación de un algoritmo genético. Consiste en convertir las posibles soluciones en un formato que el algoritmo pueda procesar. Por ejemplo, supongamos que estamos diseñando un algoritmo genético para optimizar las rutas de reparto de nuestros camiones. ¿Cómo se convierte una lista de lugares de entrega en un cromosoma para introducirlo en un AG? La codificación transforma esta lista en una secuencia que el algoritmo puede manipular, como una lista ordenada de lugares que representan una posible ruta de entrega.
Una codificación adecuada garantiza que el algoritmo genético pueda explorar eficazmente el espacio de soluciones y combinar o mutar soluciones de forma significativa. Sin ella, el algoritmo no podría manejar datos complejos. Datos como los lugares de entrega, los problemas de programación o las interacciones con el servicio de atención al cliente sólo son accesibles a los ordenadores mediante la codificación.
Puedes elegir entre varios esquemas de codificación. Puedes obtener más información para decidir cuál puede ser el mejor para tu situación en Trabajar con datos categóricos en Python o Cómo convertir cadenas en bytes en Python.
Convergencia prematura
La convergencia prematura es un problema que se produce cuando la población se vuelve demasiado similar. Esto puede hacer que el algoritmo se atasque en un óptimo local en lugar de encontrar el óptimo global. Esencialmente, si no hay suficiente diversidad en la población, las soluciones se vuelven "endogámicas" y no obtienes la mejor solución.
Hay algunas técnicas que puedes probar para evitar la convergencia prematura. Una tasa de mutación más alta puede introducir más diversidad en la población, ayudando a explorar nuevas áreas del espacio de soluciones. También puedes intentar empezar con una población inicial más diversa para garantizar una amplia exploración del espacio de soluciones desde el principio.
Si sigues teniendo problemas de convergencia prematura, puedes probar a ajustar dinámicamente parámetros como la tasa de mutación y la tasa de cruce en función del progreso del algoritmo. Como alternativa, puedes utilizar varias poblaciones (piensa en ellas como si fueran islas) que intercambien ocasionalmente individuos para mantener la diversidad.
Puedes obtener más información sobre la convergencia prematura y otros errores del aprendizaje automático en la sección Supervisión de conceptos de aprendizaje automático curso.
Elitismo
Por otra parte, un cruce y mutación fuertes o cualquier aleatoriedad en la selección pueden hacer que las grandes soluciones no acaben reproduciéndose. Ahí es donde puede entrar el elitismo. El elitismo es una técnica en la que los mejores individuos pasan directamente a la siguiente generación para preservar las buenas soluciones. Esto ayuda a que las mejores soluciones no se pierdan durante el proceso evolutivo.
Sin embargo, ten cuidado si utilizas el elitismo. Si se utiliza el elitismo sin una diversidad de población lo suficientemente alta, puedes acabar con una convergencia prematura. Si tu problema empresarial requiere técnicas de elitismo, asegúrate de combinarlas con funciones de cruce y mutación suficientemente potentes y con un tamaño de población grande. Este enfoque ayuda a mantener un equilibrio saludable entre la explotación de soluciones conocidas y la exploración de nuevas posibilidades.
Conclusión
Los algoritmos genéticos son un ejemplo fantástico de ciencia de datos que se inspira en el mundo natural. Ofrecen un potente método para resolver problemas complejos de optimización imitando el proceso de selección natural.
Si te interesan otros modelos inspirados en la biología, aprende sobre redes neuronales y otras técnicas de aprendizaje profundo en Introducción al aprendizaje profundo en Python. También te puede interesar el curso de DataCamp Conceptos de IA Generativa y Ética de la IA.
Si te animas a hacer un proyecto con datos biológicos, consulta Análisis de datos genómicos en R o Análisis de imágenes biomédicas en Python.
Obtén una certificación superior en IA

Soy doctor con 13 años de experiencia trabajando con datos en un entorno de investigación biológica. Creo software en varios lenguajes de programación, como Python, MATLAB y R. Me apasiona compartir mi amor por el aprendizaje con el mundo.