Kurs
MedGemma ist eine Sammlung von Gemma 3-Varianten die für das Verstehen von medizinischen Texten und Bildern entwickelt wurde. Die Sammlung umfasst derzeit zwei leistungsstarke Varianten: eine 4Bmultimodale Version und eine 27B reine Textversion .
Das MedGemma 4B Modell kombiniert die SigLIP Bildkodierer, der auf verschiedenen, nicht identifizierten medizinischen Datensätzen wie Röntgenbildern, dermatologischen Bildern, ophthalmologischen Bildern und histopathologischen Präparaten trainiert wurde, mit einem großen Sprachmodell (LLM), das auf einer Vielzahl von medizinischen Daten trainiert wurde.
In diesem Lernprogramm lernen wir, wie man Feinabstimmung des MedGemma 4B-Modells an einem MRT-Datensatz des Gehirns für eine Bildklassifizierungsaufgabe. Ziel ist es, das kleinere MedGemma 4B-Modell anzupassen, um MRT-Scans des Gehirns effektiv zu klassifizieren und Hirntumore mit verbesserter Genauigkeit und Effizienz vorherzusagen.
Einrichten der Umgebung
RunPod ist eine hervorragende Plattform für GPU-basierte Workloads und bietet vorkonfigurierte Umgebungen mit JupyterLab-Unterstützung. So kannst du einen Pod starten und sofort mit deinem bevorzugten Editor programmieren. Hier erfährst du, wie du deine Umgebung einrichtest:
1. Einrichten des RunPods
Einloggen bei RunPod und erstelle einen Pod mit einer A100 GPU und dem neuesten PyTorch-Image. Dann klickst du auf die Schaltfläche "On-Demand bereitstellen", um den Pod zu starten.
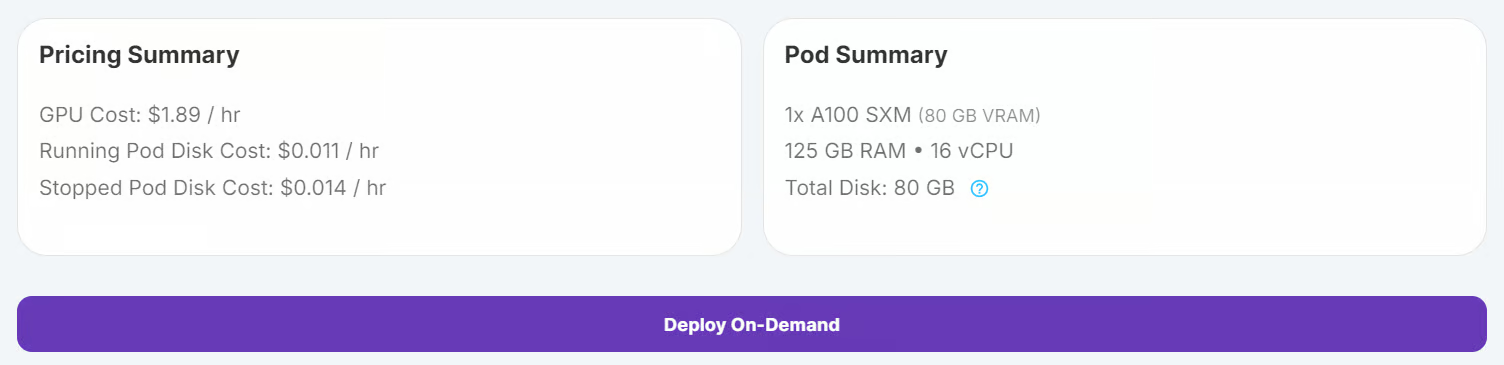
Bearbeite den Pod und füge die folgenden Umgebungsvariablen für die Integration von Hugging Face und Kaggle hinzu:
- HF_TOKEN
- KAGGLE_USERNAME
- KAGGLE_KEY
Erhöhe die Speicherkapazität auf 100 GB, um Datensätze und Modell-Checkpoints unterzubringen
2. Installation der Python-Pakete
Sobald der Pod läuft, starte die JupyterLab-Instanz, indem du auf die Schaltfläche "Verbinden" klickst. Erstelle ein neues Python-Notebook und installiere die benötigten Python-Pakete, indem du den folgenden Befehl ausführst:
! pip install --upgrade --quiet transformers bitsandbytes datasets evaluate peft trl scikit-learn kaggleLaden und Verarbeiten des Datensatzes
Wir werden den Gehirnkrebs MRT-Datensatz von Kaggle.
3. Herunterladen des Datensatzes
Lade den Datensatz herunter und entpacke ihn mit dem Kaggle CLI:
!kaggle datasets download -d orvile/brain-cancer-mri-dataset --unzipDataset URL: https://www.kaggle.com/datasets/orvile/brain-cancer-mri-dataset
License(s): CC-BY-SA-4.0
Downloading brain-cancer-mri-dataset.zip to /workspace
90%|████████████████████████████████████▉ | 130M/144M [00:00<00:00, 231MB/s]
100%|█████████████████████████████████████████| 144M/144M [00:01<00:00, 108MB/s]4. Laden des Datensatzes
Lade den Datensatz als Hugging Face-Datensatz, unterteile ihn in Trainings- und Validierungssätze und zeige seine Struktur an:
from datasets import load_dataset
data_dir = "./Brain_Cancer raw MRI data/Brain_Cancer"
# Define proportions for train and validation splits
train_size = 0.8
validation_size = 0.2
data = load_dataset("imagefolder", data_dir=data_dir, split="train")
# Split the dataset into train and validation sets
data = data.train_test_split(
train_size=train_size,
test_size=validation_size,
shuffle=True,
seed=42,
)
# Rename the 'test' split to 'validation'
data["validation"] = data.pop("test")
# Display dataset details
print(data)Ausgabe:
DatasetDict({
train: Dataset({
features: ['image', 'label'],
num_rows: 4844
})
validation: Dataset({
features: ['image', 'label'],
num_rows: 1212
})
})Überprüfe das erste Bild und das dazugehörige Label aus dem Trainingsset:
data["train"][0]["image"]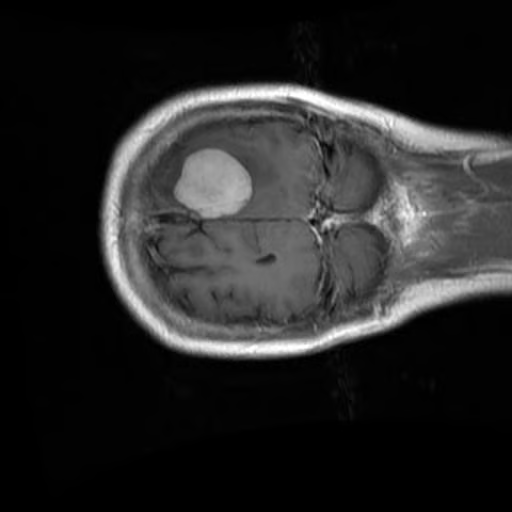
print(data["train"][0]["label"])15. Verarbeitung des Datensatzes
Bevor du den Datensatz bearbeitest, ist es wichtig, die Namen der Labels zu überprüfen, damit die Klassifizierungsaufgabe richtig bearbeitet werden kann.
BRAIN_CANCER_CLASSES = data["train"].features["label"].names
print("Detected classes:", BRAIN_CANCER_CLASSES)Die erkannten Klassen sind: ['brain_glioma', 'brain_menin', 'brain_tumor'].
Detected classes: ['brain_glioma', 'brain_menin', 'brain_tumor']Um den Klassifizierungsprozess zu verbessern, werden wir diese Klassenbezeichnungen ändern, indem wir ein Präfix (A, B, C) hinzufügen, um sie besser zu organisieren und an ein benutzerdefiniertes Prompt-Format anzupassen.
BRAIN_CANCER_CLASSES = ['A: brain glioma', 'B: brain menin', 'C: brain tumor']Als Nächstes erstellen wir eine benutzerdefinierte Eingabeaufforderung, die das Modell während der Feinabstimmung anleitet. Die Eingabeaufforderung enthält die aktualisierten Klassenbezeichnungen.
options = "\n".join(BRAIN_CANCER_CLASSES)
PROMPT = f"What is the most likely type of brain cancer shown in the MRI image?\n{options}"Um den Datensatz für die Feinabstimmung vorzubereiten, erstellen wir eine neue Spalte namens "Nachrichten". Diese Spalte enthält strukturierte Daten, die eine Benutzerabfrage (die Aufforderung) und die Antwort des Assistenten (die richtige Bezeichnung) darstellen.
def format_data(example: dict[str, any]) -> dict[str, any]:
example["messages"] = [
{
"role": "user",
"content": [
{
"type": "image",
},
{
"type": "text",
"text": PROMPT,
},
],
},
{
"role": "assistant",
"content": [
{
"type": "text",
"text": BRAIN_CANCER_CLASSES[example["label"]],
},
],
},
]
return example
# Apply the formatting to the dataset
formatted_data = data.map(format_data)Um die Formatierung zu überprüfen, können wir uns die Nachrichtenspalte aus dem ersten Beispiel ansehen:
formatted_data["train"][0]["messages"]Der resultierende formatierte Datenpunkt sieht wie folgt aus:
[{'content': [{'text': None, 'type': 'image'},
{'text': 'What is the most likely type of brain cancer shown in the MRI image?\nA: brain glioma\nB: brain menin\nC: brain tumor',
'type': 'text'}],
'role': 'user'},
{'content': [{'text': 'B: brain menin', 'type': 'text'}],
'role': 'assistant'}]Feinabstimmung von MedGemma mit dem Gehirn-MRT-Datensatz
In diesem Abschnitt werden wir das MedGemma 4B Instruct-Modell mit dem Gehirn-MRT-Datensatz feinabstimmen. Dazu musst du das Modell und den Prozessor herunterladen, den LoRA-Adapter einrichten, den Trainer konfigurieren und den Trainingsprozess starten.
6. Laden des Modells und des Tokenizers
Da MedGemma ein Gated Model ist, musst du dich bei der Hugging Face CLI mit deinem API-Schlüssel anmelden. So kannst du dein fein abgestimmtes Modell auch im Hugging Face Hub speichern. Schau dir unseren Kurs an: Arbeiten mit Hugging Face: Dein Leitfaden für den Hub, falls du eine Auffrischung brauchst .
from huggingface_hub import login
import os
hf_token = os.environ.get("HF_TOKEN")
login(hf_token)Wir verwenden die Transformers-Bibliothek, um das MedGemma 4B Instruct-Modell und seinen Prozessor zu laden. Das Modell ist so konfiguriert, dass es die Genauigkeit von bfloat16 für effiziente Berechnungen auf GPUs verwendet.
import torch
from transformers import AutoProcessor, AutoModelForImageTextToText, BitsAndBytesConfig
model_id = "google/medgemma-4b-it"
# Check if GPU supports bfloat16
if torch.cuda.get_device_capability()[0] < 8:
raise ValueError("GPU does not support bfloat16, please use a GPU that supports bfloat16.")
model_kwargs = dict(
attn_implementation="eager",
torch_dtype=torch.bfloat16,
device_map="auto",
)
model = AutoModelForImageTextToText.from_pretrained(model_id, **model_kwargs)
processor = AutoProcessor.from_pretrained(model_id)
# Use right padding to avoid issues during training
processor.tokenizer.padding_side = "right"7. Einrichten des Modells
Zur effizienten Feinabstimmung des MedGemma 4B Instruct-Modells werden wir Folgendes verwenden Low-Rank Adaptation (LoRA)eine parameter-effiziente Methode zur Feinabstimmung.
Mit LoRA können wir große Modelle anpassen, indem wir nur eine kleine Anzahl zusätzlicher Parameter trainieren und so die Rechenkosten bei gleichbleibender Leistung deutlich senken.
from peft import LoraConfig
peft_config = LoraConfig(
lora_alpha=16,
lora_dropout=0.05,
r=16,
bias="none",
target_modules="all-linear",
task_type="CAUSAL_LM",
modules_to_save=[
"lm_head",
"embed_tokens",
],
)Um sowohl Bild- als auch Texteingaben während des Trainings zu verarbeiten, definieren wir eine eigene Sortierfunktion. Diese Funktion verarbeitet die Datensatzbeispiele in ein für das Modell geeignetes Format, einschließlich der Tokenisierung von Text und der Aufbereitung von Bilddaten.
def collate_fn(examples: list[dict[str, any]]):
texts = []
images = []
for example in examples:
images.append([example["image"]])
texts.append(
processor.apply_chat_template(
example["messages"], add_generation_prompt=False, tokenize=False
).strip()
)
# Tokenize the texts and process the images
batch = processor(text=texts, images=images, return_tensors="pt", padding=True)
# The labels are the input_ids, with the padding and image tokens masked in
# the loss computation
labels = batch["input_ids"].clone()
# Mask image tokens
image_token_id = [
processor.tokenizer.convert_tokens_to_ids(
processor.tokenizer.special_tokens_map["boi_token"]
)
]
# Mask tokens that are not used in the loss computation
labels[labels == processor.tokenizer.pad_token_id] = -100
labels[labels == image_token_id] = -100
labels[labels == 262144] = -100
batch["labels"] = labels
return batchWir verwenden die Klasse SFTConfig aus der Bibliothektrl, um die Trainingsargumente zu definieren. Diese Argumente steuern den Feinabstimmungsprozess, einschließlich Stapelgröße, Lernrate und Gradientenakkumulationsschritte.
from trl import SFTConfig
args = SFTConfig(
output_dir="medgemma-brain-cancer",
num_train_epochs=1,
per_device_train_batch_size=8,
per_device_eval_batch_size=8,
gradient_accumulation_steps=8,
gradient_checkpointing=True,
optim="adamw_torch_fused",
logging_steps=0.1,
save_strategy="epoch",
eval_strategy="steps",
eval_steps=0.1,
learning_rate=2e-4,
bf16=True,
max_grad_norm=0.3,
warmup_ratio=0.03,
lr_scheduler_type="linear",
push_to_hub=True,
report_to="none",
gradient_checkpointing_kwargs={"use_reentrant": False},
dataset_kwargs={"skip_prepare_dataset": True},
remove_unused_columns = False,
label_names=["labels"],
)Der SFTTrainer vereinfacht den Feinabstimmungsprozess, indem er das Modell, den Datensatz, den Datensammler, die Trainingsargumente und die LoRA-Konfiguration in einem einzigen Arbeitsablauf vereint. Das macht den Prozess schlank und benutzerfreundlich.
from trl import SFTTrainer
trainer = SFTTrainer(
model=model,
args=args,
train_dataset=formatted_data["train"],
eval_dataset=formatted_data["validation"].shuffle().select(range(50)),
peft_config=peft_config,
processing_class=processor,
data_collator=collate_fn,
)8. Model Ausbildung
Sobald das Modell, der Datensatz und die Trainingskonfigurationen eingerichtet sind, können wir mit der Feinabstimmung beginnen. Der SFTTrainer vereinfacht diesen Schritt und ermöglicht es uns, das Modell mit nur einem einzigen Befehl zu trainieren:
trainer.train()Die Ausbildung dauerte etwa 1 Stunde und 8 Minuten. Während dieser Zeit sanken der Trainingsverlust und der Validierungsverlust mit jedem Schritt, was darauf hindeutet, dass das Modell effektiv lernt.
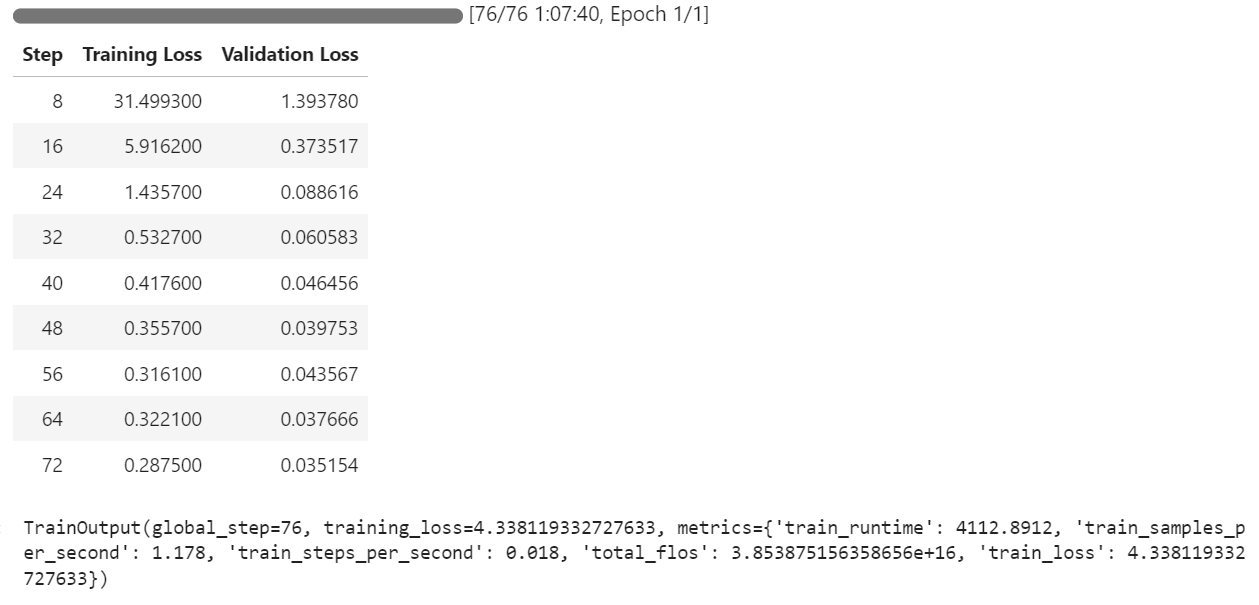
Nachdem das Training abgeschlossen ist, kann das fein abgestimmte Modell lokal gespeichert und mit der Methode save_model() an den Hugging Face Hub übertragen werden.
trainer.save_model()Das Modell ist jetzt verfügbar kingabzpro/medgemma-brain-cancer - Hugging Face
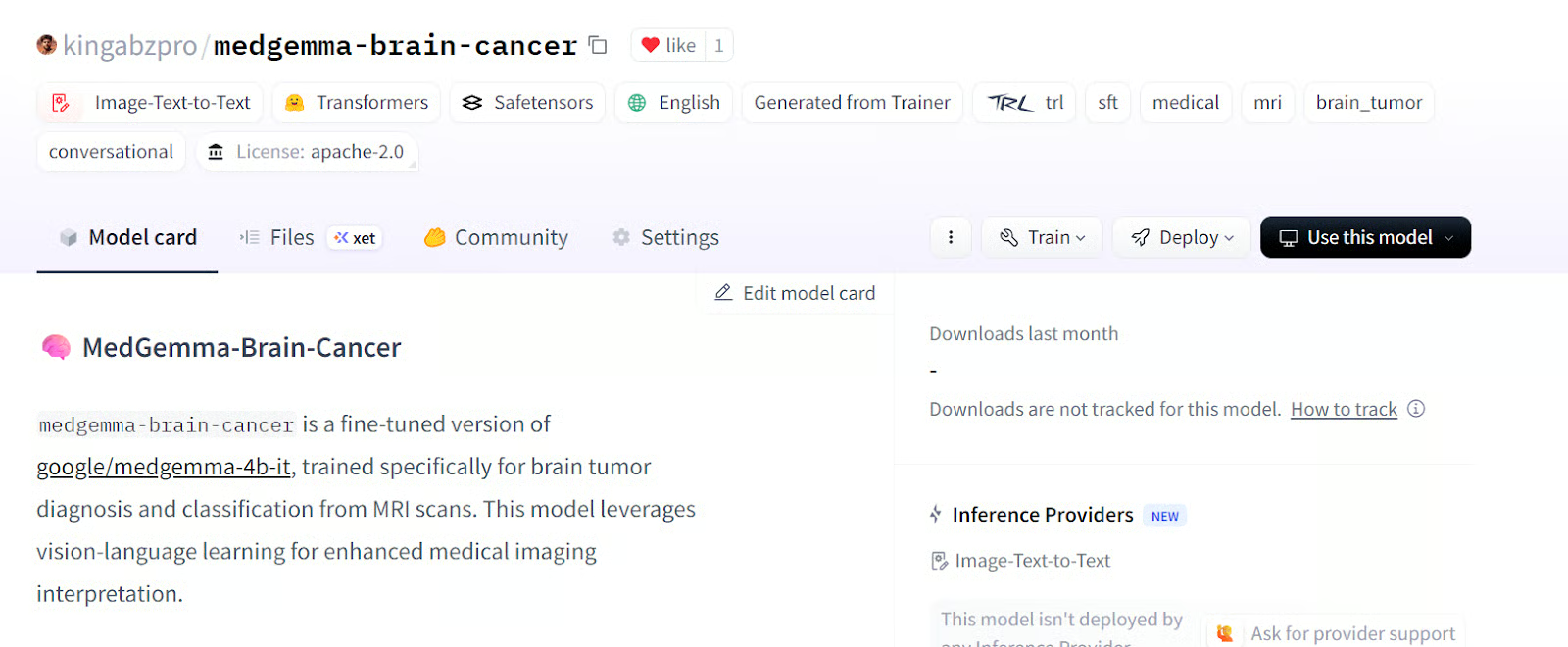
Quelle kingabzpro/medgemma-hirn-krebs
Modellbewertung
Um die Leistung des MedGemma 4B-Modells zu bewerten, testen wir sowohl das Basismodell als auch das fein abgestimmte Modell auf dem Validierungsdatensatz. Dieser Prozess umfasst das Löschen des Speichers, die Vorbereitung der Testdaten, die Generierung der Antwort und die Berechnung von Schlüsselkennzahlen wie Genauigkeit und F1-Punktzahl.
Bevor wir mit der Auswertung beginnen, entfernen wir die Trainingseinstellungen, um GPU-Speicher freizugeben und eine saubere Umgebung für die Tests zu gewährleisten.
del model
del trainer
torch.cuda.empty_cache()9. Vorbereitungen für Modellversuche
Wir formatieren den Validierungsdatensatz so, dass er der vom Modell benötigten Eingabestruktur entspricht. Dazu musst du eine Spalte "Meldungen" erstellen, die die Benutzeraufforderung für jedes Beispiel enthält.
def format_test_data(example: dict[str, any]) -> dict[str, any]:
example["messages"] = [
{
"role": "user",
"content": [
{
"type": "image",
},
{
"type": "text",
"text": PROMPT,
},
],
},
]
return example
test_data = data["validation"]
test_data = test_data.map(format_test_data)Um die Leistung des Modells zu bewerten, verwenden wir die Evaluierungsbibliothek, die vorgefertigte Metriken für Aufgaben wie die Klassifizierung bietet. Nach dem Importieren der Bibliothek und dem Laden der erforderlichen Metriken extrahieren wir aus dem Testdatensatz die wahren Bezeichnungen (ground-truth labels). Dann wird eine Hilfsfunktion compute_metrics definiert, um die Genauigkeit und den F1-Score zu berechnen, indem die Vorhersagen mit diesen Labels verglichen werden.
import evaluate
accuracy_metric = evaluate.load("accuracy")
f1_metric = evaluate.load("f1")
# Ground-truth labels
REFERENCES = test_data["label"]
def compute_metrics(predictions: list[int]) -> dict[str, float]:
metrics = {}
metrics.update(
accuracy_metric.compute(
predictions=predictions,
references=REFERENCES,
)
)
metrics.update(
f1_metric.compute(
predictions=predictions,
references=REFERENCES,
average="weighted",
)
)
return metricsUm eine einheitliche Handhabung der Labels zu gewährleisten, wird die Spalte "Label" des Datensatzes in einen ClassLabel Typ umgewandelt. Dies ermöglicht eine effiziente Zuordnung zwischen Label-Indizes und ihren entsprechenden Namen. Außerdem definieren wir alternative Label-Mappings, um Variationen in der Label-Formatierung während der Nachbearbeitung zu behandeln.
from datasets import ClassLabel
test_data = test_data.cast_column(
"label",
ClassLabel(names=BRAIN_CANCER_CLASSES)
)
LABEL_FEATURE = test_data.features["label"]
ALT_LABELS = dict([
(label, f"({label.replace(': ', ') ')}") for label in BRAIN_CANCER_CLASSES
])Um die Vorhersagen des Modells den richtigen Klassenbezeichnungen zuzuordnen, definieren wir eine postprocess Funktion. Diese Funktion stellt sicher, dass die Vorhersagen mit dem richtigen Label abgeglichen werden, wobei sowohl kanonische als auch alternative Labelformate berücksichtigt werden.
def postprocess(prediction, do_full_match: bool = False) -> int:
if isinstance(prediction, str):
response_text = prediction
else:
response_text = prediction[0]["generated_text"]
if do_full_match:
return LABEL_FEATURE.str2int(response_text)
for label in BRAIN_CANCER_CLASSES:
# accept canonical or alternative wording
if label in response_text or ALT_LABELS[label] in response_text:
return LABEL_FEATURE.str2int(label)
return -110. Modellleistung beim Basismodell
Um die Leistung des Basismodells zu bewerten, laden wir das vortrainierte Modell und den Prozessor, konfigurieren die Generierungseinstellungen und bereiten die Prompts und Bilder für den Test vor.
import torch
from transformers import AutoModelForImageTextToText, AutoProcessor
model_kwargs = dict(
torch_dtype=torch.bfloat16,
device_map="auto",
)
model = AutoModelForImageTextToText.from_pretrained(
model_id, **model_kwargs
)
from transformers import GenerationConfig
gen_cfg = GenerationConfig.from_pretrained(model_id)
gen_cfg.update(
do_sample = False,
top_k = None,
top_p = None,
cache_implementation = "dynamic"
)
model.generation_config = gen_cfg
processor = AutoProcessor.from_pretrained(args.output_dir)
tok = processor.tokenizer
model.config.pad_token_id = tok.pad_token_id
model.generation_config.pad_token_id = tok.pad_token_id
def chat_to_prompt(chat_turns):
return processor.apply_chat_template(
chat_turns,
add_generation_prompt=True, # tells the model "your turn"
tokenize=False # we want raw text, not ids
)
prompts = [chat_to_prompt(c) for c in test_data["messages"]]
images = test_data["image"] # already a list of PIL images
assert len(prompts) == len(images), "1 prompt must match 1 image!"Wir haben eine batch_predict Funktion erstellt, die den Testdatensatz in Stapeln verarbeitet. Diese Funktion erstellt Vorhersagen für jedes Prompt-Bild-Paar und wendet eine Nachbearbeitung an, um die Ausgaben den richtigen Bezeichnungen zuzuordnen.
import torch
from typing import List, Any, Callable
def batch_predict(
prompts,
images,
model,
processor,
postprocess,
*,
batch_size=64,
device="cuda",
dtype=torch.bfloat16,
**gen_kwargs
):
preds = []
for i in range(0, len(prompts), batch_size):
texts = prompts[i : i + batch_size]
imgs = [[img] for img in images[i : i + batch_size]]
enc = processor(text=texts, images=imgs, padding=True, return_tensors="pt").to(
device, dtype=dtype
)
lens = enc["attention_mask"].sum(dim=1)
with torch.inference_mode():
out = model.generate(
**enc,
disable_compile=True,
**gen_kwargs
)
for seq, ln in zip(out, lens):
ans = processor.decode(seq[ln:], skip_special_tokens=True)
preds.append(postprocess(ans))
return predsWir werden die Funktion batch_predict verwenden, um Vorhersagen für das Basismodell auf dem Testdatensatz zu erstellen. Die Vorhersagen werden dann mit der Funktion compute_metrics ausgewertet.
bf_preds = batch_predict(
model = model,
processor = processor,
prompts = prompts,
images = images,
batch_size = 64,
max_new_tokens= 40, # forwarded to generate
postprocess= postprocess, # your label-mapping function
)
bf_metrics = compute_metrics(bf_preds)
print(f"Baseline metrics: {bf_metrics}")Das Ergebnis ist nicht beeindruckend. Wir haben eine Genauigkeit von 33%, was ziemlich schlecht ist.
Baseline metrics: {'accuracy': 0.33745874587458746, 'f1': 0.1737287617650654}Um das Verhalten des Modells besser zu verstehen, können wir Vorhersagen für ein einzelnes Beispiel aus dem Datensatz erstellen. Dazu musst du eine Hilfsfunktion erstellen, die die Eingabe verarbeitet und die Antwort des Modells zurückgibt.
Die Funktion predict_one nimmt eine Eingabeaufforderung und ein Bild als Input, verarbeitet sie mit dem Prozessor des Modells und erzeugt eine Antwort. Die Funktion stellt sicher, dass die Ausgabe des Modells in einen für Menschen lesbaren Text umgewandelt wird.
import torch
from typing import Union, Dict, Any, List
from transformers import AutoModelForImageTextToText, AutoProcessor
def predict_one(
prompt,
image,
model,
processor,
*,
device="cuda",
dtype=torch.bfloat16,
disable_compile=True,
**gen_kwargs
) -> str:
inputs = processor(text=prompt, images=image, return_tensors="pt").to(
device, dtype=dtype
)
plen = inputs["input_ids"].shape[-1]
with torch.inference_mode():
ids = model.generate(
**inputs,
disable_compile=disable_compile,
**gen_kwargs
)
return processor.decode(ids[0, plen:], skip_special_tokens=True)Wir nutzen die predict_one, um eine Antwort für die 11. Stichprobe aus dem Datensatz zu erstellen. Dazu musst du die Eingabeaufforderung vorbereiten und die Vorhersagefunktion ausführen.
idx = 10
chat = test_data["messages"][idx]
prompt = processor.apply_chat_template(
chat,
add_generation_prompt=True,
tokenize=False
)
# run the one-sample helper
answer = predict_one(
prompt = prompt,
image = test_data["image"][idx],
model = model,
processor= processor,
max_new_tokens = 40
)
print("Model answer:", answer)Daraufhin erhielten wir einen langen Satz, in dem erklärt wurde, warum das Hirngliom ausgewählt wurde. Die Antwort ist völlig falsch, sogar die Klassifizierung ist falsch.
Model answer: Based on the MRI image, the most likely type of brain cancer is **A: brain glioma**.
Here's why:
* **Gliomas** are a common type of brain tumor11. Leistung des Modells beim feinabgestimmten Modell
Um das feinabgestimmte Modell zu bewerten, wiederholen wir den Bewertungsprozess, indem wir das Modell aus dem Ausgabeverzeichnis laden, Vorhersagen erstellen und die Metriken berechnen.
Wir laden das feinabgestimmte Modell und den Prozessor aus dem Ausgabeverzeichnis, führen die Funktion batch_predict aus und berechnen die Metriken .
model = AutoModelForImageTextToText.from_pretrained(
args.output_dir, **model_kwargs
)
model.generation_config = gen_cfg
processor = AutoProcessor.from_pretrained(args.output_dir)
tok = processor.tokenizer
model.config.pad_token_id = tok.pad_token_id
model.generation_config.pad_token_id = tok.pad_token_id
af_preds = batch_predict(
model = model,
processor = processor,
prompts = prompts,
images = images,
batch_size = 64,
max_new_tokens= 40, # forwarded to generate
postprocess= postprocess, # your label-mapping function
)
af_metrics = compute_metrics(af_preds)
print(f"Fine-tuned metrics: {af_metrics}")Diese Ergebnisse zeigen eine deutliche Verbesserung gegenüber dem Basismodell und unterstreichen die Wirksamkeit der Feinabstimmung. Die Genauigkeit sprang von 33% auf 89% mit nur einer Epoche.
Fine-tuned metrics: {'accuracy': 0.8927392739273927, 'f1': 0.892641793935792}Um die Leistung des feinabgestimmten Modells weiter zu analysieren, erstellen wir eine Vorhersage für ein einzelnes Beispiel aus dem Testdatensatz.
idx = 10
chat = test_data["messages"][idx]
prompt = processor.apply_chat_template(
chat,
add_generation_prompt=True,
tokenize=False
)
# run the one-sample helper
answer = predict_one(
prompt = prompt,
image = test_data["image"][idx],
model = model,
processor= processor,
max_new_tokens = 40 # any generate-kwargs you need
)
print("Model answer:", answer)Das Modell lieferte ein klares und präzises Ergebnis, wobei die Klassifizierung sowohl genau als auch gut strukturiert war.
Model answer: C: brain tumorWenn du Probleme beim Ausführen des obigen Codes hast, sieh dir bitte das Begleitheft an: Fine_tuning_MedGemma.ipynb
Fazit
MedGemma ist ein bedeutender Schritt vorwärts bei der Nutzung von KI für die medizinischen Wissenschaften. Indem sie Ärzte und Ärztinnen in die Lage versetzt, schnellere und genauere Urteile zu fällen, ermöglicht sie schnellere Diagnosen und effektivere Behandlungspläne für Patienten.
Die Feinabstimmung von Bildsprachmodellen wie MedGemma 4B Instruct ermöglicht die Anpassung an verschiedene medizinische Aufgaben, von der Bildklassifizierung bis hin zur Integration von Argumentationsfähigkeiten.
In diesem Tutorial haben wir gelernt, wie man ein Vision-Language-Modell auf einem Gehirn-MRT-Datensatz für die Klassifizierung von Hirntumoren fein abstimmt. Die Ergebnisse waren bemerkenswert: Die Genauigkeit des Modells verbesserte sich von 33 % auf 89 % - ein deutlicher Sprung, der das Potenzial der Feinabstimmung bei medizinischen KI-Anwendungen verdeutlicht.
Wenn du mehr darüber erfahren möchtest, empfehle ich dir, diese Ressourcen auszuprobieren:

Als zertifizierter Data Scientist ist es meine Leidenschaft, modernste Technologien zu nutzen, um innovative Machine Learning-Anwendungen zu entwickeln. Mit meinem fundierten Hintergrund in den Bereichen Spracherkennung, Datenanalyse und Reporting, MLOps, KI und NLP habe ich meine Fähigkeiten bei der Entwicklung intelligenter Systeme verfeinert, die wirklich etwas bewirken können. Neben meinem technischen Fachwissen bin ich auch ein geschickter Kommunikator mit dem Talent, komplexe Konzepte in eine klare und prägnante Sprache zu fassen. Das hat dazu geführt, dass ich ein gefragter Blogger zum Thema Datenwissenschaft geworden bin und meine Erkenntnisse und Erfahrungen mit einer wachsenden Gemeinschaft von Datenexperten teile. Zurzeit konzentriere ich mich auf die Erstellung und Bearbeitung von Inhalten und arbeite mit großen Sprachmodellen, um aussagekräftige und ansprechende Inhalte zu entwickeln, die sowohl Unternehmen als auch Privatpersonen helfen, das Beste aus ihren Daten zu machen.
