Curso
O MedGemma é uma coleção de variantes do Gemma 3 projetadas para você se destacar na compreensão de textos e imagens médicas. Atualmente, a coleção inclui duas variantes poderosas: uma versão multimodal de 4B e uma versão somente de texto de 27B.
O modelo MedGemma 4B combina o SigLIP pré-treinado em diversos conjuntos de dados médicos desidentificados, como radiografias de tórax, imagens de dermatologia, imagens de oftalmologia e lâminas de histopatologia, com um modelo de linguagem grande (LLM) treinado em uma ampla variedade de dados médicos.
Neste tutorial, aprenderemos como você pode fazer o ajuste fino o modelo MedGemma 4B em um conjunto de dados de ressonância magnética do cérebro para uma tarefa de classificação de imagens. O objetivo é adaptar o modelo menor MedGemma 4B para classificar com eficácia os exames de ressonância magnética do cérebro e prever o câncer cerebral com maior precisão e eficiência.
Configuração do ambiente
O RunPod é uma excelente plataforma para executar cargas de trabalho baseadas em GPU, oferecendo ambientes pré-configurados com suporte ao JupyterLab. Isso permite que você inicie um pod e comece a programar imediatamente usando o editor de sua preferência. Veja a seguir como você pode configurar seu ambiente:
1. Configurando o RunPod
Faça login no RunPod e crie um pod com uma GPU A100 e a imagem mais recente do PyTorch. Em seguida, clique no botão "Implantar sob demanda" para iniciar o pod.
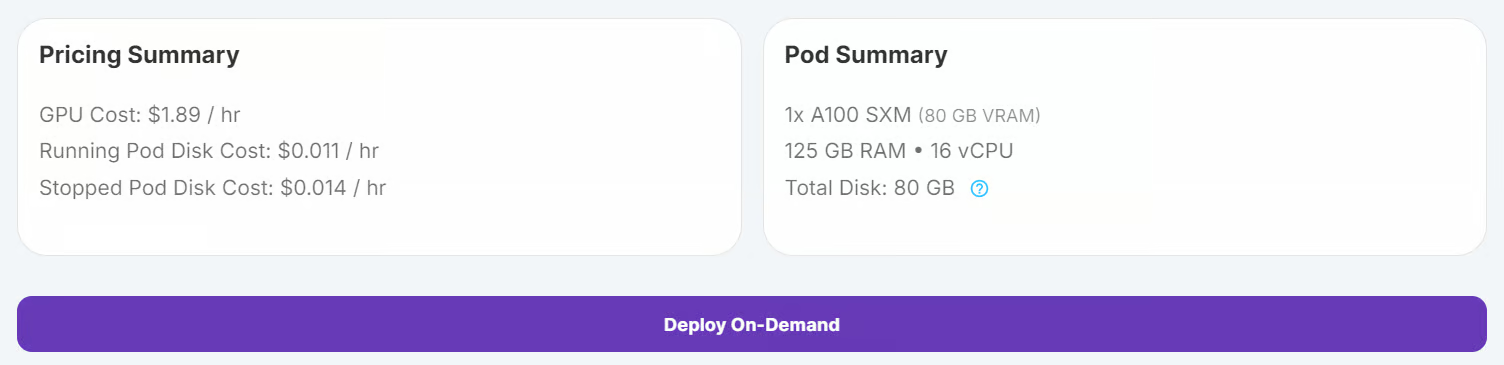
Edite o pod e adicione as seguintes variáveis de ambiente para a integração do Hugging Face e do Kaggle:
- HF_TOKEN
- KAGGLE_USERNAME
- KAGGLE_KEY
Aumente a capacidade de armazenamento para 100 GB para acomodar conjuntos de dados e pontos de verificação de modelos
2. Instalando os pacotes Python
Quando o pod estiver em execução, inicie a instância do JupyterLab clicando no botão "Connect" (Conectar). Crie um novo notebook Python e instale os pacotes Python necessários executando o seguinte comando:
! pip install --upgrade --quiet transformers bitsandbytes datasets evaluate peft trl scikit-learn kaggleCarregamento e processamento do conjunto de dados
Usaremos o Câncer cerebral do Kaggle.
3. Baixando o conjunto de dados
Faça o download e descompacte o conjunto de dados usando a CLI do Kaggle:
!kaggle datasets download -d orvile/brain-cancer-mri-dataset --unzipDataset URL: https://www.kaggle.com/datasets/orvile/brain-cancer-mri-dataset
License(s): CC-BY-SA-4.0
Downloading brain-cancer-mri-dataset.zip to /workspace
90%|████████████████████████████████████▉ | 130M/144M [00:00<00:00, 231MB/s]
100%|█████████████████████████████████████████| 144M/144M [00:01<00:00, 108MB/s]4. Carregando o conjunto de dados
Carregue o conjunto de dados como um conjunto de dados Hugging Face, divida-o em conjuntos de treinamento e validação e exiba sua estrutura:
from datasets import load_dataset
data_dir = "./Brain_Cancer raw MRI data/Brain_Cancer"
# Define proportions for train and validation splits
train_size = 0.8
validation_size = 0.2
data = load_dataset("imagefolder", data_dir=data_dir, split="train")
# Split the dataset into train and validation sets
data = data.train_test_split(
train_size=train_size,
test_size=validation_size,
shuffle=True,
seed=42,
)
# Rename the 'test' split to 'validation'
data["validation"] = data.pop("test")
# Display dataset details
print(data)Saída:
DatasetDict({
train: Dataset({
features: ['image', 'label'],
num_rows: 4844
})
validation: Dataset({
features: ['image', 'label'],
num_rows: 1212
})
})Verifique a primeira imagem e o rótulo correspondente do conjunto de treinamento:
data["train"][0]["image"]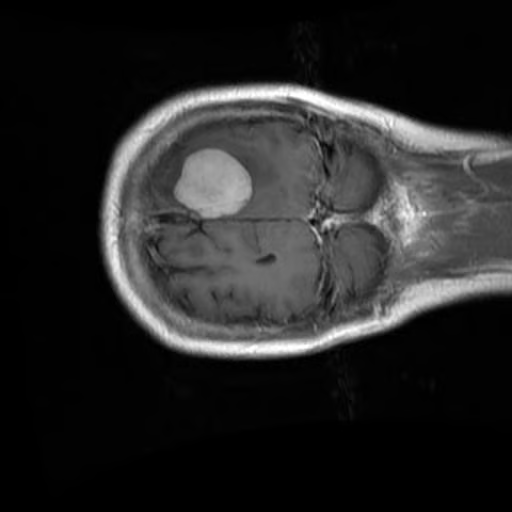
print(data["train"][0]["label"])15. Processamento do conjunto de dados
Antes de processar o conjunto de dados, é importante verificar primeiro os nomes dos rótulos para garantir o tratamento adequado da tarefa de classificação.
BRAIN_CANCER_CLASSES = data["train"].features["label"].names
print("Detected classes:", BRAIN_CANCER_CLASSES)As classes detectadas são: ['brain_glioma', 'brain_menin', 'brain_tumor'].
Detected classes: ['brain_glioma', 'brain_menin', 'brain_tumor']Para aprimorar o processo de classificação, modificaremos esses rótulos de classe adicionando um prefixo (A, B, C) para melhor organização e alinhamento com um formato de prompt personalizado.
BRAIN_CANCER_CLASSES = ['A: brain glioma', 'B: brain menin', 'C: brain tumor']Em seguida, criamos um prompt personalizado que será usado para orientar o modelo durante o ajuste fino. O prompt inclui os rótulos de classe atualizados.
options = "\n".join(BRAIN_CANCER_CLASSES)
PROMPT = f"What is the most likely type of brain cancer shown in the MRI image?\n{options}"Para preparar o conjunto de dados para o ajuste fino, criaremos uma nova coluna chamada "messages" (mensagens). Essa coluna conterá dados estruturados que representam uma consulta do usuário (o prompt) e a resposta do assistente (o rótulo correto).
def format_data(example: dict[str, any]) -> dict[str, any]:
example["messages"] = [
{
"role": "user",
"content": [
{
"type": "image",
},
{
"type": "text",
"text": PROMPT,
},
],
},
{
"role": "assistant",
"content": [
{
"type": "text",
"text": BRAIN_CANCER_CLASSES[example["label"]],
},
],
},
]
return example
# Apply the formatting to the dataset
formatted_data = data.map(format_data)Para verificar a formatação, podemos inspecionar a coluna de mensagens da primeira amostra:
formatted_data["train"][0]["messages"]O ponto de dados formatado resultante tem a seguinte aparência:
[{'content': [{'text': None, 'type': 'image'},
{'text': 'What is the most likely type of brain cancer shown in the MRI image?\nA: brain glioma\nB: brain menin\nC: brain tumor',
'type': 'text'}],
'role': 'user'},
{'content': [{'text': 'B: brain menin', 'type': 'text'}],
'role': 'assistant'}]Ajuste fino do MedGemma no conjunto de dados de ressonância magnética do cérebro
Nesta seção, faremos o ajuste fino do modelo MedGemma 4B Instruct no conjunto de dados de ressonância magnética do cérebro. Isso envolve o download do modelo e do processador, a instalação do adaptador LoRA, a configuração do instrutor e o início do processo de treinamento.
6. Carregando o modelo e o tokenizador
Como a MedGemma é um modelo fechado, você precisa fazer login na CLI da Hugging Face usando sua chave de API. Isso também permite que você salve seu modelo ajustado no Hugging Face Hub. Confira nosso curso, Trabalhando com Hugging Face: Your Guide to the Hub, se você precisar de uma atualização .
from huggingface_hub import login
import os
hf_token = os.environ.get("HF_TOKEN")
login(hf_token)Usamos a biblioteca Transformers para carregar o modelo MedGemma 4B Instruct e seu processador. O modelo está configurado para usar a precisão bfloat16 para computação eficiente em GPUs.
import torch
from transformers import AutoProcessor, AutoModelForImageTextToText, BitsAndBytesConfig
model_id = "google/medgemma-4b-it"
# Check if GPU supports bfloat16
if torch.cuda.get_device_capability()[0] < 8:
raise ValueError("GPU does not support bfloat16, please use a GPU that supports bfloat16.")
model_kwargs = dict(
attn_implementation="eager",
torch_dtype=torch.bfloat16,
device_map="auto",
)
model = AutoModelForImageTextToText.from_pretrained(model_id, **model_kwargs)
processor = AutoProcessor.from_pretrained(model_id)
# Use right padding to avoid issues during training
processor.tokenizer.padding_side = "right"7. Configuração do modelo
Para ajustar o modelo MedGemma 4B Instruct de forma eficiente, usaremos Low-Rank Adaptation (LoRA)um método de ajuste fino eficiente em termos de parâmetros.
O LoRA nos permite adaptar modelos grandes treinando apenas um pequeno número de parâmetros adicionais, reduzindo significativamente os custos de computação e mantendo o desempenho.
from peft import LoraConfig
peft_config = LoraConfig(
lora_alpha=16,
lora_dropout=0.05,
r=16,
bias="none",
target_modules="all-linear",
task_type="CAUSAL_LM",
modules_to_save=[
"lm_head",
"embed_tokens",
],
)Para lidar com as entradas de imagem e texto durante o treinamento, definimos uma função de agrupamento personalizada. Essa função processa os exemplos do conjunto de dados em um formato adequado para o modelo, incluindo tokenização de texto e preparação de dados de imagem.
def collate_fn(examples: list[dict[str, any]]):
texts = []
images = []
for example in examples:
images.append([example["image"]])
texts.append(
processor.apply_chat_template(
example["messages"], add_generation_prompt=False, tokenize=False
).strip()
)
# Tokenize the texts and process the images
batch = processor(text=texts, images=images, return_tensors="pt", padding=True)
# The labels are the input_ids, with the padding and image tokens masked in
# the loss computation
labels = batch["input_ids"].clone()
# Mask image tokens
image_token_id = [
processor.tokenizer.convert_tokens_to_ids(
processor.tokenizer.special_tokens_map["boi_token"]
)
]
# Mask tokens that are not used in the loss computation
labels[labels == processor.tokenizer.pad_token_id] = -100
labels[labels == image_token_id] = -100
labels[labels == 262144] = -100
batch["labels"] = labels
return batchUsamos a classe SFTConfig da bibliotecatrl para definir os argumentos de treinamento. Esses argumentos controlam o processo de ajuste fino, incluindo o tamanho do lote, a taxa de aprendizado e as etapas de acumulação de gradiente.
from trl import SFTConfig
args = SFTConfig(
output_dir="medgemma-brain-cancer",
num_train_epochs=1,
per_device_train_batch_size=8,
per_device_eval_batch_size=8,
gradient_accumulation_steps=8,
gradient_checkpointing=True,
optim="adamw_torch_fused",
logging_steps=0.1,
save_strategy="epoch",
eval_strategy="steps",
eval_steps=0.1,
learning_rate=2e-4,
bf16=True,
max_grad_norm=0.3,
warmup_ratio=0.03,
lr_scheduler_type="linear",
push_to_hub=True,
report_to="none",
gradient_checkpointing_kwargs={"use_reentrant": False},
dataset_kwargs={"skip_prepare_dataset": True},
remove_unused_columns = False,
label_names=["labels"],
)O SFTTrainer simplifica o processo de ajuste fino, combinando o modelo, o conjunto de dados, o coletor de dados, os argumentos de treinamento e a configuração do LoRA em um único fluxo de trabalho. Isso torna o processo simplificado e fácil de usar.
from trl import SFTTrainer
trainer = SFTTrainer(
model=model,
args=args,
train_dataset=formatted_data["train"],
eval_dataset=formatted_data["validation"].shuffle().select(range(50)),
peft_config=peft_config,
processing_class=processor,
data_collator=collate_fn,
)8. Treinamento de modelos
Depois que o modelo, o conjunto de dados e as configurações de treinamento estiverem definidos, podemos iniciar o processo de ajuste fino. O SFTTrainer simplifica essa etapa, permitindo que você treine o modelo com apenas um único comando:
trainer.train()O processo de treinamento levou aproximadamente 1 hora e 8 minutos para ser concluído. Durante esse tempo, a perda de treinamento e a perda de validação diminuíram constantemente a cada etapa, indicando que o modelo estava aprendendo de forma eficaz.
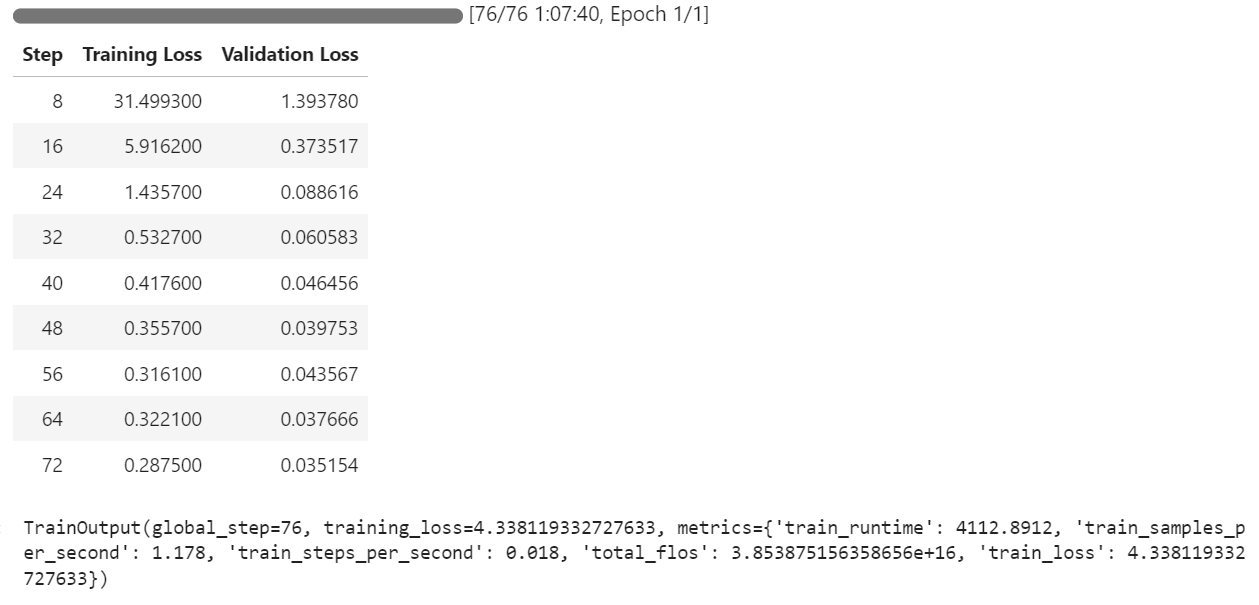
Após a conclusão do treinamento, o modelo ajustado pode ser salvo localmente e enviado para o Hugging Face Hub usando o método save_model().
trainer.save_model()O modelo já está disponível kingabzpro/medgemma-brain-cancer - Hugging Face
Source kingabzpro/medgemma-brain-cancer
Avaliação do modelo
Para avaliar o desempenho do modelo MedGemma 4B, testaremos o modelo básico e o modelo ajustado no conjunto de dados de validação. Esse processo envolve limpar a memória, preparar os dados de teste, gerar a resposta e calcular as principais métricas, como precisão e pontuação F1.
Antes de iniciar a avaliação, removemos a configuração de treinamento para liberar a memória da GPU e garantir um ambiente limpo para os testes
del model
del trainer
torch.cuda.empty_cache()9. Preparação para testes de modelos
Formatamos o conjunto de dados de validação para corresponder à estrutura de entrada exigida pelo modelo. Isso envolve a criação de uma coluna "messages" que contém o prompt do usuário para cada exemplo.
def format_test_data(example: dict[str, any]) -> dict[str, any]:
example["messages"] = [
{
"role": "user",
"content": [
{
"type": "image",
},
{
"type": "text",
"text": PROMPT,
},
],
},
]
return example
test_data = data["validation"]
test_data = test_data.map(format_test_data)Para avaliar o desempenho do modelo, usamos a biblioteca evaluate, que fornece métricas pré-criadas para tarefas como classificação. Depois de importar a biblioteca e carregar as métricas necessárias, extraímos rótulos de verdade do conjunto de dados de teste. Uma função auxiliar, compute_metrics, é então definida para calcular a precisão e a pontuação F1, comparando as previsões com esses rótulos.
import evaluate
accuracy_metric = evaluate.load("accuracy")
f1_metric = evaluate.load("f1")
# Ground-truth labels
REFERENCES = test_data["label"]
def compute_metrics(predictions: list[int]) -> dict[str, float]:
metrics = {}
metrics.update(
accuracy_metric.compute(
predictions=predictions,
references=REFERENCES,
)
)
metrics.update(
f1_metric.compute(
predictions=predictions,
references=REFERENCES,
average="weighted",
)
)
return metricsPara garantir a consistência no tratamento de rótulos, convertemos a coluna "label" do conjunto de dados em um tipo ClassLabel. Isso permite um mapeamento eficiente entre os índices de rótulos e seus nomes correspondentes. Além disso, definimos mapeamentos alternativos de rótulos para lidar com variações na formatação de rótulos durante o pós-processamento.
from datasets import ClassLabel
test_data = test_data.cast_column(
"label",
ClassLabel(names=BRAIN_CANCER_CLASSES)
)
LABEL_FEATURE = test_data.features["label"]
ALT_LABELS = dict([
(label, f"({label.replace(': ', ') ')}") for label in BRAIN_CANCER_CLASSES
])Para mapear as previsões do modelo para os rótulos de classe corretos, definimos uma função postprocess. Essa função garante que as previsões sejam combinadas com o rótulo apropriado, levando em conta os formatos de rótulo canônico e alternativo.
def postprocess(prediction, do_full_match: bool = False) -> int:
if isinstance(prediction, str):
response_text = prediction
else:
response_text = prediction[0]["generated_text"]
if do_full_match:
return LABEL_FEATURE.str2int(response_text)
for label in BRAIN_CANCER_CLASSES:
# accept canonical or alternative wording
if label in response_text or ALT_LABELS[label] in response_text:
return LABEL_FEATURE.str2int(label)
return -110. Desempenho do modelo no modelo básico
Para avaliar o desempenho do modelo básico, carregamos o modelo e o processador pré-treinados, definimos as configurações de geração e preparamos os prompts e as imagens para teste.
import torch
from transformers import AutoModelForImageTextToText, AutoProcessor
model_kwargs = dict(
torch_dtype=torch.bfloat16,
device_map="auto",
)
model = AutoModelForImageTextToText.from_pretrained(
model_id, **model_kwargs
)
from transformers import GenerationConfig
gen_cfg = GenerationConfig.from_pretrained(model_id)
gen_cfg.update(
do_sample = False,
top_k = None,
top_p = None,
cache_implementation = "dynamic"
)
model.generation_config = gen_cfg
processor = AutoProcessor.from_pretrained(args.output_dir)
tok = processor.tokenizer
model.config.pad_token_id = tok.pad_token_id
model.generation_config.pad_token_id = tok.pad_token_id
def chat_to_prompt(chat_turns):
return processor.apply_chat_template(
chat_turns,
add_generation_prompt=True, # tells the model "your turn"
tokenize=False # we want raw text, not ids
)
prompts = [chat_to_prompt(c) for c in test_data["messages"]]
images = test_data["image"] # already a list of PIL images
assert len(prompts) == len(images), "1 prompt must match 1 image!"Criamos uma função batch_predict que processa o conjunto de dados de teste em lotes. Essa função gera previsões para cada par de imagem-pedido e aplica o pós-processamento para mapear os resultados para os rótulos corretos.
import torch
from typing import List, Any, Callable
def batch_predict(
prompts,
images,
model,
processor,
postprocess,
*,
batch_size=64,
device="cuda",
dtype=torch.bfloat16,
**gen_kwargs
):
preds = []
for i in range(0, len(prompts), batch_size):
texts = prompts[i : i + batch_size]
imgs = [[img] for img in images[i : i + batch_size]]
enc = processor(text=texts, images=imgs, padding=True, return_tensors="pt").to(
device, dtype=dtype
)
lens = enc["attention_mask"].sum(dim=1)
with torch.inference_mode():
out = model.generate(
**enc,
disable_compile=True,
**gen_kwargs
)
for seq, ln in zip(out, lens):
ans = processor.decode(seq[ln:], skip_special_tokens=True)
preds.append(postprocess(ans))
return predsUsaremos a função batch_predict para gerar previsões para o modelo básico no conjunto de dados de teste. Em seguida, as previsões são avaliadas usando a função compute_metrics.
bf_preds = batch_predict(
model = model,
processor = processor,
prompts = prompts,
images = images,
batch_size = 64,
max_new_tokens= 40, # forwarded to generate
postprocess= postprocess, # your label-mapping function
)
bf_metrics = compute_metrics(bf_preds)
print(f"Baseline metrics: {bf_metrics}")O resultado não é impressionante. Obtivemos 33% de precisão, o que é muito ruim.
Baseline metrics: {'accuracy': 0.33745874587458746, 'f1': 0.1737287617650654}Para entender melhor o comportamento do modelo, podemos gerar previsões para um único exemplo do conjunto de dados. Isso envolve a criação de uma função auxiliar para processar a entrada e retornar a resposta do modelo.
A função predict_one recebe um prompt e uma imagem como entrada, processa-os usando o processador do modelo e gera uma resposta. A função garante que a saída do modelo seja decodificada em um texto legível por humanos.
import torch
from typing import Union, Dict, Any, List
from transformers import AutoModelForImageTextToText, AutoProcessor
def predict_one(
prompt,
image,
model,
processor,
*,
device="cuda",
dtype=torch.bfloat16,
disable_compile=True,
**gen_kwargs
) -> str:
inputs = processor(text=prompt, images=image, return_tensors="pt").to(
device, dtype=dtype
)
plen = inputs["input_ids"].shape[-1]
with torch.inference_mode():
ids = model.generate(
**inputs,
disable_compile=disable_compile,
**gen_kwargs
)
return processor.decode(ids[0, plen:], skip_special_tokens=True)Usaremos o site predict_one para gerar uma resposta para a 11ª amostra do conjunto de dados. Isso envolve a preparação do prompt e a execução da função de previsão.
idx = 10
chat = test_data["messages"][idx]
prompt = processor.apply_chat_template(
chat,
add_generation_prompt=True,
tokenize=False
)
# run the one-sample helper
answer = predict_one(
prompt = prompt,
image = test_data["image"][idx],
model = model,
processor= processor,
max_new_tokens = 40
)
print("Model answer:", answer)Como resultado, recebemos uma longa sentença explicando por que o glioma cerebral foi escolhido. A resposta está completamente errada, até mesmo a classificação está errada.
Model answer: Based on the MRI image, the most likely type of brain cancer is **A: brain glioma**.
Here's why:
* **Gliomas** are a common type of brain tumor11. Desempenho do modelo no modelo com ajuste fino
Para avaliar o modelo com ajuste fino, repetimos o processo de avaliação carregando o modelo do diretório de saída, gerando previsões e calculando métricas.
Carregamos o modelo ajustado e o processador do diretório de saída, executamos a função batch_predict e calculamos as métricas .
model = AutoModelForImageTextToText.from_pretrained(
args.output_dir, **model_kwargs
)
model.generation_config = gen_cfg
processor = AutoProcessor.from_pretrained(args.output_dir)
tok = processor.tokenizer
model.config.pad_token_id = tok.pad_token_id
model.generation_config.pad_token_id = tok.pad_token_id
af_preds = batch_predict(
model = model,
processor = processor,
prompts = prompts,
images = images,
batch_size = 64,
max_new_tokens= 40, # forwarded to generate
postprocess= postprocess, # your label-mapping function
)
af_metrics = compute_metrics(af_preds)
print(f"Fine-tuned metrics: {af_metrics}")Esses resultados mostram uma melhoria significativa em relação ao modelo básico, destacando a eficácia do ajuste fino. A precisão saltou de 33% para 89% com apenas uma época.
Fine-tuned metrics: {'accuracy': 0.8927392739273927, 'f1': 0.892641793935792}Para analisar melhor o desempenho do modelo ajustado, geramos uma previsão para um único exemplo do conjunto de dados de teste.
idx = 10
chat = test_data["messages"][idx]
prompt = processor.apply_chat_template(
chat,
add_generation_prompt=True,
tokenize=False
)
# run the one-sample helper
answer = predict_one(
prompt = prompt,
image = test_data["image"][idx],
model = model,
processor= processor,
max_new_tokens = 40 # any generate-kwargs you need
)
print("Model answer:", answer)O modelo produziu o resultado de forma clara e precisa, com a classificação sendo exata e bem estruturada.
Model answer: C: brain tumorSe você estiver enfrentando problemas ao executar o código acima, consulte o notebook complementar: Fine_tuning_MedGemma.ipynb
Conclusão
O MedGemma representa um avanço significativo no uso da IA para ciências médicas. Ao capacitar médicos e profissionais de saúde a fazer julgamentos mais rápidos e precisos, ele permite diagnósticos mais rápidos e planos de tratamento mais eficazes para os pacientes.
O ajuste fino dos modelos de linguagem de visão, como o MedGemma 4B Instruct, permite a adaptabilidade em várias tarefas médicas, desde a classificação de imagens até a integração de recursos de raciocínio.
Neste tutorial, aprendemos a fazer o ajuste fino de um modelo de visão-linguagem em um conjunto de dados de ressonância magnética do cérebro para classificação de câncer cerebral. Os resultados foram notáveis, com a precisão do modelo melhorando de 33% para 89%, um salto substancial que destaca o potencial do ajuste fino em aplicações de IA médica.
Se você estiver interessado em saber mais, recomendo que consulte estes recursos:

Sou um cientista de dados certificado que gosta de criar aplicativos de aprendizado de máquina e escrever blogs sobre ciência de dados. No momento, estou me concentrando na criação e edição de conteúdo e no trabalho com modelos de linguagem de grande porte.


