Cours
MedGemma est une collection de variantes de Gemma 3 conçues pour exceller dans la compréhension de textes et d'images médicales. La collection comprend actuellement deux variantes puissantes : une version multimodale 4B et une version textuelle 27B.
Le modèle MedGemma 4B combine la technologie SigLIP pré-entraîné sur divers ensembles de données médicales dépersonnalisées telles que des radiographies thoraciques, des images dermatologiques, des images ophtalmologiques et des lames histopathologiques, avec un modèle de langage étendu (LLM) entraîné sur un large éventail de données médicales.
Dans ce tutoriel, nous apprendrons à peaufiner le modèle MedGemma 4B sur un jeu de données d'IRM cérébrale pour une tâche de classification d'images. L'objectif est d'adapter le modèle MedGemma 4B, plus petit, pour classifier efficacement les IRM cérébrales et prédire le cancer du cerveau avec une précision et une efficacité accrues.
Mise en place de l'environnement
RunPod est une excellente plateforme pour l'exécution de charges de travail basées sur le GPU, offrant des environnements préconfigurés avec le support de JupyterLab. Cela vous permet de lancer un pod et de commencer à coder immédiatement en utilisant l'éditeur de votre choix. Voici comment configurer votre environnement :
1. Mise en place du RunPod
Connectez-vous à RunPod et créez un pod avec un GPU A100 et la dernière image PyTorch. Cliquez ensuite sur le bouton "Deploy On-Demand" pour lancer le pod.
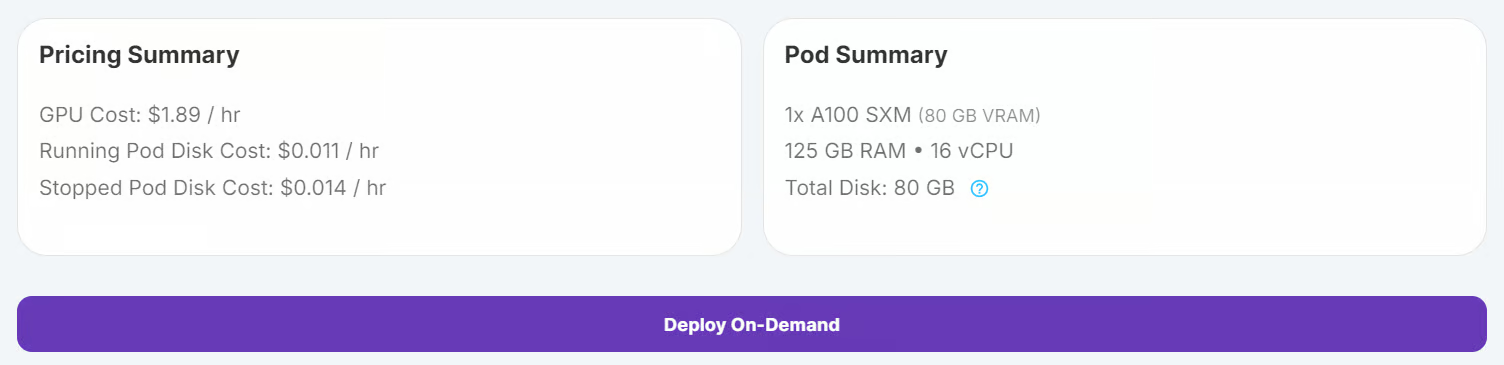
Modifiez le pod et ajoutez les variables d'environnement suivantes pour l'intégration de Hugging Face et de Kaggle :
- HF_TOKEN
- KAGGLE_USERNAME
- KAGGLE_KEY
Augmenter la capacité de stockage à 100 Go pour accueillir les ensembles de données et les points de contrôle des modèles.
2. Installation des paquets Python
Une fois que le pod fonctionne, lancez l'instance JupyterLab en cliquant sur le bouton "Connect". Créez un nouveau notebook Python et installez les paquets Python requis en exécutant la commande suivante :
! pip install --upgrade --quiet transformers bitsandbytes datasets evaluate peft trl scikit-learn kaggleChargement et traitement de l'ensemble de données
Nous utiliserons l'outil Cancer du cerveau MRI de Kaggle.
3. Télécharger l'ensemble de données
Téléchargez et décompressez le jeu de données à l'aide de l'interface de commande Kaggle :
!kaggle datasets download -d orvile/brain-cancer-mri-dataset --unzipDataset URL: https://www.kaggle.com/datasets/orvile/brain-cancer-mri-dataset
License(s): CC-BY-SA-4.0
Downloading brain-cancer-mri-dataset.zip to /workspace
90%|████████████████████████████████████▉ | 130M/144M [00:00<00:00, 231MB/s]
100%|█████████████████████████████████████████| 144M/144M [00:01<00:00, 108MB/s]4. Chargement du jeu de données
Chargez l'ensemble de données en tant qu'ensemble de données Hugging Face, divisez-le en ensembles d'entraînement et de validation, et affichez sa structure :
from datasets import load_dataset
data_dir = "./Brain_Cancer raw MRI data/Brain_Cancer"
# Define proportions for train and validation splits
train_size = 0.8
validation_size = 0.2
data = load_dataset("imagefolder", data_dir=data_dir, split="train")
# Split the dataset into train and validation sets
data = data.train_test_split(
train_size=train_size,
test_size=validation_size,
shuffle=True,
seed=42,
)
# Rename the 'test' split to 'validation'
data["validation"] = data.pop("test")
# Display dataset details
print(data)Sortie :
DatasetDict({
train: Dataset({
features: ['image', 'label'],
num_rows: 4844
})
validation: Dataset({
features: ['image', 'label'],
num_rows: 1212
})
})Vérifiez la première image et son étiquette correspondante dans l'ensemble d'apprentissage :
data["train"][0]["image"]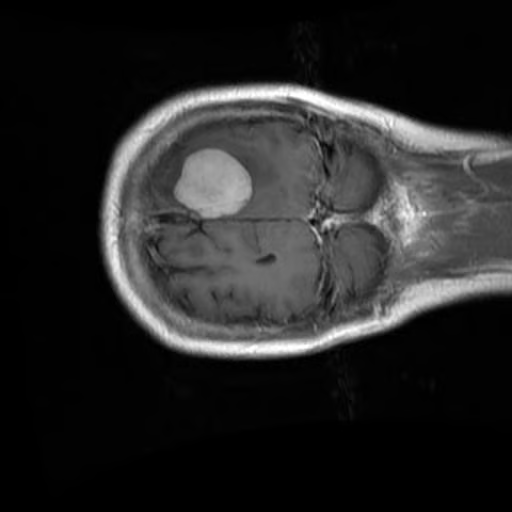
print(data["train"][0]["label"])15. Traitement de l'ensemble des données
Avant de traiter l'ensemble de données, il est important de vérifier les noms des étiquettes afin de garantir un traitement correct de la tâche de classification.
BRAIN_CANCER_CLASSES = data["train"].features["label"].names
print("Detected classes:", BRAIN_CANCER_CLASSES)Les classes détectées sont les suivantes : ['brain_glioma', 'brain_menin', 'brain_tumor'].
Detected classes: ['brain_glioma', 'brain_menin', 'brain_tumor']Pour améliorer le processus de classification, nous modifierons ces étiquettes de classe en ajoutant un préfixe (A, B, C) pour une meilleure organisation et pour s'aligner sur un format d'invite personnalisé.
BRAIN_CANCER_CLASSES = ['A: brain glioma', 'B: brain menin', 'C: brain tumor']Ensuite, nous créons une invite personnalisée qui sera utilisée pour guider le modèle lors de la mise au point. L'invite comprend les étiquettes de classe mises à jour.
options = "\n".join(BRAIN_CANCER_CLASSES)
PROMPT = f"What is the most likely type of brain cancer shown in the MRI image?\n{options}"Pour préparer l'ensemble de données à un réglage fin, nous allons créer une nouvelle colonne appelée "messages". Cette colonne contient des données structurées représentant une requête de l'utilisateur (l'invite) et la réponse de l'assistant (l'étiquette correcte).
def format_data(example: dict[str, any]) -> dict[str, any]:
example["messages"] = [
{
"role": "user",
"content": [
{
"type": "image",
},
{
"type": "text",
"text": PROMPT,
},
],
},
{
"role": "assistant",
"content": [
{
"type": "text",
"text": BRAIN_CANCER_CLASSES[example["label"]],
},
],
},
]
return example
# Apply the formatting to the dataset
formatted_data = data.map(format_data)Pour vérifier le formatage, nous pouvons inspecter la colonne des messages du premier échantillon :
formatted_data["train"][0]["messages"]Le point de données formaté qui en résulte ressemble à ceci :
[{'content': [{'text': None, 'type': 'image'},
{'text': 'What is the most likely type of brain cancer shown in the MRI image?\nA: brain glioma\nB: brain menin\nC: brain tumor',
'type': 'text'}],
'role': 'user'},
{'content': [{'text': 'B: brain menin', 'type': 'text'}],
'role': 'assistant'}]Mise au point de MedGemma sur l'ensemble de données d'IRM cérébrale
Dans cette section, nous allons affiner le modèle MedGemma 4B Instruct sur l'ensemble de données d'IRM cérébrale. Cela implique le téléchargement du modèle et du processeur, la mise en place de l'adaptateur LoRA, la configuration de l'unité didactique et le démarrage du processus d'apprentissage.
6. Chargement du modèle et du tokenizer
MedGemma étant un modèle à accès limité, vous devez vous connecter à l'interface de gestion de Hugging Face à l'aide de votre clé API. Cela vous permet également d'enregistrer votre modèle peaufiné dans le Hub Hugging Face. Consultez notre cours, Travailler avec Hugging Face : Your Guide to the Hub, si vous avez besoin d'une mise à jour .
from huggingface_hub import login
import os
hf_token = os.environ.get("HF_TOKEN")
login(hf_token)Nous utilisons la bibliothèque Transformers pour charger le modèle MedGemma 4B Instruct et son processeur. Le modèle est configuré pour utiliser la précision bfloat16 pour un calcul efficace sur les GPU.
import torch
from transformers import AutoProcessor, AutoModelForImageTextToText, BitsAndBytesConfig
model_id = "google/medgemma-4b-it"
# Check if GPU supports bfloat16
if torch.cuda.get_device_capability()[0] < 8:
raise ValueError("GPU does not support bfloat16, please use a GPU that supports bfloat16.")
model_kwargs = dict(
attn_implementation="eager",
torch_dtype=torch.bfloat16,
device_map="auto",
)
model = AutoModelForImageTextToText.from_pretrained(model_id, **model_kwargs)
processor = AutoProcessor.from_pretrained(model_id)
# Use right padding to avoid issues during training
processor.tokenizer.padding_side = "right"7. Mise en place du modèle
Pour affiner efficacement le modèle MedGemma 4B Instruct, nous utiliserons les méthodes suivantes l'adaptation de faible rang (LoRA)une méthode de réglage fin efficace en termes de paramètres.
LoRA nous permet d'adapter de grands modèles en n'entraînant qu'un petit nombre de paramètres supplémentaires, ce qui réduit considérablement les coûts de calcul tout en maintenant les performances.
from peft import LoraConfig
peft_config = LoraConfig(
lora_alpha=16,
lora_dropout=0.05,
r=16,
bias="none",
target_modules="all-linear",
task_type="CAUSAL_LM",
modules_to_save=[
"lm_head",
"embed_tokens",
],
)Pour gérer les entrées d'images et de texte pendant la formation, nous définissons une fonction de collationnement personnalisée. Cette fonction traite les exemples d'ensembles de données dans un format adapté au modèle, y compris la symbolisation du texte et la préparation des données d'image.
def collate_fn(examples: list[dict[str, any]]):
texts = []
images = []
for example in examples:
images.append([example["image"]])
texts.append(
processor.apply_chat_template(
example["messages"], add_generation_prompt=False, tokenize=False
).strip()
)
# Tokenize the texts and process the images
batch = processor(text=texts, images=images, return_tensors="pt", padding=True)
# The labels are the input_ids, with the padding and image tokens masked in
# the loss computation
labels = batch["input_ids"].clone()
# Mask image tokens
image_token_id = [
processor.tokenizer.convert_tokens_to_ids(
processor.tokenizer.special_tokens_map["boi_token"]
)
]
# Mask tokens that are not used in the loss computation
labels[labels == processor.tokenizer.pad_token_id] = -100
labels[labels == image_token_id] = -100
labels[labels == 262144] = -100
batch["labels"] = labels
return batchNous utilisons la classe SFTConfig de la bibliothèquetrl pour définir les arguments d'apprentissage. Ces arguments contrôlent le processus de réglage fin, y compris la taille du lot, le taux d'apprentissage et les étapes d'accumulation du gradient.
from trl import SFTConfig
args = SFTConfig(
output_dir="medgemma-brain-cancer",
num_train_epochs=1,
per_device_train_batch_size=8,
per_device_eval_batch_size=8,
gradient_accumulation_steps=8,
gradient_checkpointing=True,
optim="adamw_torch_fused",
logging_steps=0.1,
save_strategy="epoch",
eval_strategy="steps",
eval_steps=0.1,
learning_rate=2e-4,
bf16=True,
max_grad_norm=0.3,
warmup_ratio=0.03,
lr_scheduler_type="linear",
push_to_hub=True,
report_to="none",
gradient_checkpointing_kwargs={"use_reentrant": False},
dataset_kwargs={"skip_prepare_dataset": True},
remove_unused_columns = False,
label_names=["labels"],
)Le SFTTrainer simplifie le processus de réglage fin en combinant le modèle, le jeu de données, le collecteur de données, les arguments d'entraînement et la configuration LoRA en un seul flux de travail. Le processus est ainsi rationalisé et convivial.
from trl import SFTTrainer
trainer = SFTTrainer(
model=model,
args=args,
train_dataset=formatted_data["train"],
eval_dataset=formatted_data["validation"].shuffle().select(range(50)),
peft_config=peft_config,
processing_class=processor,
data_collator=collate_fn,
)8. Modèle de formation
Une fois que le modèle, l'ensemble de données et les configurations d'entraînement sont mis en place, nous pouvons commencer le processus d'ajustement. Le SFTTrainer simplifie cette étape, en nous permettant d'entraîner le modèle avec une seule commande :
trainer.train()Le processus de formation a duré environ 1 heure et 8 minutes. Pendant cette période, la perte d'apprentissage et la perte de validation ont diminué régulièrement à chaque étape, ce qui indique que le modèle apprend efficacement.
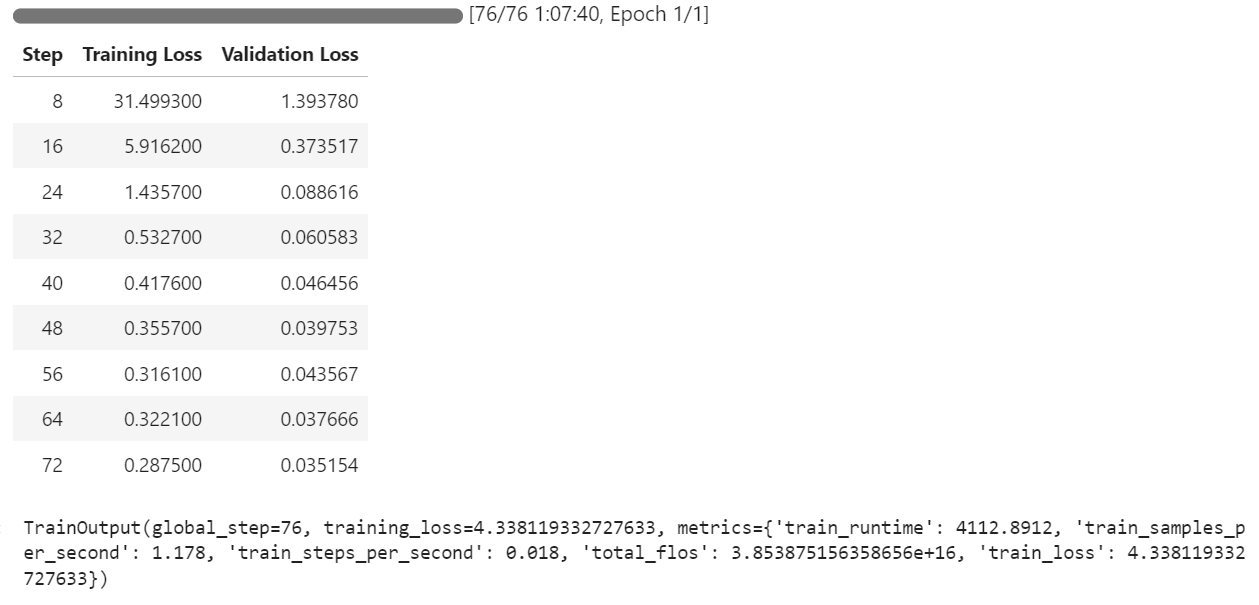
Une fois la formation terminée, le modèle affiné peut être enregistré localement et poussé vers le Hugging Face Hub à l'aide de la méthode save_model() .
trainer.save_model()Le modèle est maintenant disponible kingabzpro/medgemma-brain-cancer - Hugging Face
Source : Kingabzpro/Medgemma kingabzpro/medgemma-cancer-du-cerveau
Évaluation du modèle
Pour évaluer les performances du modèle MedGemma 4B, nous testerons à la fois le modèle de base et le modèle affiné sur l'ensemble de données de validation. Ce processus consiste à vider la mémoire, à préparer les données du test, à générer la réponse et à calculer des paramètres clés tels que la précision et le score F1.
Avant de commencer l'évaluation, nous supprimons la configuration d'entraînement afin de libérer la mémoire du GPU et de garantir un environnement propre pour les tests
del model
del trainer
torch.cuda.empty_cache()9. Mise en place de l'essai du modèle
Nous formatons l'ensemble de données de validation pour qu'il corresponde à la structure d'entrée requise par le modèle. Il s'agit de créer une colonne "messages" qui contient l'invite de l'utilisateur pour chaque exemple.
def format_test_data(example: dict[str, any]) -> dict[str, any]:
example["messages"] = [
{
"role": "user",
"content": [
{
"type": "image",
},
{
"type": "text",
"text": PROMPT,
},
],
},
]
return example
test_data = data["validation"]
test_data = test_data.map(format_test_data)Pour évaluer les performances du modèle, nous utilisons la bibliothèque evaluate, qui fournit des mesures prédéfinies pour des tâches telles que la classification. Après avoir importé la bibliothèque et chargé les métriques nécessaires, nous extrayons les étiquettes de vérité de base de l'ensemble de données de test. Une fonction d'aide, compute_metrics, est ensuite définie pour calculer la précision et le score F1 en comparant les prédictions à ces étiquettes.
import evaluate
accuracy_metric = evaluate.load("accuracy")
f1_metric = evaluate.load("f1")
# Ground-truth labels
REFERENCES = test_data["label"]
def compute_metrics(predictions: list[int]) -> dict[str, float]:
metrics = {}
metrics.update(
accuracy_metric.compute(
predictions=predictions,
references=REFERENCES,
)
)
metrics.update(
f1_metric.compute(
predictions=predictions,
references=REFERENCES,
average="weighted",
)
)
return metricsPour assurer la cohérence du traitement des étiquettes, nous transformons la colonne "étiquette" de l'ensemble de données en un type ClassLabel. Cela permet d'établir une correspondance efficace entre les indices des étiquettes et les noms correspondants. En outre, nous définissons des mappages d'étiquettes alternatifs pour gérer les variations de formatage des étiquettes au cours du post-traitement.
from datasets import ClassLabel
test_data = test_data.cast_column(
"label",
ClassLabel(names=BRAIN_CANCER_CLASSES)
)
LABEL_FEATURE = test_data.features["label"]
ALT_LABELS = dict([
(label, f"({label.replace(': ', ') ')}") for label in BRAIN_CANCER_CLASSES
])Pour faire correspondre les prédictions du modèle aux étiquettes de classe correctes, nous définissons une fonction postprocess. Cette fonction garantit que les prédictions sont associées à l'étiquette appropriée, en tenant compte des formats d'étiquettes canoniques et alternatifs.
def postprocess(prediction, do_full_match: bool = False) -> int:
if isinstance(prediction, str):
response_text = prediction
else:
response_text = prediction[0]["generated_text"]
if do_full_match:
return LABEL_FEATURE.str2int(response_text)
for label in BRAIN_CANCER_CLASSES:
# accept canonical or alternative wording
if label in response_text or ALT_LABELS[label] in response_text:
return LABEL_FEATURE.str2int(label)
return -110. Performances du modèle de base
Pour évaluer les performances du modèle de base, nous chargeons le modèle et le processeur pré-entraînés, nous configurons les paramètres de génération et nous préparons les messages-guides et les images pour le test.
import torch
from transformers import AutoModelForImageTextToText, AutoProcessor
model_kwargs = dict(
torch_dtype=torch.bfloat16,
device_map="auto",
)
model = AutoModelForImageTextToText.from_pretrained(
model_id, **model_kwargs
)
from transformers import GenerationConfig
gen_cfg = GenerationConfig.from_pretrained(model_id)
gen_cfg.update(
do_sample = False,
top_k = None,
top_p = None,
cache_implementation = "dynamic"
)
model.generation_config = gen_cfg
processor = AutoProcessor.from_pretrained(args.output_dir)
tok = processor.tokenizer
model.config.pad_token_id = tok.pad_token_id
model.generation_config.pad_token_id = tok.pad_token_id
def chat_to_prompt(chat_turns):
return processor.apply_chat_template(
chat_turns,
add_generation_prompt=True, # tells the model "your turn"
tokenize=False # we want raw text, not ids
)
prompts = [chat_to_prompt(c) for c in test_data["messages"]]
images = test_data["image"] # already a list of PIL images
assert len(prompts) == len(images), "1 prompt must match 1 image!"Nous avons créé une fonction batch_predict qui traite l'ensemble de données de test par lots. Cette fonction génère des prédictions pour chaque paire invite-image et applique un post-traitement pour associer les sorties aux étiquettes correctes.
import torch
from typing import List, Any, Callable
def batch_predict(
prompts,
images,
model,
processor,
postprocess,
*,
batch_size=64,
device="cuda",
dtype=torch.bfloat16,
**gen_kwargs
):
preds = []
for i in range(0, len(prompts), batch_size):
texts = prompts[i : i + batch_size]
imgs = [[img] for img in images[i : i + batch_size]]
enc = processor(text=texts, images=imgs, padding=True, return_tensors="pt").to(
device, dtype=dtype
)
lens = enc["attention_mask"].sum(dim=1)
with torch.inference_mode():
out = model.generate(
**enc,
disable_compile=True,
**gen_kwargs
)
for seq, ln in zip(out, lens):
ans = processor.decode(seq[ln:], skip_special_tokens=True)
preds.append(postprocess(ans))
return predsNous utiliserons la fonction batch_predict pour générer des prédictions pour le modèle de base sur l'ensemble de données de test. Les prédictions sont ensuite évaluées à l'aide de la fonction compute_metrics.
bf_preds = batch_predict(
model = model,
processor = processor,
prompts = prompts,
images = images,
batch_size = 64,
max_new_tokens= 40, # forwarded to generate
postprocess= postprocess, # your label-mapping function
)
bf_metrics = compute_metrics(bf_preds)
print(f"Baseline metrics: {bf_metrics}")Le résultat n'est pas impressionnant. Nous avons obtenu une précision de 33 %, ce qui est assez mauvais.
Baseline metrics: {'accuracy': 0.33745874587458746, 'f1': 0.1737287617650654}Pour mieux comprendre le comportement du modèle, nous pouvons générer des prédictions pour un seul exemple de l'ensemble de données. Cela implique la création d'une fonction d'aide pour traiter l'entrée et renvoyer la réponse du modèle.
La fonction predict_one prend en entrée une invite et une image, les traite à l'aide du processeur du modèle et génère une réponse. Cette fonction garantit que les résultats du modèle sont décodés en texte lisible par l'homme.
import torch
from typing import Union, Dict, Any, List
from transformers import AutoModelForImageTextToText, AutoProcessor
def predict_one(
prompt,
image,
model,
processor,
*,
device="cuda",
dtype=torch.bfloat16,
disable_compile=True,
**gen_kwargs
) -> str:
inputs = processor(text=prompt, images=image, return_tensors="pt").to(
device, dtype=dtype
)
plen = inputs["input_ids"].shape[-1]
with torch.inference_mode():
ids = model.generate(
**inputs,
disable_compile=disable_compile,
**gen_kwargs
)
return processor.decode(ids[0, plen:], skip_special_tokens=True)Nous utiliserons le site predict_one pour générer une réponse pour le 11e échantillon de l'ensemble de données. Il s'agit de préparer l'invite et d'exécuter la fonction de prédiction.
idx = 10
chat = test_data["messages"][idx]
prompt = processor.apply_chat_template(
chat,
add_generation_prompt=True,
tokenize=False
)
# run the one-sample helper
answer = predict_one(
prompt = prompt,
image = test_data["image"][idx],
model = model,
processor= processor,
max_new_tokens = 40
)
print("Model answer:", answer)En conséquence, nous avons reçu une longue phrase expliquant pourquoi le gliome cérébral avait été choisi. La réponse est complètement erronée, même la classification est erronée.
Model answer: Based on the MRI image, the most likely type of brain cancer is **A: brain glioma**.
Here's why:
* **Gliomas** are a common type of brain tumor11. Performance du modèle sur le modèle affiné
Pour évaluer le modèle affiné, nous répétons le processus d'évaluation en chargeant le modèle à partir du répertoire de sortie, en générant des prédictions et en calculant des mesures.
Nous chargeons le modèle affiné et le processeur à partir du répertoire de sortie, nous exécutons la fonction batch_predict et nous calculons les mesures.
model = AutoModelForImageTextToText.from_pretrained(
args.output_dir, **model_kwargs
)
model.generation_config = gen_cfg
processor = AutoProcessor.from_pretrained(args.output_dir)
tok = processor.tokenizer
model.config.pad_token_id = tok.pad_token_id
model.generation_config.pad_token_id = tok.pad_token_id
af_preds = batch_predict(
model = model,
processor = processor,
prompts = prompts,
images = images,
batch_size = 64,
max_new_tokens= 40, # forwarded to generate
postprocess= postprocess, # your label-mapping function
)
af_metrics = compute_metrics(af_preds)
print(f"Fine-tuned metrics: {af_metrics}")Ces résultats montrent une amélioration significative par rapport au modèle de base, soulignant l'efficacité du réglage fin. La précision est passée de 33 % à 89 % avec seulement 1 épisode.
Fine-tuned metrics: {'accuracy': 0.8927392739273927, 'f1': 0.892641793935792}Pour analyser plus en détail les performances du modèle affiné, nous générons une prédiction pour un seul exemple de l'ensemble de données de test.
idx = 10
chat = test_data["messages"][idx]
prompt = processor.apply_chat_template(
chat,
add_generation_prompt=True,
tokenize=False
)
# run the one-sample helper
answer = predict_one(
prompt = prompt,
image = test_data["image"][idx],
model = model,
processor= processor,
max_new_tokens = 40 # any generate-kwargs you need
)
print("Model answer:", answer)Le modèle a produit le résultat de manière claire et précise, la classification étant à la fois exacte et bien structurée.
Model answer: C: brain tumorSi vous rencontrez des problèmes lors de l'exécution du code ci-dessus, veuillez vous référer au cahier d'accompagnement : Fine_tuning_MedGemma.ipynb
Conclusion
MedGemma représente une avancée significative dans l'utilisation de l'IA pour les sciences médicales. En permettant aux médecins de porter des jugements plus rapides et plus précis, il permet des diagnostics plus rapides et des plans de traitement plus efficaces pour les patients.
L'affinement des modèles de langage de vision comme MedGemma 4B Instruct permet de s'adapter à diverses tâches médicales, de la classification d'images à l'intégration de capacités de raisonnement.
Dans ce tutoriel, nous avons appris à affiner un modèle vision-langage sur un ensemble de données d'IRM cérébrale pour la classification du cancer du cerveau. Les résultats ont été remarquables : la précision du modèle est passée de 33 % à 89 %, un bond considérable qui met en évidence le potentiel de l'ajustement dans les applications médicales de l'IA.
Si vous souhaitez en savoir plus, je vous recommande de consulter ces ressources :

En tant que data scientist certifié, je suis passionné par l'utilisation des technologies de pointe pour créer des applications innovantes d'apprentissage automatique. Avec une solide expérience en reconnaissance vocale, en analyse de données et en reporting, en MLOps, en IA conversationnelle et en NLP, j'ai affiné mes compétences dans le développement de systèmes intelligents qui peuvent avoir un impact réel. En plus de mon expertise technique, je suis également un communicateur compétent, doué pour distiller des concepts complexes dans un langage clair et concis. En conséquence, je suis devenu un blogueur recherché dans le domaine de la science des données, partageant mes idées et mes expériences avec une communauté grandissante de professionnels des données. Actuellement, je me concentre sur la création et l'édition de contenu, en travaillant avec de grands modèles linguistiques pour développer un contenu puissant et attrayant qui peut aider les entreprises et les particuliers à tirer le meilleur parti de leurs données.