Curso
MedGemma es una colección de variantes de Gemma 3 diseñadas para sobresalir en la comprensión de textos e imágenes médicas. La colección incluye actualmente dos potentes variantes: una versión multimodal 4B y una versión sólo texto 27B.
El modelo MedGemma 4B combina el SigLIP preentrenado en diversos conjuntos de datos médicos desidentificados, como radiografías de tórax, imágenes dermatológicas, imágenes oftalmológicas y preparaciones histopatológicas, con un gran modelo de lenguaje (LLM) entrenado en un amplio arreglo de datos médicos.
En este tutorial, aprenderemos a afinar el modelo MedGemma 4B en un conjunto de datos de IRM cerebral para una tarea de clasificación de imágenes. El objetivo es adaptar el modelo MedGemma 4B, más pequeño, para clasificar eficazmente las resonancias magnéticas cerebrales y predecir el cáncer cerebral con mayor precisión y eficacia.
Configurar el entorno
RunPod es una plataforma excelente para ejecutar cargas de trabajo basadas en la GPU, ya que ofrece entornos preconfigurados compatibles con JupyterLab. Esto te permite lanzar un pod y empezar a codificar inmediatamente utilizando tu editor preferido. A continuación te explicamos cómo configurar tu entorno:
1. Configurar el RunPod
Conéctate a RunPod y crea un pod con una GPU A100 y la última imagen de PyTorch. A continuación, haz clic en el botón "Desplegar bajo demanda" para lanzar el pod.
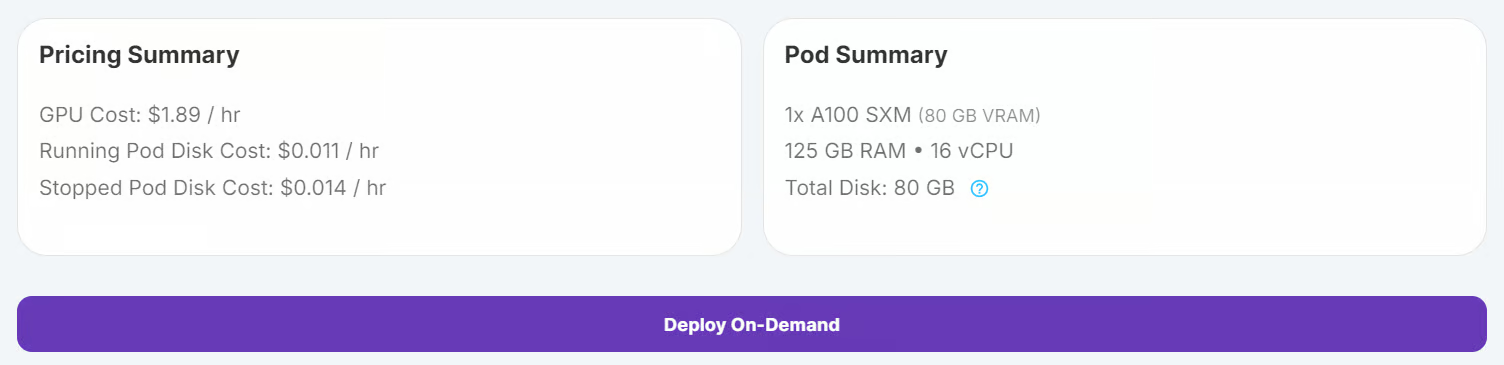
Edita el pod y añade las siguientes variables de entorno para la integración de Hugging Face y Kaggle:
- HF_TOKEN
- KAGGLE_USERNAME
- KAGGLE_KEY
Aumenta la capacidad de almacenamiento a 100 GB para alojar conjuntos de datos y puntos de comprobación de modelos
2. Instalar los paquetes de Python
Una vez que el pod esté en marcha, inicia la instancia de JupyterLab pulsando el botón "Conectar". Crea un nuevo cuaderno de Python e instala los paquetes de Python necesarios ejecutando el siguiente comando:
! pip install --upgrade --quiet transformers bitsandbytes datasets evaluate peft trl scikit-learn kaggleCargar y procesar el conjunto de datos
Utilizaremos el Cáncer cerebral de Kaggle.
3. Descargar el conjunto de datos
Descarga y descomprime el conjunto de datos utilizando la CLI de Kaggle:
!kaggle datasets download -d orvile/brain-cancer-mri-dataset --unzipDataset URL: https://www.kaggle.com/datasets/orvile/brain-cancer-mri-dataset
License(s): CC-BY-SA-4.0
Downloading brain-cancer-mri-dataset.zip to /workspace
90%|████████████████████████████████████▉ | 130M/144M [00:00<00:00, 231MB/s]
100%|█████████████████████████████████████████| 144M/144M [00:01<00:00, 108MB/s]4. Cargar el conjunto de datos
Carga el conjunto de datos como conjunto de datos Hugging Face, divídelo en conjuntos de entrenamiento y validación, y muestra su estructura:
from datasets import load_dataset
data_dir = "./Brain_Cancer raw MRI data/Brain_Cancer"
# Define proportions for train and validation splits
train_size = 0.8
validation_size = 0.2
data = load_dataset("imagefolder", data_dir=data_dir, split="train")
# Split the dataset into train and validation sets
data = data.train_test_split(
train_size=train_size,
test_size=validation_size,
shuffle=True,
seed=42,
)
# Rename the 'test' split to 'validation'
data["validation"] = data.pop("test")
# Display dataset details
print(data)Salida:
DatasetDict({
train: Dataset({
features: ['image', 'label'],
num_rows: 4844
})
validation: Dataset({
features: ['image', 'label'],
num_rows: 1212
})
})Comprueba la primera imagen y su etiqueta correspondiente del conjunto de entrenamiento:
data["train"][0]["image"]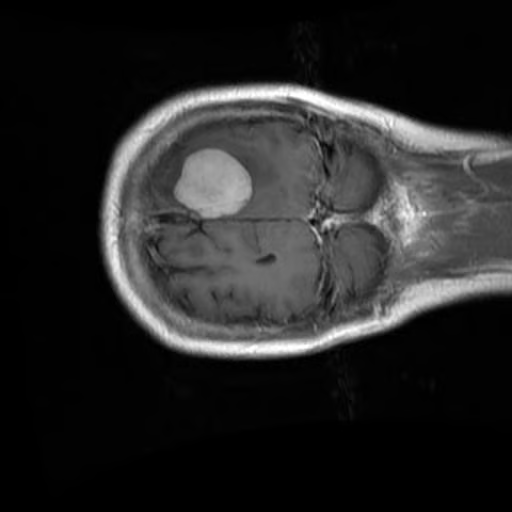
print(data["train"][0]["label"])15. Procesar el conjunto de datos
Antes de procesar el conjunto de datos, es importante comprobar primero los nombres de las etiquetas para garantizar un manejo adecuado de la tarea de clasificación.
BRAIN_CANCER_CLASSES = data["train"].features["label"].names
print("Detected classes:", BRAIN_CANCER_CLASSES)Las clases detectadas son: ['cerebro_glioma', 'cerebro_menina', 'cerebro_tumor'].
Detected classes: ['brain_glioma', 'brain_menin', 'brain_tumor']Para mejorar el proceso de clasificación, modificaremos estas etiquetas de clase añadiendo un prefijo (A, B, C) para organizarlas mejor y alinearlas con un formato de aviso personalizado.
BRAIN_CANCER_CLASSES = ['A: brain glioma', 'B: brain menin', 'C: brain tumor']A continuación, creamos una indicación personalizada que se utilizará para guiar al modelo durante el ajuste fino. El aviso incluye las etiquetas de clase actualizadas.
options = "\n".join(BRAIN_CANCER_CLASSES)
PROMPT = f"What is the most likely type of brain cancer shown in the MRI image?\n{options}"Para preparar el conjunto de datos para el ajuste fino, crearemos una nueva columna llamada "mensajes". Esta columna contendrá datos estructurados que representan una consulta del usuario (la pregunta) y la respuesta del asistente (la etiqueta correcta).
def format_data(example: dict[str, any]) -> dict[str, any]:
example["messages"] = [
{
"role": "user",
"content": [
{
"type": "image",
},
{
"type": "text",
"text": PROMPT,
},
],
},
{
"role": "assistant",
"content": [
{
"type": "text",
"text": BRAIN_CANCER_CLASSES[example["label"]],
},
],
},
]
return example
# Apply the formatting to the dataset
formatted_data = data.map(format_data)Para comprobar el formato, podemos inspeccionar la columna de mensajes de la primera muestra:
formatted_data["train"][0]["messages"]El punto de datos formateado resultante tiene este aspecto:
[{'content': [{'text': None, 'type': 'image'},
{'text': 'What is the most likely type of brain cancer shown in the MRI image?\nA: brain glioma\nB: brain menin\nC: brain tumor',
'type': 'text'}],
'role': 'user'},
{'content': [{'text': 'B: brain menin', 'type': 'text'}],
'role': 'assistant'}]Ajuste de MedGemma en el conjunto de datos de IRM cerebral
En este apartado, pondremos a punto el modelo MedGemma 4B Instruct en el conjunto de datos de RM cerebral. Esto implica descargar el modelo y el procesador, configurar el adaptador LoRA, configurar el entrenador e iniciar el proceso de entrenamiento.
6. Cargar el modelo y el tokenizador
Como MedGemma es un modelo cerrado, tienes que iniciar sesión en la CLI de Hugging Face utilizando tu clave API. Esto también te permite guardar tu modelo afinado en el Hub de Hugging Face. Consulta nuestro curso, Trabajar con Hugging Face: Tu guía del Hub, si necesitas un repaso .
from huggingface_hub import login
import os
hf_token = os.environ.get("HF_TOKEN")
login(hf_token)Utilizamos la biblioteca Transformers para cargar el modelo MedGemma 4B Instruct y su procesador. El modelo está configurado para utilizar precisión bfloat16 para un cálculo eficiente en GPUs.
import torch
from transformers import AutoProcessor, AutoModelForImageTextToText, BitsAndBytesConfig
model_id = "google/medgemma-4b-it"
# Check if GPU supports bfloat16
if torch.cuda.get_device_capability()[0] < 8:
raise ValueError("GPU does not support bfloat16, please use a GPU that supports bfloat16.")
model_kwargs = dict(
attn_implementation="eager",
torch_dtype=torch.bfloat16,
device_map="auto",
)
model = AutoModelForImageTextToText.from_pretrained(model_id, **model_kwargs)
processor = AutoProcessor.from_pretrained(model_id)
# Use right padding to avoid issues during training
processor.tokenizer.padding_side = "right"7. Configurar el modelo
Para ajustar eficazmente el modelo MedGemma 4B Instruct, utilizaremos Adaptación de bajo rango (LoRA)un método de ajuste fino eficiente desde el punto de vista de los parámetros.
LoRA nos permite adaptar grandes modelos entrenando sólo un pequeño número de parámetros adicionales, reduciendo significativamente los costes computacionales y manteniendo el rendimiento.
from peft import LoraConfig
peft_config = LoraConfig(
lora_alpha=16,
lora_dropout=0.05,
r=16,
bias="none",
target_modules="all-linear",
task_type="CAUSAL_LM",
modules_to_save=[
"lm_head",
"embed_tokens",
],
)Para manejar tanto las entradas de imagen como las de texto durante el entrenamiento, definimos una función de cotejo personalizada. Esta función procesa los ejemplos del conjunto de datos en un formato adecuado para el modelo, incluyendo la tokenización del texto y la preparación de los datos de imagen.
def collate_fn(examples: list[dict[str, any]]):
texts = []
images = []
for example in examples:
images.append([example["image"]])
texts.append(
processor.apply_chat_template(
example["messages"], add_generation_prompt=False, tokenize=False
).strip()
)
# Tokenize the texts and process the images
batch = processor(text=texts, images=images, return_tensors="pt", padding=True)
# The labels are the input_ids, with the padding and image tokens masked in
# the loss computation
labels = batch["input_ids"].clone()
# Mask image tokens
image_token_id = [
processor.tokenizer.convert_tokens_to_ids(
processor.tokenizer.special_tokens_map["boi_token"]
)
]
# Mask tokens that are not used in the loss computation
labels[labels == processor.tokenizer.pad_token_id] = -100
labels[labels == image_token_id] = -100
labels[labels == 262144] = -100
batch["labels"] = labels
return batchUtilizamos la clase SFTConfig de la bibliotecatrl para definir los argumentos de entrenamiento. Estos argumentos controlan el proceso de ajuste fino, incluyendo el tamaño del lote, el ritmo de aprendizaje y los pasos de acumulación del gradiente.
from trl import SFTConfig
args = SFTConfig(
output_dir="medgemma-brain-cancer",
num_train_epochs=1,
per_device_train_batch_size=8,
per_device_eval_batch_size=8,
gradient_accumulation_steps=8,
gradient_checkpointing=True,
optim="adamw_torch_fused",
logging_steps=0.1,
save_strategy="epoch",
eval_strategy="steps",
eval_steps=0.1,
learning_rate=2e-4,
bf16=True,
max_grad_norm=0.3,
warmup_ratio=0.03,
lr_scheduler_type="linear",
push_to_hub=True,
report_to="none",
gradient_checkpointing_kwargs={"use_reentrant": False},
dataset_kwargs={"skip_prepare_dataset": True},
remove_unused_columns = False,
label_names=["labels"],
)El SFTTrainer simplifica el proceso de ajuste combinando el modelo, el conjunto de datos, el cotejador de datos, los argumentos de entrenamiento y la configuración de LoRA en un único flujo de trabajo. Esto hace que el proceso sea ágil y fácil de usar.
from trl import SFTTrainer
trainer = SFTTrainer(
model=model,
args=args,
train_dataset=formatted_data["train"],
eval_dataset=formatted_data["validation"].shuffle().select(range(50)),
peft_config=peft_config,
processing_class=processor,
data_collator=collate_fn,
)8. Formación de modelos
Una vez establecidos el modelo, el conjunto de datos y las configuraciones de entrenamiento, podemos empezar el proceso de ajuste. El SFTTrainer simplifica este paso, permitiéndonos entrenar el modelo con un solo comando:
trainer.train()El proceso de formación duró aproximadamente 1 hora y 8 minutos. Durante este tiempo, la pérdida de entrenamiento y la pérdida de validación disminuían constantemente con cada paso, lo que indicaba que el modelo estaba aprendiendo eficazmente.
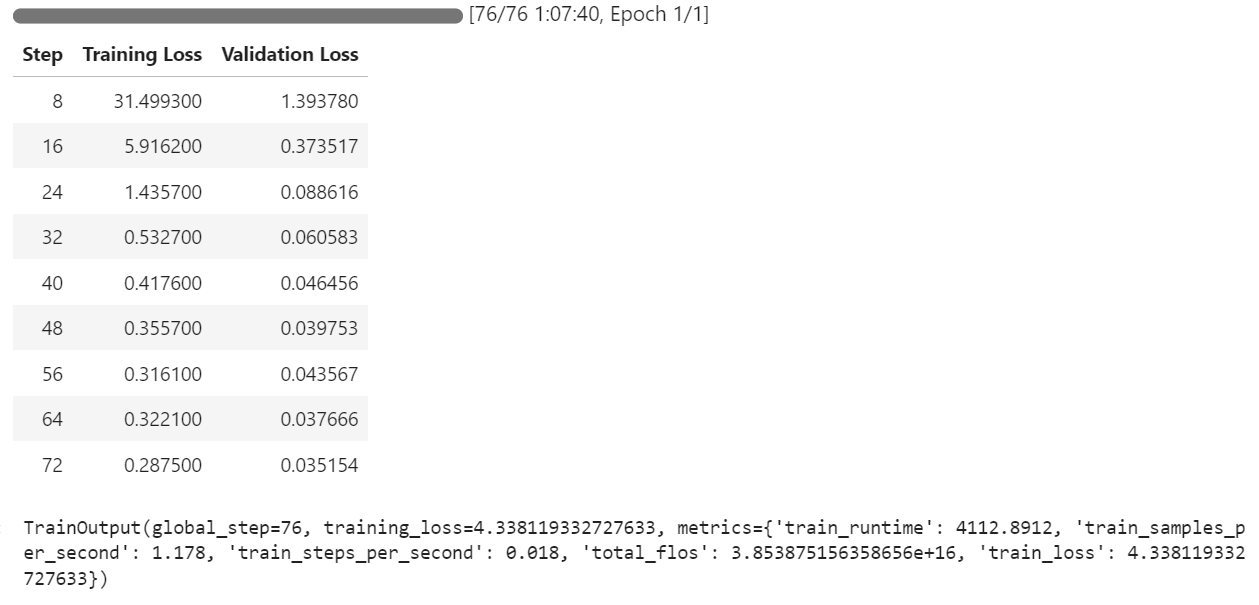
Una vez completado el entrenamiento, el modelo ajustado puede guardarse localmente y enviarse al Hub de Hugging Face mediante el método save_model().
trainer.save_model()El modelo ya está disponible kingabzpro/medgemma-cáncer-cerebral - Hugging Face
Fuente kingabzpro/medgemma-cáncer cerebral
Evaluación del modelo
Para evaluar el rendimiento del modelo MedGemma 4B, probaremos tanto el modelo base como el modelo afinado en el conjunto de datos de validación. Este proceso implica borrar la memoria, preparar los datos de la prueba, generar la respuesta y calcular métricas clave como la precisión y la puntuación F1.
Antes de iniciar la evaluación, eliminamos la configuración de entrenamiento para liberar memoria de la GPU y garantizar un entorno limpio para la prueba
del model
del trainer
torch.cuda.empty_cache()9. Preparación para la prueba de modelos
Formateamos el conjunto de datos de validación para que coincida con la estructura de entrada requerida por el modelo. Para ello hay que crear una columna "mensajes" que contenga el mensaje de usuario de cada ejemplo.
def format_test_data(example: dict[str, any]) -> dict[str, any]:
example["messages"] = [
{
"role": "user",
"content": [
{
"type": "image",
},
{
"type": "text",
"text": PROMPT,
},
],
},
]
return example
test_data = data["validation"]
test_data = test_data.map(format_test_data)Para evaluar el rendimiento del modelo, utilizamos la biblioteca evaluate, que proporciona métricas preconstruidas para tareas como la clasificación. Tras importar la biblioteca y cargar las métricas necesarias, extraemos las etiquetas verdaderas del conjunto de datos de prueba. A continuación, se define una función de ayuda, compute_metrics, para calcular la precisión y la puntuación F1 comparando las predicciones con estas etiquetas.
import evaluate
accuracy_metric = evaluate.load("accuracy")
f1_metric = evaluate.load("f1")
# Ground-truth labels
REFERENCES = test_data["label"]
def compute_metrics(predictions: list[int]) -> dict[str, float]:
metrics = {}
metrics.update(
accuracy_metric.compute(
predictions=predictions,
references=REFERENCES,
)
)
metrics.update(
f1_metric.compute(
predictions=predictions,
references=REFERENCES,
average="weighted",
)
)
return metricsPara garantizar la coherencia en el tratamiento de las etiquetas, convertimos la columna "etiqueta" del conjunto de datos en un tipo ClassLabel. Esto permite una correspondencia eficaz entre los índices de las etiquetas y sus nombres correspondientes. Además, definimos mapeados de etiquetas alternativos para manejar las variaciones en el formato de las etiquetas durante el postprocesado.
from datasets import ClassLabel
test_data = test_data.cast_column(
"label",
ClassLabel(names=BRAIN_CANCER_CLASSES)
)
LABEL_FEATURE = test_data.features["label"]
ALT_LABELS = dict([
(label, f"({label.replace(': ', ') ')}") for label in BRAIN_CANCER_CLASSES
])Para asignar las predicciones del modelo a las etiquetas de clase correctas, definimos una función postprocess. Esta función garantiza que las predicciones coincidan con la etiqueta adecuada, teniendo en cuenta los formatos de etiqueta canónicos y alternativos.
def postprocess(prediction, do_full_match: bool = False) -> int:
if isinstance(prediction, str):
response_text = prediction
else:
response_text = prediction[0]["generated_text"]
if do_full_match:
return LABEL_FEATURE.str2int(response_text)
for label in BRAIN_CANCER_CLASSES:
# accept canonical or alternative wording
if label in response_text or ALT_LABELS[label] in response_text:
return LABEL_FEATURE.str2int(label)
return -110. Rendimiento del modelo base
Para evaluar el rendimiento del modelo base, cargamos el modelo y el procesador preentrenados, configuramos los ajustes de generación y preparamos los avisos y las imágenes para la prueba.
import torch
from transformers import AutoModelForImageTextToText, AutoProcessor
model_kwargs = dict(
torch_dtype=torch.bfloat16,
device_map="auto",
)
model = AutoModelForImageTextToText.from_pretrained(
model_id, **model_kwargs
)
from transformers import GenerationConfig
gen_cfg = GenerationConfig.from_pretrained(model_id)
gen_cfg.update(
do_sample = False,
top_k = None,
top_p = None,
cache_implementation = "dynamic"
)
model.generation_config = gen_cfg
processor = AutoProcessor.from_pretrained(args.output_dir)
tok = processor.tokenizer
model.config.pad_token_id = tok.pad_token_id
model.generation_config.pad_token_id = tok.pad_token_id
def chat_to_prompt(chat_turns):
return processor.apply_chat_template(
chat_turns,
add_generation_prompt=True, # tells the model "your turn"
tokenize=False # we want raw text, not ids
)
prompts = [chat_to_prompt(c) for c in test_data["messages"]]
images = test_data["image"] # already a list of PIL images
assert len(prompts) == len(images), "1 prompt must match 1 image!"Hemos creado una función batch_predict que procesa el conjunto de datos de prueba por lotes. Esta función genera predicciones para cada par pregunta-imagen y aplica un postprocesamiento para asignar las salidas a las etiquetas correctas.
import torch
from typing import List, Any, Callable
def batch_predict(
prompts,
images,
model,
processor,
postprocess,
*,
batch_size=64,
device="cuda",
dtype=torch.bfloat16,
**gen_kwargs
):
preds = []
for i in range(0, len(prompts), batch_size):
texts = prompts[i : i + batch_size]
imgs = [[img] for img in images[i : i + batch_size]]
enc = processor(text=texts, images=imgs, padding=True, return_tensors="pt").to(
device, dtype=dtype
)
lens = enc["attention_mask"].sum(dim=1)
with torch.inference_mode():
out = model.generate(
**enc,
disable_compile=True,
**gen_kwargs
)
for seq, ln in zip(out, lens):
ans = processor.decode(seq[ln:], skip_special_tokens=True)
preds.append(postprocess(ans))
return predsUtilizaremos la función batch_predict para generar predicciones para el modelo base en el conjunto de datos de prueba. A continuación, las predicciones se evalúan mediante la función compute_metrics.
bf_preds = batch_predict(
model = model,
processor = processor,
prompts = prompts,
images = images,
batch_size = 64,
max_new_tokens= 40, # forwarded to generate
postprocess= postprocess, # your label-mapping function
)
bf_metrics = compute_metrics(bf_preds)
print(f"Baseline metrics: {bf_metrics}")El resultado no es impresionante. Obtuvimos un 33% de precisión, lo que es bastante malo.
Baseline metrics: {'accuracy': 0.33745874587458746, 'f1': 0.1737287617650654}Para comprender mejor el comportamiento del modelo, podemos generar predicciones para un solo ejemplo del conjunto de datos. Esto implica crear una función auxiliar que procese la entrada y devuelva la respuesta del modelo.
La función predict_one toma como entrada una pregunta y una imagen, las procesa utilizando el procesador del modelo y genera una respuesta. La función garantiza que la salida del modelo se descodifique en texto legible por humanos.
import torch
from typing import Union, Dict, Any, List
from transformers import AutoModelForImageTextToText, AutoProcessor
def predict_one(
prompt,
image,
model,
processor,
*,
device="cuda",
dtype=torch.bfloat16,
disable_compile=True,
**gen_kwargs
) -> str:
inputs = processor(text=prompt, images=image, return_tensors="pt").to(
device, dtype=dtype
)
plen = inputs["input_ids"].shape[-1]
with torch.inference_mode():
ids = model.generate(
**inputs,
disable_compile=disable_compile,
**gen_kwargs
)
return processor.decode(ids[0, plen:], skip_special_tokens=True)Utilizaremos la dirección predict_one para generar una respuesta para la 11ª muestra del conjunto de datos. Esto implica preparar el aviso y ejecutar la función de predicción.
idx = 10
chat = test_data["messages"][idx]
prompt = processor.apply_chat_template(
chat,
add_generation_prompt=True,
tokenize=False
)
# run the one-sample helper
answer = predict_one(
prompt = prompt,
image = test_data["image"][idx],
model = model,
processor= processor,
max_new_tokens = 40
)
print("Model answer:", answer)Como resultado, recibimos una larga sentencia en la que se explicaba por qué se había elegido el glioma cerebral. La respuesta es completamente errónea, incluso la clasificación es errónea.
Model answer: Based on the MRI image, the most likely type of brain cancer is **A: brain glioma**.
Here's why:
* **Gliomas** are a common type of brain tumor11. Rendimiento del modelo ajustado
Para evaluar el modelo afinado, repetimos el proceso de evaluación cargando el modelo desde el directorio de salida, generando predicciones y calculando métricas.
Cargamos el modelo afinado y el procesador desde el directorio de salida, ejecutamos la función batch_predict y calculamos las métricas .
model = AutoModelForImageTextToText.from_pretrained(
args.output_dir, **model_kwargs
)
model.generation_config = gen_cfg
processor = AutoProcessor.from_pretrained(args.output_dir)
tok = processor.tokenizer
model.config.pad_token_id = tok.pad_token_id
model.generation_config.pad_token_id = tok.pad_token_id
af_preds = batch_predict(
model = model,
processor = processor,
prompts = prompts,
images = images,
batch_size = 64,
max_new_tokens= 40, # forwarded to generate
postprocess= postprocess, # your label-mapping function
)
af_metrics = compute_metrics(af_preds)
print(f"Fine-tuned metrics: {af_metrics}")Estos resultados muestran una mejora significativa respecto al modelo base, lo que pone de relieve la eficacia del ajuste fino. La precisión pasó del 33% al 89% con sólo 1 epoch.
Fine-tuned metrics: {'accuracy': 0.8927392739273927, 'f1': 0.892641793935792}Para seguir analizando el rendimiento del modelo afinado, generamos una predicción para un solo ejemplo del conjunto de datos de prueba.
idx = 10
chat = test_data["messages"][idx]
prompt = processor.apply_chat_template(
chat,
add_generation_prompt=True,
tokenize=False
)
# run the one-sample helper
answer = predict_one(
prompt = prompt,
image = test_data["image"][idx],
model = model,
processor= processor,
max_new_tokens = 40 # any generate-kwargs you need
)
print("Model answer:", answer)El modelo produjo el resultado de forma clara y precisa, con una clasificación exacta y bien estructurada.
Model answer: C: brain tumorSi tienes problemas para ejecutar el código anterior, consulta el bloc de notas complementario: Fine_tuning_MedGemma.ipynb
Conclusión
MedGemma representa un importante paso adelante en el uso de la IA para las ciencias médicas. Al capacitar a médicos y facultativos para emitir juicios más rápidos y precisos, permite diagnósticos más rápidos y planes de tratamiento más eficaces para los pacientes.
El perfeccionamiento de los modelos de lenguaje visual, como MedGemma 4B Instruct, permite la adaptabilidad a diversas tareas médicas, desde la clasificación de imágenes hasta la integración de capacidades de razonamiento.
En este tutorial, hemos aprendido a afinar un modelo de visión-lenguaje en un conjunto de datos de resonancia magnética cerebral para la clasificación del cáncer cerebral. Los resultados fueron notables, ya que la precisión del modelo mejoró del 33% al 89%, un salto sustancial que pone de relieve el potencial del ajuste fino en las aplicaciones médicas de la IA.
Si te interesa saber más, te recomiendo que consultes estos recursos:

Soy un científico de datos certificado que disfruta creando aplicaciones de aprendizaje automático y escribiendo blogs sobre ciencia de datos. Actualmente me centro en la creación de contenidos, la edición y el trabajo con grandes modelos lingüísticos.


