Kurs
SVM bietet eine sehr hohe Genauigkeit im Vergleich zu anderen Klassifizierern wie der logistischen Regression und Entscheidungsbäumen. Es ist bekannt für seinen Kernel-Trick, um nichtlineare Eingangsräume zu behandeln. Sie wird in einer Vielzahl von Anwendungen eingesetzt, z. B. bei der Gesichtserkennung, der Erkennung von Eindringlingen, der Klassifizierung von E-Mails, Nachrichtenartikeln und Webseiten, der Klassifizierung von Genen und der Handschrifterkennung.
In diesem Lernprogramm wirst du scikit-learn in Python verwenden. Wenn du mehr über dieses Python-Paket erfahren möchtest, empfehle ich dir unseren Kurs Supervised Learning with scikit-learn.
Werde ein ML-Wissenschaftler
SVM ist ein spannender Algorithmus und die Konzepte sind relativ einfach. Der Klassifikator trennt Datenpunkte mithilfe einer Hyperebene mit dem größten Spielraum. Deshalb wird ein SVM-Klassifikator auch als diskriminativer Klassifikator bezeichnet. SVM findet eine optimale Hyperebene, die bei der Klassifizierung neuer Datenpunkte hilft.
In diesem Lernprogramm wirst du folgende Themen behandeln:
- Support-Vektor-Maschinen
- Wie funktioniert das?
- Kerne
- Erstellung von Klassifikatoren in Scikit-learn
- Hyperparameter abstimmen
- Vorteile und Nachteile
In diesem Video aus unserem Kurs erfährst du mehr über Support Vector Machines mit Scikit-learn.
Support-Vektor-Maschinen
Support Vector Machines werden im Allgemeinen als Klassifizierungsansatz betrachtet, können aber sowohl bei Klassifizierungs- als auch bei Regressionsproblemen eingesetzt werden. Es kann problemlos mehrere kontinuierliche und kategoriale Variablen verarbeiten. SVM konstruiert eine Hyperebene im mehrdimensionalen Raum, um verschiedene Klassen zu trennen. SVM erzeugt iterativ eine optimale Hyperebene, die zur Minimierung eines Fehlers verwendet wird. Die Kernidee von SVM ist es, eine maximale marginale Hyperebene (MMH) zu finden, die den Datensatz am besten in Klassen unterteilt.
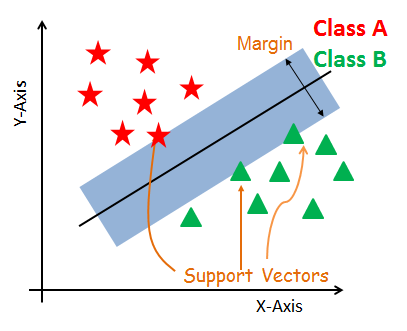
Vektoren unterstützen
Stützvektoren sind die Datenpunkte, die der Hyperebene am nächsten sind. Mit diesen Punkten wird die Trennlinie durch die Berechnung der Ränder besser definiert. Diese Punkte sind eher für die Konstruktion des Klassifikators relevant.
Hyperplane
Eine Hyperebene ist eine Entscheidungsebene, die zwischen einer Menge von Objekten mit unterschiedlichen Klassenzugehörigkeiten trennt.
Marge
Ein Rand ist eine Lücke zwischen den beiden Linien an den nächstgelegenen Klassenpunkten. Dieser wird als senkrechter Abstand von der Linie zu Stützvektoren oder nächstgelegenen Punkten berechnet. Wenn die Spanne zwischen den Klassen größer ist, wird sie als gut angesehen, eine kleinere Spanne ist eine schlechte Spanne.
Wie funktioniert die SVM?
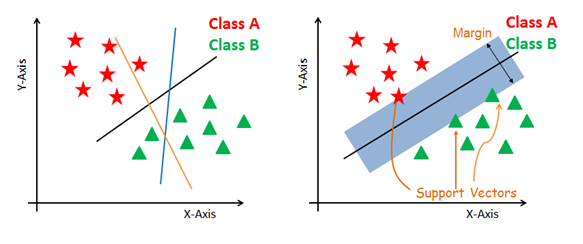
Das Hauptziel ist es, den gegebenen Datensatz auf die bestmögliche Weise zu trennen. Der Abstand zwischen den beiden nächstgelegenen Punkten wird als Marge bezeichnet. Das Ziel ist es, eine Hyperebene mit dem größtmöglichen Abstand zwischen den Stützvektoren im gegebenen Datensatz auszuwählen. SVM sucht in den folgenden Schritten nach der maximal marginalen Hyperebene:
-
Erstelle Hyperebenen, die die Klassen am besten voneinander trennen. Die Abbildung auf der linken Seite zeigt die drei Hyperebenen Schwarz, Blau und Orange. Hier haben Blau und Orange einen höheren Klassifizierungsfehler, aber das Schwarz trennt die beiden Klassen richtig.
-
Wähle die rechte Hyperebene mit der maximalen Trennung der beiden nächstgelegenen Datenpunkte aus, wie in der Abbildung rechts dargestellt.
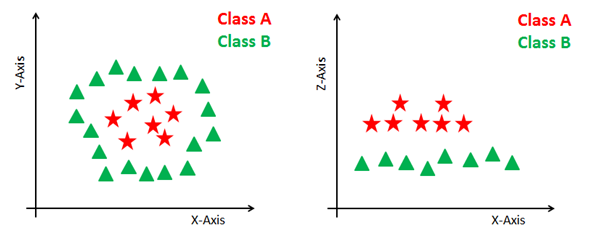
Umgang mit nicht-linearen und untrennbaren Ebenen
Einige Probleme können nicht mit der linearen Hyperebene gelöst werden, wie in der Abbildung unten (linke Seite) zu sehen ist.
In einer solchen Situation verwendet die SVM einen Kernel-Trick, um den Eingaberaum in einen höherdimensionalen Raum zu transformieren, wie rechts dargestellt. Die Datenpunkte werden auf der x-Achse und der z-Achse aufgetragen (Z ist die Quadratsumme von x und y: z=x^2=y^2). Jetzt kannst du diese Punkte ganz einfach mit der linearen Separation trennen.
SVM-Kernel
Der SVM-Algorithmus wird in der Praxis mit einem Kernel umgesetzt. Ein Kernel transformiert einen Eingabedatenraum in die gewünschte Form. SVM verwendet eine Technik namens Kernel-Trick. Dabei nimmt der Kernel einen niedrigdimensionalen Eingaberaum und transformiert ihn in einen höherdimensionalen Raum. Mit anderen Worten, man kann sagen, dass es nicht trennbare Probleme in trennbare Probleme umwandelt, indem es ihnen mehr Dimensionen verleiht. Sie ist besonders nützlich bei nichtlinearen Trennungsproblemen. Der Kernel-Trick hilft dir, einen genaueren Klassifikator zu erstellen.
- Linearer Kernel Ein linearer Kernel kann als normales Punktprodukt zweier gegebener Beobachtungen verwendet werden. Das Produkt zwischen zwei Vektoren ist die Summe der Multiplikation jedes Paares von Eingangswerten.
K(x, xi) = sum(x * xi)
- Polynomieller Kernel Ein polynomieller Kernel ist eine verallgemeinerte Form des linearen Kernels. Der Polynomkernel kann gekrümmte oder nichtlineare Eingangsräume unterscheiden.
K(x,xi) = 1 + sum(x * xi)^d
Dabei ist d der Grad des Polynoms. d=1 ist ähnlich wie die lineare Transformation. Der Grad muss im Lernalgorithmus manuell festgelegt werden.
- Radialer Basisfunktions-Kernel Der Radialer Basisfunktions-Kernel ist eine beliebte Kernel-Funktion, die häufig bei der Klassifizierung mit Support-Vektor-Maschinen verwendet wird. RBF kann einen Eingaberaum in einem unendlich dimensionalen Raum abbilden.
K(x,xi) = exp(-gamma * sum((x – xi^2))
Hier ist gamma ein Parameter, der von 0 bis 1 reicht. Ein höherer Gamma-Wert passt den Trainingsdatensatz perfekt an, was zu einer Überanpassung führt. Gamma=0,1 wird als guter Standardwert angesehen. Der Wert von Gamma muss im Lernalgorithmus manuell festgelegt werden.
Erstellung von Klassifikatoren in Scikit-learn
Bis jetzt hast du den theoretischen Hintergrund der SVM kennengelernt. Jetzt lernst du die Umsetzung in Python mit scikit-learn kennen.
Für die Modellbildung kannst du den Krebsdatensatz verwenden, der ein sehr bekanntes Mehrklassen-Klassifizierungsproblem darstellt. Dieser Datensatz wird aus einem digitalisierten Bild einer Feinnadelaspiration (FNA) einer Brustmasse berechnet. Sie beschreiben die Merkmale der im Bild vorhandenen Zellkerne.
Der Datensatz umfasst 30 Merkmale (mittlerer Radius, mittlere Textur, mittlerer Umfang, mittlere Fläche, mittlere Glätte, mittlere Kompaktheit, mittlere Konkavität, mittlere konkave Punkte, mittlere Symmetrie, mittlere fraktale Dimension, Radiusfehler, Texturfehler, Umfangsfehler, Flächenfehler, Glättungsfehler, Kompaktheitsfehler, Konkavitätsfehler, Konkavitätspunktefehler, Symmetriefehler, Fraktaldimensionsfehler, schlechtester Radius, schlechteste Textur, schlechtester Umfang, schlechteste Fläche, schlechteste Glätte, schlechteste Kompaktheit, schlechteste Konkavität, schlechteste Konkavität, schlechteste Symmetrie und schlechteste Fraktaldimension) und ein Ziel (Krebsart).
Diese Daten haben zwei Arten von Krebsklassen: bösartige (schädliche) und gutartige (nicht schädliche). Hier kannst du ein Modell erstellen, um die Art des Krebses zu klassifizieren. Der Datensatz ist in der scikit-learn-Bibliothek verfügbar oder du kannst ihn auch von der UCI Machine Learning Library herunterladen.
Code aus diesem Tutorial online ausführen und bearbeiten
Code ausführenDaten laden
Laden wir zunächst den benötigten Datensatz, den du verwenden wirst.
#Import scikit-learn dataset library
from sklearn import datasets
#Load dataset
cancer = datasets.load_breast_cancer()
Daten erforschen
Nachdem du den Datensatz geladen hast, möchtest du vielleicht noch ein bisschen mehr über ihn wissen. Du kannst die Namen von Merkmalen und Zielen überprüfen.
# print the names of the 13 features
print("Features: ", cancer.feature_names)
# print the label type of cancer('malignant' 'benign')
print("Labels: ", cancer.target_names)
Features: ['mean radius' 'mean texture' 'mean perimeter' 'mean area'
'mean smoothness' 'mean compactness' 'mean concavity'
'mean concave points' 'mean symmetry' 'mean fractal dimension'
'radius error' 'texture error' 'perimeter error' 'area error'
'smoothness error' 'compactness error' 'concavity error'
'concave points error' 'symmetry error' 'fractal dimension error'
'worst radius' 'worst texture' 'worst perimeter' 'worst area'
'worst smoothness' 'worst compactness' 'worst concavity'
'worst concave points' 'worst symmetry' 'worst fractal dimension']
Labels: ['malignant' 'benign']
Lass uns das noch ein bisschen genauer untersuchen. Du kannst auch die Form des Datensatzes mit shape überprüfen.
# print data(feature)shape
cancer.data.shape
(569, 30)
Schauen wir uns die Top 5 Datensätze des Featuresets an.
# print the cancer data features (top 5 records)
print(cancer.data[0:5])
[[1.799e+01 1.038e+01 1.228e+02 1.001e+03 1.184e-01 2.776e-01 3.001e-01
1.471e-01 2.419e-01 7.871e-02 1.095e+00 9.053e-01 8.589e+00 1.534e+02
6.399e-03 4.904e-02 5.373e-02 1.587e-02 3.003e-02 6.193e-03 2.538e+01
1.733e+01 1.846e+02 2.019e+03 1.622e-01 6.656e-01 7.119e-01 2.654e-01
4.601e-01 1.189e-01]
[2.057e+01 1.777e+01 1.329e+02 1.326e+03 8.474e-02 7.864e-02 8.690e-02
7.017e-02 1.812e-01 5.667e-02 5.435e-01 7.339e-01 3.398e+00 7.408e+01
5.225e-03 1.308e-02 1.860e-02 1.340e-02 1.389e-02 3.532e-03 2.499e+01
2.341e+01 1.588e+02 1.956e+03 1.238e-01 1.866e-01 2.416e-01 1.860e-01
2.750e-01 8.902e-02]
[1.969e+01 2.125e+01 1.300e+02 1.203e+03 1.096e-01 1.599e-01 1.974e-01
1.279e-01 2.069e-01 5.999e-02 7.456e-01 7.869e-01 4.585e+00 9.403e+01
6.150e-03 4.006e-02 3.832e-02 2.058e-02 2.250e-02 4.571e-03 2.357e+01
2.553e+01 1.525e+02 1.709e+03 1.444e-01 4.245e-01 4.504e-01 2.430e-01
3.613e-01 8.758e-02]
[1.142e+01 2.038e+01 7.758e+01 3.861e+02 1.425e-01 2.839e-01 2.414e-01
1.052e-01 2.597e-01 9.744e-02 4.956e-01 1.156e+00 3.445e+00 2.723e+01
9.110e-03 7.458e-02 5.661e-02 1.867e-02 5.963e-02 9.208e-03 1.491e+01
2.650e+01 9.887e+01 5.677e+02 2.098e-01 8.663e-01 6.869e-01 2.575e-01
6.638e-01 1.730e-01]
[2.029e+01 1.434e+01 1.351e+02 1.297e+03 1.003e-01 1.328e-01 1.980e-01
1.043e-01 1.809e-01 5.883e-02 7.572e-01 7.813e-01 5.438e+00 9.444e+01
1.149e-02 2.461e-02 5.688e-02 1.885e-02 1.756e-02 5.115e-03 2.254e+01
1.667e+01 1.522e+02 1.575e+03 1.374e-01 2.050e-01 4.000e-01 1.625e-01
2.364e-01 7.678e-02]]
Werfen wir einen Blick auf die Zielvorgabe.
# print the cancer labels (0:malignant, 1:benign)
print(cancer.target)
[0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 1 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0
1 0 0 0 0 0 0 0 0 1 0 1 1 1 1 1 0 0 1 0 0 1 1 1 1 0 1 0 0 1 1 1 1 0 1 0 0
1 0 1 0 0 1 1 1 0 0 1 0 0 0 1 1 1 0 1 1 0 0 1 1 1 0 0 1 1 1 1 0 1 1 0 1 1
1 1 1 1 1 1 0 0 0 1 0 0 1 1 1 0 0 1 0 1 0 0 1 0 0 1 1 0 1 1 0 1 1 1 1 0 1
1 1 1 1 1 1 1 1 0 1 1 1 1 0 0 1 0 1 1 0 0 1 1 0 0 1 1 1 1 0 1 1 0 0 0 1 0
1 0 1 1 1 0 1 1 0 0 1 0 0 0 0 1 0 0 0 1 0 1 0 1 1 0 1 0 0 0 0 1 1 0 0 1 1
1 0 1 1 1 1 1 0 0 1 1 0 1 1 0 0 1 0 1 1 1 1 0 1 1 1 1 1 0 1 0 0 0 0 0 0 0
0 0 0 0 0 0 0 1 1 1 1 1 1 0 1 0 1 1 0 1 1 0 1 0 0 1 1 1 1 1 1 1 1 1 1 1 1
1 0 1 1 0 1 0 1 1 1 1 1 1 1 1 1 1 1 1 1 1 0 1 1 1 0 1 0 1 1 1 1 0 0 0 1 1
1 1 0 1 0 1 0 1 1 1 0 1 1 1 1 1 1 1 0 0 0 1 1 1 1 1 1 1 1 1 1 1 0 0 1 0 0
0 1 0 0 1 1 1 1 1 0 1 1 1 1 1 0 1 1 1 0 1 1 0 0 1 1 1 1 1 1 0 1 1 1 1 1 1
1 0 1 1 1 1 1 0 1 1 0 1 1 1 1 1 1 1 1 1 1 1 1 0 1 0 0 1 0 1 1 1 1 1 0 1 1
0 1 0 1 1 0 1 0 1 1 1 1 1 1 1 1 0 0 1 1 1 1 1 1 0 1 1 1 1 1 1 1 1 1 1 0 1
1 1 1 1 1 1 0 1 0 1 1 0 1 1 1 1 1 0 0 1 0 1 0 1 1 1 1 1 0 1 1 0 1 0 1 0 0
1 1 1 0 1 1 1 1 1 1 1 1 1 1 1 0 1 0 0 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1
1 1 1 1 1 1 1 0 0 0 0 0 0 1]
Daten aufteilen
Um die Leistung des Modells zu verstehen, ist es eine gute Strategie, den Datensatz in einen Trainings- und einen Testsatz zu unterteilen.
Teile den Datensatz mit der Funktion train_test_split() auf. Du musst 3 Parameter übergeben: features, target und test_set size. Außerdem kannst du random_state verwenden, um Datensätze zufällig auszuwählen.
# Import train_test_split function
from sklearn.model_selection import train_test_split
# Split dataset into training set and test set
X_train, X_test, y_train, y_test = train_test_split(cancer.data, cancer.target, test_size=0.3,random_state=109) # 70% training and 30% test
Modell generieren
Lass uns ein Support-Vektor-Maschinenmodell erstellen. Importiere zunächst das SVM-Modul und erstelle ein Support-Vektor-Klassifikator-Objekt, indem du das Argument kernel als linearen Kernel in der Funktion SVC() übergibst.
Dann passt du dein Modell mit fit() an die Trainingsmenge an und machst mit predict() eine Vorhersage für die Testmenge.
#Import svm model
from sklearn import svm
#Create a svm Classifier
clf = svm.SVC(kernel='linear') # Linear Kernel
#Train the model using the training sets
clf.fit(X_train, y_train)
#Predict the response for test dataset
y_pred = clf.predict(X_test)
Bewertung des Modells
Lass uns abschätzen, wie genau der Klassifikator oder das Modell den Brustkrebs der Patientinnen vorhersagen kann.
Die Genauigkeit kann berechnet werden, indem die tatsächlichen Werte der Testreihe mit den vorhergesagten Werten verglichen werden.
#Import scikit-learn metrics module for accuracy calculation
from sklearn import metrics
# Model Accuracy: how often is the classifier correct?
print("Accuracy:",metrics.accuracy_score(y_test, y_pred))
Accuracy: 0.9649122807017544
Nun, du hast eine Klassifizierungsrate von 96,49%, was als sehr gute Genauigkeit gilt.
Zur weiteren Auswertung kannst du auch die Genauigkeit und die Wiedererkennung des Modells überprüfen.
# Model Precision: what percentage of positive tuples are labeled as such?
print("Precision:",metrics.precision_score(y_test, y_pred))
# Model Recall: what percentage of positive tuples are labelled as such?
print("Recall:",metrics.recall_score(y_test, y_pred))
Precision: 0.9811320754716981
Recall: 0.9629629629629629
Nun, du hast eine Genauigkeit von 98% und einen Recall von 96%, was als sehr gute Werte gilt.
Hyperparameter abstimmen
- Kernel: Die Hauptfunktion des Kernels besteht darin, die gegebenen Eingabedaten in die gewünschte Form umzuwandeln. Es gibt verschiedene Arten von Funktionen wie lineare, polynomiale und radiale Basisfunktionen (RBF). Polynom und RBF sind nützlich für nichtlineare Hyperebenen. Polynomiale und RBF-Kerne berechnen die Trennlinie in der höheren Dimension. Bei einigen Anwendungen wird vorgeschlagen, einen komplexeren Kernel zu verwenden, um die gekrümmten oder nichtlinearen Klassen zu trennen. Diese Umwandlung kann zu genaueren Klassifikatoren führen.
- Regularisierung: Regularisierungsparameter in Pythons Scikit-learn C-Parameter, der zur Aufrechterhaltung der Regularisierung verwendet wird. Hier ist C der Strafparameter, der die Fehlklassifizierung oder den Fehlerterm darstellt. Der Fehlklassifizierungs- oder Fehlerterm sagt der SVM-Optimierung, wie viel Fehler vertretbar ist. So kannst du den Kompromiss zwischen Entscheidungsgrenze und Fehlklassifizierungsterm kontrollieren. Ein kleinerer Wert von C erzeugt eine Hyperebene mit kleinem Rand und ein größerer Wert von C erzeugt eine Hyperebene mit größerem Rand.
- Gamma: Ein niedriger Wert von Gamma passt sich dem Trainingsdatensatz locker an, während ein höherer Wert von Gamma den Trainingsdatensatz genau anpasst, was zu einer Überanpassung führt. Mit anderen Worten: Ein niedriger Wert von Gamma berücksichtigt bei der Berechnung der Trennlinie nur nahe gelegene Punkte, während ein Wert von Gamma alle Datenpunkte bei der Berechnung der Trennlinie berücksichtigt.
Vorteile
SVM-Klassifikatoren bieten eine gute Genauigkeit und eine schnellere Vorhersage als der Naïve Bayes-Algorithmus. Außerdem benötigen sie weniger Speicherplatz, weil sie in der Entscheidungsphase eine Teilmenge der Trainingspunkte verwenden. SVM funktioniert gut bei einer klaren Trennungsgrenze und bei einem hochdimensionalen Raum.
Benachteiligungen
SVM ist wegen seiner hohen Trainingszeit nicht für große Datensätze geeignet und benötigt im Vergleich zu Naïve Bayes auch mehr Zeit für das Training. Es funktioniert schlecht bei sich überschneidenden Klassen und ist auch empfindlich gegenüber der Art des verwendeten Kernels.
Fazit
Glückwunsch, du hast es bis zum Ende dieses Tutorials geschafft!
In diesem Tutorium hast du viel über den Support-Vector-Machine-Algorithmus, seine Funktionsweise, die Kernel, die Abstimmung der Hyperparameter, den Aufbau des Modells und die Auswertung des Brustkrebs-Datensatzes mit dem Scikit-Learn-Paket gelernt. Du hast auch auf die Vor- und Nachteile eingegangen. Ich hoffe, du hast etwas Wertvolles gelernt!
Um mehr über diese Art von Klassifikatoren zu erfahren, solltest du dir unseren Kurs Lineare Klassifikatoren in Python ansehen. Es werden andere Arten von Regressions- und Verlustfunktionen sowie Support Vector Machines vorgestellt.
Ich freue mich über jedes Feedback und jede Frage. Du kannst die Frage stellen, indem du einen Kommentar hinterlässt und ich werde mein Bestes tun, um sie zu beantworten.