Curso
Ministral 3 es una nueva familia de modelos multimodales optimizados para el borde, disponibles en tamaños de 3B, 8B y 14B, cada uno de los cuales ofrece variantes Base, Instruct y Reasoning con soporte visual completo.
Diseñado para ofrecer un rendimiento de vanguardia sin renunciar a la ligereza necesaria para su implementación local y privada, Ministral 3 combina capacidades avanzadas con limitaciones prácticas de hardware. Su modelo insignia, el 14B Instruct, cuenta con 24 GB de VRAM en FP8 y rivaliza con sistemas más grandes como el Mistral Small 3.2 24B. Con una amplia ventana de contexto de 256 k, una sólida cobertura multilingüe, llamadas a funciones nativas, un estricto cumplimiento de las indicaciones del sistema y licencia abierta Apache 2.0.
En este tutorial, ajustaremos el modelo Ministral 3 14B Instruct en un conjunto de datos de radiografías de fracturas óseas para mejorar su capacidad de detectar fracturas y clasificar sus categorías con precisión.
Aprenderás a:
- Carga el conjunto de datos desde Kaggle.
- Conviértelo en un conjunto de datos de Hugging Face.
- Crea una indicación clara para el entrenamiento en la clasificación de rayos X.
- Configura el proceso de entrenamiento de lenguaje visual Ministral 3 14B.
- Ajusta el modelo y guarda el punto de control.
- Carga el adaptador del modelo entrenado para la inferencia.
- Compara los resultados antes y después del ajuste.
Si deseas obtener más información sobre los modelos de IA de Mistral, te recomiendo nuestra guía Mistral 3 y tutorial de Mistral 3 Large.
Descargo de responsabilidad: Este tutorial muestra un proceso de ajuste técnico con fines exclusivamente educativos. El modelo resultante es no está destinado a uso médico o clínico y no debe utilizarse como base para el diagnóstico, la toma de decisiones o la atención al paciente.
1. Configuración del entorno para el ajuste fino de Ministral 3 Vision-Language
Aunque estamos ajustando el modelo 14B, el entrenamiento del lenguaje visual requiere mucha más memoria que el ajuste solo de texto. El codificador de imágenes aumenta la carga de la GPU, lo que significa que necesitamos acceder a una GPU A100 con 80 GB de VRAM para ejecutar este proceso sin problemas.
Para este proyecto, estoy utilizando Runpod y lanzando un pod A100.
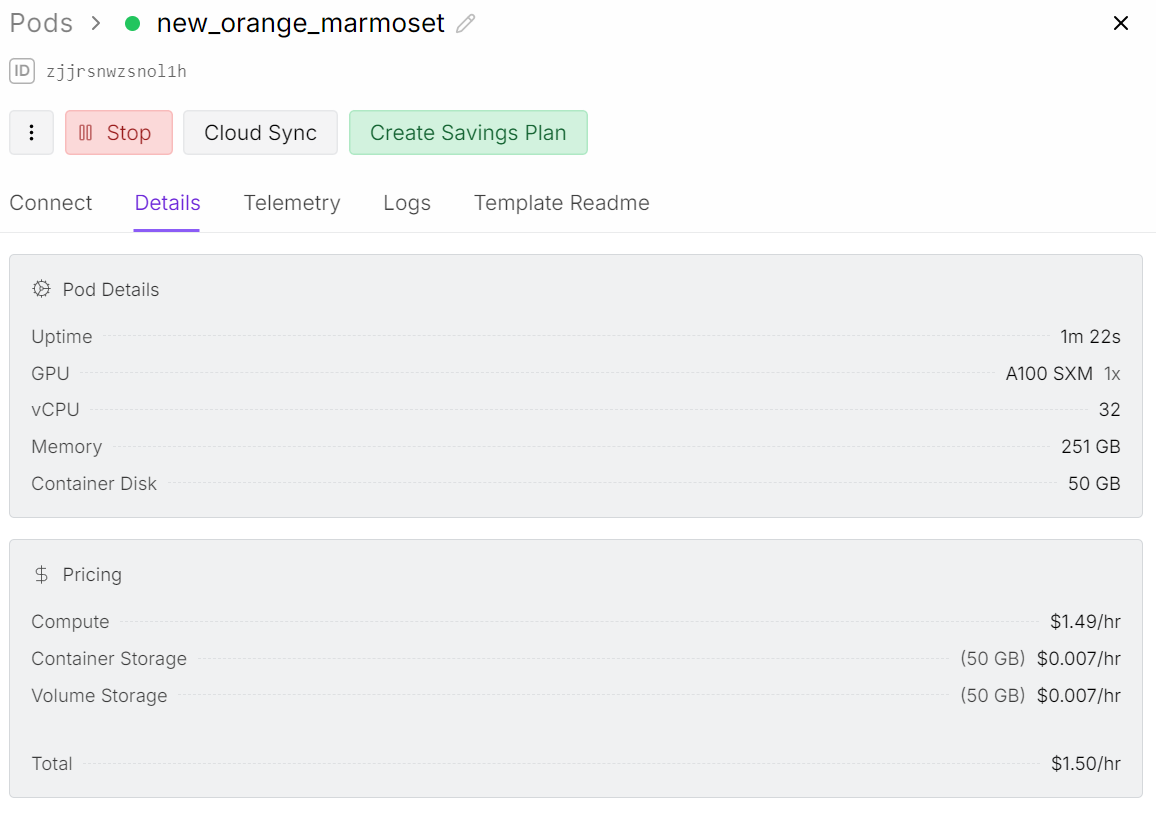
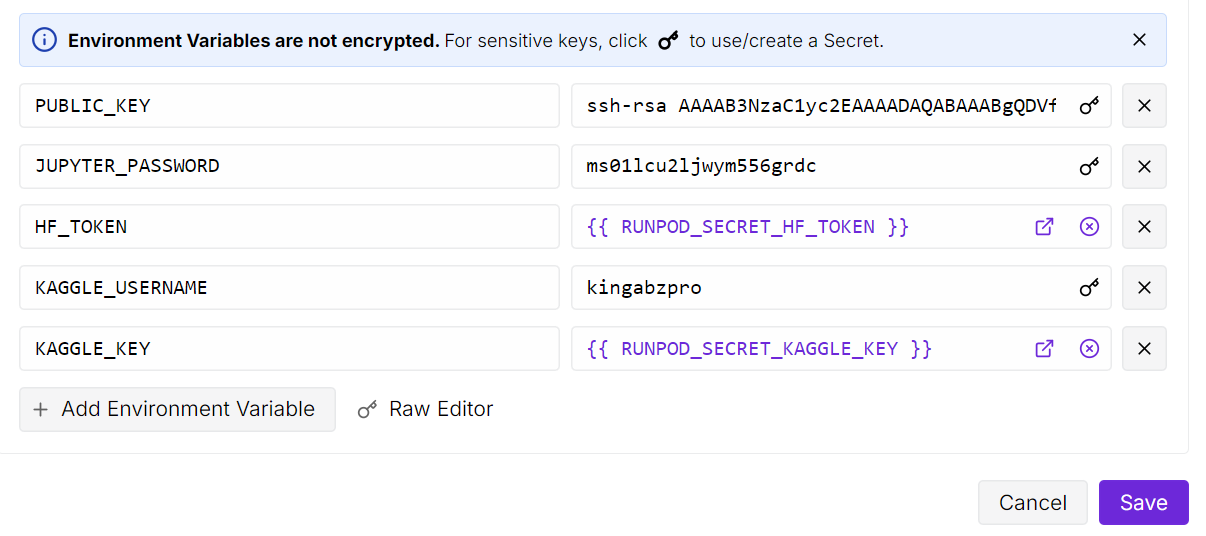
Después de iniciar el pod, aumenta el almacenamiento del contenedor a 50 GB. A continuación, añade las variables de entorno para las claves API de Kaggle y Hugging Face.
Kaggle nos permitirá descargar directamente el conjunto de datos de radiografías óseas, y Hugging Face nos permitirá volver a enviar nuestro adaptador LoRA ajustado al centro de modelos.
A continuación, abre la instancia de JupyterLab y crea un cuaderno nuevo. Antes de empezar con la formación, es necesario instalar todos los paquetes de Python necesarios.
Asegúrate de que la biblioteca Transformers esté actualizada a la última versión, ya que recientemente se ha añadido la compatibilidad con Ministral 3 y las versiones anteriores no cargarán el modelo correctamente.
! pip install --upgrade --quiet bitsandbytes datasets evaluate hf_transfer peft scikit-learn matplotlib Pillow kaggle ipywidgets
!pip install --quiet git+https://github.com/huggingface/transformers.git@bf3f0ae70d0e902efab4b8517fce88f6697636ce
!pip install --quiet --no-deps trl==0.22.2Una vez que todo esté instalado, estarás listo para cargar el conjunto de datos, preparar el clasificador y comenzar con el ajuste fino.
2. Descarga del conjunto de datos sobre fracturas óseas de Kaggle
En el siguiente paso, creamos una carpeta raíz para vuestro conjunto de datos y utilizamos la API de Python de Kaggle para descargar el conjunto de datos Bone Break Classification Image.
Este conjunto de datos contiene varias categorías de fracturas óseas en formato de rayos X y se utilizará para ajustar Ministral 3 para la clasificación visual.
import os
import glob
from kaggle.api.kaggle_api_extended import KaggleApi
DATA_ROOT = "/bone-break"
os.makedirs(DATA_ROOT, exist_ok=True)
kaggle_dataset = "pkdarabi/bone-break-classification-image-dataset"
# Initialize and authenticate Kaggle API (it will read KAGGLE_USERNAME / KAGGLE_KEY)
api = KaggleApi()
api.authenticate()
# Download and unzip the dataset using the Python API
api.dataset_download_files(
kaggle_dataset,
path=DATA_ROOT,
unzip=True,
)Puedes ver el conjunto de datos en Kaggle aquí:
Dataset URL: https://www.kaggle.com/datasets/pkdarabi/bone-break-classification-image-dataset3. Creación de un conjunto de datos Hugging Face
El conjunto de datos descargado no está organizado con la estructura habitual. Cada categoría de fractura contiene una carpeta de entrenamiento y una carpeta de pruebas en su interior.
Así que, en primer lugar, necesitamos un método para recorrer todas las carpetas de clases, separar las dos divisiones y, a continuación, crear un conjunto de datos combinado de Hugging Face con una asignación limpia de entrenamiento y prueba.
import glob, os
from typing import Any, Dict, List
from datasets import Dataset, DatasetDict, Features, ClassLabel, Image
inner_root = os.path.join(
DATA_ROOT, "Bone Break Classification", "Bone Break Classification"
)
classes = []
train_records: List[Dict[str, Any]] = []
test_records: List[Dict[str, Any]] = []
for class_name in sorted(os.listdir(inner_root)):
class_dir = os.path.join(inner_root, class_name)
if not os.path.isdir(class_dir):
continue
classes.append(class_name)
for split_name, split_key in [("Train", "train"), ("Test", "test")]:
split_dir = os.path.join(class_dir, split_name)
if not os.path.isdir(split_dir):
continue
for ext in ("*.png", "*.jpg", "*.jpeg"):
for img_path in glob.glob(os.path.join(split_dir, ext)):
rec = {"image": img_path, "label_name": class_name}
if split_key == "train":
train_records.append(rec)
else:
test_records.append(rec)
print("Classes:", classes)
print("Train samples:", len(train_records))
print("Test samples:", len(test_records))Tenemos un total de 10 clasificaciones, con 989 muestras de entrenamiento y 140 muestras de prueba.
Classes: ['Avulsion fracture', 'Comminuted fracture', 'Fracture Dislocation', 'Greenstick fracture', 'Hairline Fracture', 'Impacted fracture', 'Longitudinal fracture', 'Oblique fracture', 'Pathological fracture', 'Spiral Fracture']
Train samples: 989
Test samples: 140Ahora codificaremos las etiquetas del conjunto de datos Hugging Face para que cada clase de etiqueta se considere como etiqueta de clase y cada imagen se trate como característica de imagen.
Crearemos dos conjuntos de datos: uno para entrenamiento y otro para pruebas. Por último, los combinaremos en un único conjunto de datos que contenga tanto subconjuntos de entrenamiento como de prueba.
# Encode labels
classes = sorted(list(set(classes)))
label2id = {name: i for i, name in enumerate(classes)}
for rec in train_records:
rec["label"] = label2id[rec["label_name"]]
for rec in test_records:
rec["label"] = label2id[rec["label_name"]]
features = Features(
{
"image": Image(),
"label": ClassLabel(names=classes),
}
)
train_ds = Dataset.from_list(
[{"image": r["image"], "label": r["label"]} for r in train_records],
features=features,
)
test_ds = Dataset.from_list(
[{"image": r["image"], "label": r["label"]} for r in test_records],
features=features,
)
data = DatasetDict({"train": train_ds, "test": test_ds})
dataAquí tienes una descripción general del conjunto de datos.
DatasetDict({
train: Dataset({
features: ['image', 'label'],
num_rows: 989
})
test: Dataset({
features: ['image', 'label'],
num_rows: 140
})
})4. Exploración de la distribución de clases y ejemplos de imágenes de rayos X
Antes de realizar ajustes, es útil comprender qué tan equilibrado está el conjunto de datos y cómo se ven las imágenes. Comenzaremos por comprobar cuántas muestras de entrenamiento existen para cada categoría de fractura.
from collections import Counter
label_names = data["train"].features["label"].names
label_ids = data["train"]["label"]
counts = Counter(label_ids)
print("Training Class counts:")
for lbl_id, cnt in counts.items():
print(f"{lbl_id:2d} ({label_names[lbl_id]}): {cnt}")
FRACTURE_CLASSES = label_namesLas categorías están bastante equilibradas, aunque el tamaño total de la muestra es pequeño para una tarea compleja de clasificación de imágenes.
Por ahora, continuaremos con este conjunto de datos, pero los resultados deben interpretarse con expectativas realistas debido al volumen limitado de la muestra.
Training Class counts:
0 (Avulsion fracture): 109
1 (Comminuted fracture): 134
2 (Fracture Dislocation): 137
3 (Greenstick fracture): 106
4 (Hairline Fracture): 101
5 (Impacted fracture): 75
6 (Longitudinal fracture): 68
7 (Oblique fracture): 69
8 (Pathological fracture): 116
9 (Spiral Fracture): 74
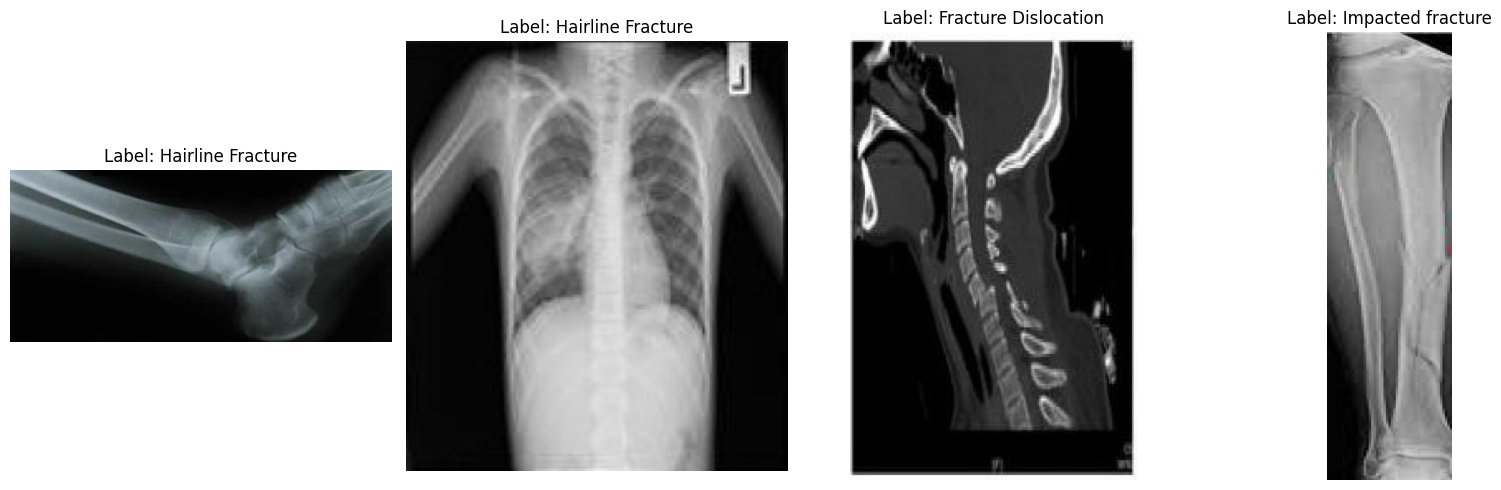
A continuación, veamos algunas imágenes aleatorias para comprender mejor el conjunto de datos de forma visual.
import matplotlib.pyplot as plt
from datasets import Image
# Cast the 'image' column to Hugging Face Image feature
dataset_images = data.cast_column("image", Image())
# Select random samples from the training set
samples = dataset_images["train"].shuffle(seed=420).select(range(4))
# Plot
fig, axes = plt.subplots(1, 4, figsize=(16, 5))
for i, sample in enumerate(samples):
img_object = sample["image"] # PIL Image
label_id = sample["label"] # int
label_name = label_names[label_id] # string
axes[i].imshow(img_object)
axes[i].set_title(f"Label: {label_name}")
axes[i].axis("off")
plt.tight_layout()
plt.show()Al observar las imágenes de muestra, podemos apreciar diferencias en cuanto a tamaño, resolución, orientación y calidad de escaneo. Esto no afecta al ajuste fino de un modelo de lenguaje visual, pero puede influir en la coherencia con la que el modelo aprende cada categoría de fractura.
Detectar una fractura a simple vista suele ser sencillo, pero clasificarla en uno de los diez tipos médicos de fracturas sutiles requiere datos más estructurados que los que se proporcionan aquí.
5. Diseño rápido para la clasificación de fracturas óseas
A continuación, debemos decidir cómo queremos que se comporte el modelo. Para esta tarea, nuestro objetivo es muy estricto: dada una imagen de rayos X, el modelo debe generar solo el nombre de una clase de fractura y nada más. Sin explicaciones, sin descuentos, sin texto adicional.
Escribiremos una instrucción clara y la utilizaremos para crear una columna « messages » en el conjunto de datos.
PROMPT = f"""
You are a radiology assistant specialised in bone fracture classification.
You must classify the X-ray image into exactly ONE of the following classes:
{", ".join(FRACTURE_CLASSES)}
Rules:
- Reply with ONLY the class name.
- No explanations, no extra words, no markdown, no punctuation.
- Output must be EXACTLY one of: {", ".join(FRACTURE_CLASSES)}
""".strip()
from typing import Dict, Any
def convert_to_conversation(example: Dict[str, Any]) -> Dict[str, Any]:
conversation = [
{
"role": "user",
"content": [
{"type": "text", "text": PROMPT},
{"type": "image"}, # no image bytes here
],
},
{
"role": "assistant",
"content": [
{"type": "text", "text": FRACTURE_CLASSES[example["label"]]},
],
},
]
example["messages"] = conversation
return example
formatted_data = data["train"].map(convert_to_conversation)
formatted_data["messages"][0]Esto crea una columna « messages » (Clasificación de fracturas) en la que cada registro contiene ahora una miniconversación completa: la instrucción más la clase de fractura correcta. Esto es exactamente lo que verá el modelo de lenguaje visual durante el ajuste fino.

6. Cargando el modelo de lenguaje visual Ministral 3
Ahora cargamos la versión BF16 del modelo Ministral-3 14B Instruct desde Hugging Face Hub, junto con el procesador. El procesador agrupa el tokenizador y el canal de preprocesamiento de imágenes, necesario para las entradas de lenguaje visual.
import torch
from transformers import AutoProcessor, AutoModelForImageTextToText, BitsAndBytesConfig
model_id = "mistralai/Ministral-3-14B-Instruct-2512-BF16"
model_kwargs = dict(
attn_implementation="eager",
torch_dtype=torch.bfloat16,
device_map="auto",
)
model = AutoModelForImageTextToText.from_pretrained(model_id, **model_kwargs)
processor = AutoProcessor.from_pretrained(model_id)
# Use right padding to avoid issues during training
processor.tokenizer.padding_side = "right"7. Configuración de una plantilla de chat personalizada
La plantilla de chat original proporcionada por Mistral AI está diseñada para casos de uso prolongados y conversacionales. Para este proyecto, eso se convirtió en un problema. El modelo comenzó a centrarse en las largas instrucciones de las funciones y el texto del sistema, y pasó por alto el objetivo real, que es predecir una única clase de fractura ósea a partir de la radiografía.
Para solucionar esto, definimos una plantilla de lenguaje visual más breve que solo conserva las partes importantes: las instrucciones del usuario y la respuesta del asistente. Esto reduce el ruido y ayuda al modelo a aprender una correspondencia clara entre la imagen y la etiqueta de clase.
SHORT_VLM_TEMPLATE = r"""{{ bos_token }}[INST]
{% for message in messages if message['role'] == 'user' -%}
{%- for content in message['content'] -%}
{%- if content['type'] == 'text' -%}
{{ content['text'] }}
{%- elif content['type'] == 'image' -%}
[IMG]
{%- endif -%}
{%- endfor -%}
{%- endfor -%}
[/INST]{% if not add_generation_prompt %}
{%- for message in messages if message['role'] == 'assistant' -%}
{{ message['content'][0]['text'] }}{{ eos_token }}
{%- endfor -%}
{%- endif %}"""
# Attach to processor / tokenizer
processor.chat_template = SHORT_VLM_TEMPLATE
processor.tokenizer.chat_template = SHORT_VLM_TEMPLATE8. Prueba de Ministral 3 (preajuste)
Antes de empezar con los ajustes, es útil probar el modelo original Ministral 3 14B Instruct para crear una referencia.
En primer lugar, proporcionamos al modelo una imagen aleatoria del conjunto de datos y le hacemos una pregunta general, solo para ver cómo entiende el contenido de la radiografía.
image = formatted_data[200]["image"]
messages = [
{"role": "user", "content": [
{"type": "text", "text": "What is the image about?"},
{"type": "image"},
]}
]
input_text = processor.apply_chat_template(messages, add_generation_prompt = True)
inputs = processor(
image,
input_text,
add_special_tokens = False,
return_tensors = "pt",
).to(device="cuda", dtype=torch.bfloat16)
from transformers import TextStreamer
text_streamer = TextStreamer(processor, skip_prompt = True)
_ = model.generate(**inputs, streamer = text_streamer, max_new_tokens = 1000,
use_cache = True, temperature = 1.5, min_p = 0.1)Como resultado, el modelo entendió el contenido general de la radiografía, pero luego se desvió hacia descripciones que no eran fiables para uso médico.

A continuación, probamos algo más cercano a nuestro objetivo final. Damos la misma imagen, pero esta vez utilizamos nuestra estricta clasificación de fracturas en lugar de una pregunta abierta.
image = formatted_data[200]["image"]
messages = [
{"role": "user", "content": [
{"type": "text", "text": PROMPT},
{"type": "image"},
]}
]
input_text = processor.apply_chat_template(messages, add_generation_prompt = True)
inputs = processor(
image,
input_text,
add_special_tokens = False,
return_tensors = "pt",
).to(device="cuda", dtype=torch.bfloat16)
from transformers import TextStreamer
text_streamer = TextStreamer(processor, skip_prompt = True)
_ = model.generate(**inputs, streamer = text_streamer, max_new_tokens = 1000,
use_cache = True, temperature = 1.5, min_p = 0.1Para esta muestra, el modelo respondió con:
Hairline Fracture</s>Ahora comprobamos la etiqueta de verdad fundamental real para la misma imagen.
FRACTURE_CLASSES[formatted_data[200]["label"]]Esto devuelve:
'Comminuted fracture'Por lo tanto, el modelo siguió correctamente el formato de las instrucciones y generó un único nombre de clase, pero la clasificación en sí era incorrecta.
Por eso precisamente necesitamos un ajuste fino. El modelo básico tiene una gran capacidad de comprensión visual, pero no ha sido entrenado específicamente para distinguir entre tipos detallados de fracturas óseas.
En los siguientes pasos, lo ajustaremos en vuestro conjunto de datos de fracturas para mejorar ese comportamiento.
9. Configuración de LoRA para un ajuste fino eficiente de Ministral 3
A continuación, configuramos LoRA para ajustar el modelo Ministral 3 de manera eficiente sin actualizar todos los parámetros. Esta configuración se centra en las capas de atención principal y proyección MLP utilizadas en un modelo de visión-lenguaje de tipo Mistral.
from peft import LoraConfig, TaskType, get_peft_model
# LoRA config for a Mistral-style VLM used as a causal LM
peft_config = LoraConfig(
task_type=TaskType.CAUSAL_LM,
r=16,
lora_alpha=32,
lora_dropout=0.05,
bias="none",
target_modules=[
"q_proj",
"k_proj",
"v_proj",
"o_proj",
"gate_proj",
"up_proj",
"down_proj",
],
modules_to_save=None,
)A continuación, definimos los argumentos de entrenamiento de ajuste supervisado (SFT).
Esta configuración controla todo el proceso de entrenamiento, incluyendo cómo se gestionan los lotes, la optimización de la memoria, el comportamiento de la tasa de aprendizaje, el registro, la estrategia de almacenamiento y la integración con Hugging Face Hub.
from trl import SFTConfig
args = SFTConfig(
output_dir="ministral-3-bone-fracture",
num_train_epochs=1,
per_device_train_batch_size=2,
per_device_eval_batch_size=2,
gradient_accumulation_steps=4,
gradient_checkpointing=True,
optim="adamw_8bit",
logging_steps=0.1,
save_strategy="epoch",
learning_rate=2e-4,
bf16=True,
fp16=False,
max_grad_norm=0.3,
warmup_steps=25,
lr_scheduler_type="linear",
push_to_hub=True,
report_to="none",
gradient_checkpointing_kwargs={"use_reentrant": False},
dataset_kwargs={"skip_prepare_dataset": True},
remove_unused_columns = False,
label_names=["labels"],
)10. Creación de un recopilador de datos de visión y lenguaje para la formación
Para ajustar un modelo de visión-lenguaje como Ministral 3, necesitamos un recopilador de datos personalizado que pueda agrupar imágenes y texto tipo chat en un formato coherente.
El clasificador que aparece a continuación se encarga de: configurar el tokenizador y el token de relleno, aplicar la plantilla de chat, tokenizar el texto y las imágenes conjuntamente, aplicar una longitud máxima de secuencia segura y crear etiquetas en las que se ocultan los tokens de relleno e imagen (-100) para que no afecten a la pérdida.
from typing import Any, Dict, List
import torch
# --- One-time processor setup ---
tokenizer = processor.tokenizer
tokenizer.padding_side = "right"
# Ensure we have a pad token
if tokenizer.pad_token is None:
tokenizer.add_special_tokens({"pad_token": tokenizer.eos_token})
pad_token_id = tokenizer.pad_token_id
image_token_id = getattr(processor, "image_token_id", None)
# Hard cap for sequence length
MAX_SEQ_LEN = 2048
def collate_fn(examples: List[Dict[str, Any]]) -> Dict[str, torch.Tensor]:
"""
Collate function for VLM fine-tuning.
Each example must contain:
- "image": a PIL image
- "messages": chat-style conversation (list[dict])
"""
images = [ex["image"] for ex in examples]
conversations = [ex["messages"] for ex in examples]
# Build chat prompts for the whole batch
chat_texts = processor.apply_chat_template(
conversations,
add_generation_prompt=False,
tokenize=False,
)
# Tokenize + process images
batch = processor(
text=chat_texts,
images=images,
padding="longest",
truncation=True,
max_length=MAX_SEQ_LEN,
return_tensors="pt",
)
# Labels: same as input_ids but mask pad & image tokens
labels = batch["input_ids"].clone()
if pad_token_id is not None:
labels[labels == pad_token_id] = -100
if image_token_id is not None and image_token_id != pad_token_id:
labels[labels == image_token_id] = -100
batch["labels"] = labels
return batch11. Ajuste fino de Ministral 3 utilizando TRL
Con el adaptador LoRA, la configuración de entrenamiento y el recopilador de datos listos, ahora podemos conectar todo al SFTTrainer de trl.
Este entrenador gestiona todo el ciclo de ajuste supervisado para nuestro modelo de visión-lenguaje Ministral 3, utilizando nuestro conjunto de datos formateado, la configuración LoRA y la función de recopilación personalizada para lotes de imágenes y texto.
from trl import SFTTrainer
trainer = SFTTrainer(
model=model,
args=args,
train_dataset=formatted_data,
peft_config=peft_config,
processing_class=processor,
data_collator=collate_fn,
)Ejecuta el código siguiente para iniciar el proceso de entrenamiento.
trainer.train()Durante mi ejecución, el ajuste fino utilizó alrededor de 57 GB de VRAM, y un poco más durante los pasos de evaluación que se ejecutan cada pocas iteraciones. En la práctica, esto significa que puedes ajustar cómodamente esta configuración en hardware con menos de 70 GB de VRAM.
El entrenamiento completo duró alrededor de media hora, y la pérdida de entrenamiento disminuyó de forma constante con el tiempo, lo cual es una buena señal de que el modelo está aprendiendo del conjunto de datos.
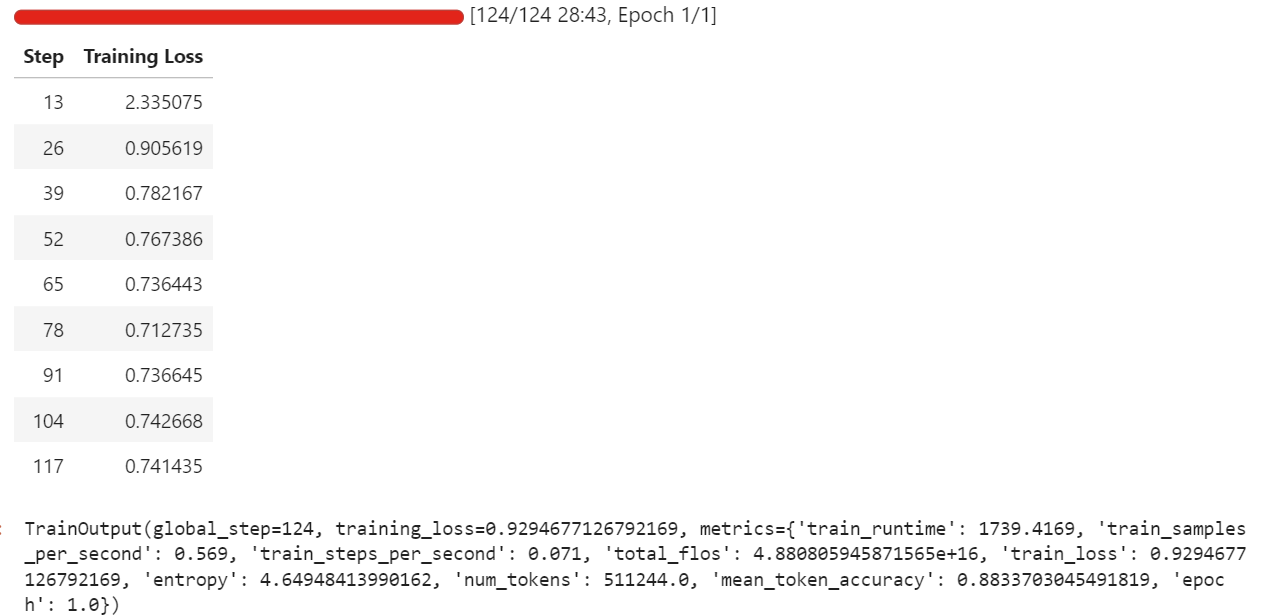
Una vez completado el entrenamiento, guardamos el modelo adaptado en Hugging Face Hub. Esto creará un nuevo repositorio y enviará todos los archivos necesarios para que puedas volver a cargar y ejecutar el modelo ajustado desde cualquier entorno.
trainer.save_model()Puedes ver el modelo en kingabzpro/ministral-3-fractura-de-hueso.
12. Evaluación del modelo Ministral 3 ajustado con precisión
Ahora cargamos el modelo y el procesador ajustados desde Hugging Face Hub. En segundo plano, esto descargará el adaptador LoRA guardado, cargará el punto de control base Ministral 3 y fusionará el adaptador con el modelo base para que esté listo para la inferencia.
import torch
from transformers import AutoProcessor, AutoModelForImageTextToText, BitsAndBytesConfig
model_id = "kingabzpro/ministral-3-bone-fracture"
model_kwargs = dict(
attn_implementation="eager",
dtype=torch.bfloat16,
device_map="auto",
)
model = AutoModelForImageTextToText.from_pretrained(model_id, **model_kwargs)
processor = AutoProcessor.from_pretrained(model_id)
processor.tokenizer.padding_side = "left"Para que la comparación sea justa, primero reutilizamos la misma imagen que probamos antes del ajuste fino (índice 200) y la ejecutamos a través del proceso de inferencia con nuestra indicación de clasificación estricta.
image = formatted_data[200]["image"]
messages = [
{"role": "user", "content": [
{"type": "text", "text": PROMPT},
{"type": "image"},
]}
]
input_text = processor.apply_chat_template(messages, add_generation_prompt = True)
inputs = processor(
image,
input_text,
add_special_tokens = False,
return_tensors = "pt",
).to(device="cuda", dtype=torch.bfloat16)
from transformers import TextStreamer
text_streamer = TextStreamer(processor, skip_prompt = True)
_ = model.generate(**inputs, streamer = text_streamer, max_new_tokens = 100)Hemos conseguido un resultado perfecto. No fue necesario ningún procesamiento posterior adicional. El modelo siguió las reglas indicadas y produjo directamente la clase correcta.
Comminuted fracture</s>A continuación, probamos otra muestra aleatoria del conjunto de datos para confirmar que este comportamiento no se limita a un solo ejemplo.
image = formatted_data[400]["image"]
messages = [
{"role": "user", "content": [
{"type": "text", "text": PROMPT},
{"type": "image"},
]}
]
input_text = processor.apply_chat_template(messages, add_generation_prompt = True)
inputs = processor(
image,
input_text,
add_special_tokens = False,
return_tensors = "pt",
).to(device="cuda", dtype=torch.bfloat16)
from transformers import TextStreamer
text_streamer = TextStreamer(processor, skip_prompt = True)
_ = model.generate(**inputs, streamer = text_streamer, max_new_tokens = 100)Resultado del modelo:
Greenstick fracture</s>Realidad sobre el terreno:
FRACTURE_CLASSES[formatted_data[400]["label"]]Salida:
'Greenstick fracture'Una vez más, la predicción es correcta y coincide plenamente con la etiqueta.
Con estas comprobaciones, podemos afirmar que nuestro modelo Ministral 3 14B se ha ajustado con éxito al conjunto de datos de radiografías de fracturas óseas y ahora es capaz de seguir las instrucciones y generar predicciones precisas de una sola clase para estos ejemplos.
Reflexiones finales
El ajuste fino del modelo Ministral 3 requiere paciencia y ajustes constantes. Realmente me llevó dos días completos comprender por qué el modelo no se estaba entrenando como esperaba y por qué, a pesar de ser un modelo relativamente pequeño, seguía provocando errores de memoria insuficiente en mi GPU.
La razón principal era simple, pero fácil de pasar por alto. No se trata solo de un modelo lingüístico. Es un modelo de lenguaje visual, lo que significa que lleva un codificador de imágenes que añade un uso adicional de memoria.
Las resoluciones de imagen más grandes provocan directamente un mayor consumo de VRAM. Reducir el tamaño de la imagen o empezar con lotes más pequeños hace que el proceso de entrenamiento sea mucho más estable.
Otro problema con el que me encontré fue el uso de la plantilla de chat predeterminada. Era demasiado largo y estructurado para este tipo de tarea. El modelo dedicaba más esfuerzo a ajustarse al formato de la plantilla que a aprender a leer las imágenes y producir una clasificación clara de las fracturas.
Una vez que simplifiqué la indicación y la convertí en una instrucción directa, el modelo finalmente comenzó a responder correctamente a la información visual, en lugar de dar cada respuesta como si fuera una conversación completa.
También supuse que, dado que el modelo tiene una gran capacidad de visión, naturalmente funcionaría bien en exploraciones médicas. Esa suposición me frenó. Llamarlo «MedGemma» en mi cabeza no era justo para el modelo ni para el proceso. No es un modelo médico especializado listo para usar, y esperar que se comportara como tal sin la formación adecuada fue un error por mi parte.
Al final, superé todos los obstáculos y convertí lo que aprendí en esta guía. El objetivo es ayudarte a evitar la misma confusión y obtener resultados significativos más rápidamente.
Con imágenes más pequeñas, indicaciones más sencillas y expectativas realistas, Ministral 3 se puede ajustar para comprender las radiografías de una manera mucho más fluida y fiable.
Si deseas adquirir más práctica con el ajuste fino, te recomiendo el curso curso Ajuste fino con Llama 3.

Soy un científico de datos certificado que disfruta creando aplicaciones de aprendizaje automático y escribiendo blogs sobre ciencia de datos. Actualmente me centro en la creación de contenidos, la edición y el trabajo con grandes modelos lingüísticos.
