Kurs
Selbstorganisierende Karten (SOMs) sind eine Art von künstlichen neuronalenNetzen, die für unbeaufsichtigte Lernaufgaben wie das Clustering verwendet werden. Bei einer Menge von Datenpunkten lernt das Modell, die Daten in Cluster einzuteilen. SOMs projizieren komplexe mehrdimensionale Daten auf ein niedrigeres (typischerweise 2-dimensionales) Gitter. Das erleichtert die Visualisierung. Daher werden SOMs auch zur Visualisierung von hochdimensionalen Daten verwendet.
In diesem Tutorium lernen wir die Kernkonzepte von SOMs, ihren Lernprozess und ihre typischen Anwendungsfälle kennen. Wir erklären, wie man ein SOM in Python mit der MiniSom-Bibliothek implementiert und wie man die Ergebnisse visuell darstellt. Zum Schluss besprechen wir wichtige Hyperparameter beim Training von SOM-Modellen und wie man sie fein abstimmt.
Selbstorganisierende Karten (SOM) verstehen
In diesem Abschnitt stellen wir die Kernkonzepte von SOMs, ihren Lernprozess und ihre Anwendungsfälle vor.
Kernkonzepte von SOMs
Selbstorganisierende Karten beinhalten eine Reihe von Konzepten, die wichtig sind, um sie zu verstehen.
Neuronen
Ein SOM ist im Wesentlichen ein Gitter aus Neuronen. Während des Trainings passt das Neuron, dessen Gewichtsvektor einem Eingabedatenpunkt am nächsten liegt, seine Gewichte und die Gewichte seiner Nachbarn an, um dem Eingabedatenpunkt noch näher zu kommen. Über viele Iterationen hinweg werden Gruppen von benachbarten Neuronen auf verwandte Datenpunkte abgebildet. Das Ergebnis ist die Clusterung des Eingabedatensatzes.
Rasterstruktur
Die Neuronen in einem SOM sind als Gitter organisiert. Benachbarte Neuronen werden auf ähnliche Datenpunkte abgebildet. Größere Datensätze brauchen ein größeres Raster. In der Regel ist dieses Raster zweidimensional. Die Gitterstruktur dient als niedrigdimensionaler Raum, auf den hochdimensionale Daten abgebildet werden. Das Raster hilft bei der Visualisierung von Datenmustern und Clustern.
Datenpunkte kartieren
Jeder Datenpunkt wird mithilfe einer Abstandsmetrik mit allen Neuronen verglichen. Das Neuron, dessen Gewichtsvektor dem Eingangsdatenpunkt am nächsten liegt, ist die Best Matching Unit (BMU) für diesen Datenpunkt .
Sobald die BMU identifiziert ist, werden die Gewichte der BMU und ihrer benachbarten Neuronen aktualisiert. Diese Aktualisierung bringt die BMU und die benachbarten Neuronen noch näher an den Eingangsdatenpunkt heran. Diese Zuordnung erhält die Topologie der Daten und stellt sicher, dass ähnliche Datenpunkte auf nahe gelegene Neuronen abgebildet werden.
Lernprozess von SOMs
Das SOM-Training gilt als unüberwacht, weil es nicht auf gelabelten Datensätzen basiert. Das Ziel des Trainings von SOMs ist es, die Gewichtsvektoren der Neuronen iterativ so anzupassen, dass ähnliche Datenpunkte auf nahe gelegene Neuronen abgebildet werden.
Wettbewerbsorientiertes Lernen
SOMs verwenden kompetitives Lernen (anstelle von Gradientenabstieg und Backpropagation). Neuronen konkurrieren darum, die Best Matching Unit (BMU) für jeden Eingangsdatenpunkt zu werden. Das Neuron, das einem Datenpunkt am nächsten liegt, wird als dessen BMU bestimmt. Die BMU und ihre benachbarten Neuronen werden aktualisiert, um ihren Abstand zum Datenpunkt weiter zu verringern. Benachbarte Neuronen bilden verwandte Datenpunkte ab. Dies führt zu einer Spezialisierung zwischen den Neuronen und einer Clusterung der Eingangsdatenpunkte.
Abstandsfunktionen
SOMs verwenden eine Distanzfunktion, um den Abstand zwischen Neuronen und Datenpunkten zu messen. Dieser Abstand wird verwendet, um die BMU eines jeden Datenpunktes zu bestimmen. MiniSom hat vier Distanzfunktionen, aus denen du wählen kannst:
- Euklidischer Abstand (dies ist die Standardeinstellung)
- Kosinusabstand
- Manhattan Entfernung
- Tschebyscheff-Abstand
Funktion in der Nachbarschaft
Nachdem die BMU eines Datenpunkts identifiziert wurde, werden die BMU und ihre benachbarten Neuronen in Richtung dieses Datenpunkts aktualisiert. Die Nachbarschaftsfunktion stellt sicher, dass die SOM die topologischen Beziehungen der Eingabedaten beibehält. Die Nachbarschaftsfunktion entscheidet:
- Welche Neuronen als in der Nachbarschaft der BMU liegend betrachtet werden
- Das Ausmaß, in dem die benachbarten Neuronen aktualisiert werden. Im Allgemeinen erhalten Neuronen, die näher an der BMU liegen, eine größere Anpassung als Neuronen, die weiter entfernt sind.
MiniSom verfügt über drei Nachbarschaftsfunktionen:
- Gauß (dies ist die Standardeinstellung).
- Blase
- Mexikanerhut
- Dreieck
Iterativer Prozess
Der Lernprozess in SOMs läuft über mehrere Iterationen ab. In jeder Iteration verarbeitet das SOM viele Eingangsdatenpunkte. Wir skizzieren den Lernprozess in den folgenden Schritten:
- Wenn das Training beginnt, werden die Gewichte aller Neuronen (zufällig) initialisiert.
- Bei einem eingegebenen Datenpunkt berechnet jedes Neuron seinen Abstand zum Eingang.
- Das Neuron mit dem kleinsten Abstand wird zur BMU erklärt.
- Die Gewichte der BMU und ihrer Nachbarn werden angepasst, um sich dem Eingangsvektor anzunähern. Das Ausmaß dieser Anpassung wird durch:
- Eine Nachbarschaftsfunktion: Neuronen, die weiter von der BMU entfernt sind, werden weniger aktualisiert als solche, die näher an der BMU liegen. Dadurch bleibt die topologische Struktur der Daten erhalten.
- Lernrate: Eine höhere Lernrate führt zu größeren Aktualisierungen.
Diese Schritte werden über viele Iterationen wiederholt, wobei viele Eingabedatenpunkte berücksichtigt werden. Die Gewichte werden schrittweise aktualisiert, sodass sich die Karte selbst organisiert und die Struktur der Daten erfasst.
Die Lernrate und der Nachbarschaftsradius nehmen in der Regel mit der Zeit ab, sodass die SOM die Gewichte allmählich fein abstimmen kann. Im weiteren Verlauf des Tutorials werden wir die Schritte des iterativen Trainingsprozesses mit Python-Code umsetzen.
Typische Anwendungsfälle von SOMs
Aufgrund ihrer einzigartigen Architektur haben SOMs mehrere typische Anwendungen im Bereich des maschinellen Lernens, der Analyse und der Visualisierung.
Clustering
SOMs werden verwendet, um hochdimensionale Daten in Clustern ähnlicher Datenpunkte zu gruppieren. Dies hilft dabei, inhärente Strukturen in den Daten zu erkennen. Jedes Neuron ordnet eng zusammenhängende Datenpunkte zu und ähnliche Datenpunkte werden denselben oder benachbarten Neuronen zugeordnet, wodurch verschiedene Cluster gebildet werden.
Im Marketing können SOMs zum Beispiel Kunden nach ihrem Kaufverhalten gruppieren. Dies würde es einer Marke ermöglichen, ihre Marketingstrategien auf verschiedene Kundensegmente (Cluster) zuzuschneiden.
Dimensionalitätsreduktion
SOMs können hochdimensionale Daten mit vielen Merkmalsvektoren auf ein Gitter mit weniger Dimensionen (typischerweise ein 2-dimensionales Gitter) abbilden. Da bei dieser Zuordnung die Beziehungen zwischen den Datenpunkten erhalten bleiben, erleichtert die reduzierte Dimensionalität die Visualisierung komplexer Datensätze ohne großen Informationsverlust.
SOMs erleichtern nicht nur Visualisierungs- und Analyseaufgaben, sondern können auch die Dimensionalität von Daten reduzieren, bevor andere maschinelle Lernalgorithmen angewendet werden. SOMs werden auch in der Bildverarbeitung eingesetzt, um die Anzahl der Merkmale zu reduzieren, indem Pixel oder Regionen mit ähnlichen Merkmalen zu Clustern zusammengefasst werden, wodurch Bilderkennungs- und Klassifizierungsaufgaben effizienter werden.
Erkennung von Anomalien
Während des Trainingsprozesses werden die meisten Datenpunkte bestimmten Neuronen zugeordnet. Anomalien (Datenpunkte, die sich keinem Cluster zuordnen lassen) werden in der Regel auf weit entfernte oder weniger bevölkerte Neuronen abgebildet. Diese Ausreißer passen nicht gut in die gelernten Cluster und werden Neuronen zugeordnet, die weit von der BMU der normalen Datenpunkte entfernt sind.
Wenn wir zum Beispiel Finanztransaktionen auf einem SOM-Gitter abbilden, können wir ungewöhnliche Aktivitäten (Ausreißer) erkennen, die auf potenziell betrügerische Aktivitäten hinweisen können.
Datenvisualisierung
Die Datenvisualisierung ist einer der häufigsten Anwendungsfälle von SOMs. Hochdimensionale Daten sind schwer zu visualisieren. SOMs erleichtern die Visualisierung, indem sie die Dimensionalität der Daten reduzieren und sie auf ein zweidimensionales Raster projizieren. Dies kann auch helfen, Cluster in den Daten und anomale Datenpunkte zu erkennen, die in den Originaldaten nur schwer zu entdecken wären.
Die Datenvisualisierung erleichtert zum Beispiel die Analyse von hochdimensionalen Bevölkerungsdaten. Wenn du die Daten auf einem 2D-Gitter abbildest, kannst du Gemeinsamkeiten und Unterschiede zwischen verschiedenen Bevölkerungsgruppen erkennen.
Nachdem wir die Kernkonzepte und Anwendungsfälle von SOMs besprochen haben, zeigen wir in den folgenden Abschnitten, wie man sie mit dem Python-Paket MiniSom implementiert .
Du kannst den kompletten Code auf diesem DataLab-Notebook aufrufen und ausführen.
Fähigkeiten im Bereich Machine Learning aufbauen
Einrichten der Umgebung für SOM
Bevor wir das SOM erstellen, müssen wir die Umgebung mit den notwendigen Paketen vorbereiten.
Installation der Python-Bibliotheken
Wir brauchen diese Pakete:
- MiniSom ist ein NumPy-basiertes Python-Tool, das SOMs erstellt und trainiert.
- NumPy wird verwendet, um auf mathematische Funktionen zuzugreifen, z. B. um Arrays aufzuteilen, eindeutige Werte zu erhalten usw.
matplotlibwird verwendet, um verschiedene Diagramme und Tabellen zur Visualisierung der Daten zu erstellen.- Das Paket
datasetsvonsklearnwird verwendet, um Datensätze zu importieren, auf die das SOM angewendet werden soll . - Das Paket
MinMaxScalervonsklearnnormalisiert den Datensatz .
Der folgende Codeschnipsel importiert diese Pakete:
from minisom import MiniSom
import numpy as np
import matplotlib.pyplot as plt
from sklearn import datasets
from sklearn.preprocessing import MinMaxScalerVorbereiten des Datensatzes
In diesem Lernprogramm verwenden wir MiniSom, um ein SOM zu erstellen und es dann auf dem kanonischen IRIS-Datensatzzu trainieren. Diesesaset besteht aus 3 Klassen von Schwertlilienpflanzen. Jede Klasse hat 50 Instanzen. Um die Daten vorzubereiten, gehen wir folgendermaßen vor:
- Importiere den Iris-Datensatz von
sklearn, - Extrahiere die Datenvektoren und die Zielskalare.
- Normalisiere die Datenvektoren. In diesem Lernprogramm verwenden wir den MinMaxScaler von scikit-learn.
- Erkläre eine Reihe von Etiketten für jede der drei Klassen von Iris-Pflanzen.
Der folgende Code setzt diese Schritte um:
dataset_iris = datasets.load_iris()
data_iris = dataset_iris.data
target_iris = dataset_iris.target
data_iris_normalized = MinMaxScaler().fit_transform(data_iris)
labels_iris = {1:'1', 2:'2', 3:'3'}
data = data_iris_normalized
target = target_irisImplementierung von Self-Organizing Maps (SOM) in Python
Um ein SOM in Python zu implementieren, definieren und initialisieren wir das Gitter, bevor wir es mit dem Datensatz trainieren. Wir können dann die trainierten Neuronen und den geclusterten Datensatz visualisieren.
Festlegen des SOM-Gitters
Wie bereits erklärt, ist ein SOM ein Gitter aus Neuronen. Mit MiniSom können wir 2-dimensionale Raster erstellen. Die X- und Y-Dimensionen des Rasters sind die Anzahl der Neuronen entlang jeder Achse. Um das SOM-Gitter zu definieren, müssen wir auch angeben:
- Die X- und Y-Abmessungen des Rasters
- Die Anzahl der Eingabevariablen - das ist die Anzahl der Datenzeilen.
Deklariere diese Parameter als Python-Konstanten:
SOM_X_AXIS_NODES = 8
SOM_Y_AXIS_NODES = 8
SOM_N_VARIABLES = data.shape[1]Der Beispielcode unten zeigt, wie du das Raster mit MiniSom deklarierst:
som = MiniSom(SOM_X_AXIS_NODES, SOM_Y_AXIS_NODES, SOM_N_VARIABLES)Die ersten beiden Parameter sind die Anzahl der Neuronen entlang der X- und Y-Achse und der dritte Parameter ist die Anzahl der Variablen.
Bei der Erstellung des SOM-Gitters geben wir weitere Parameter und Hyperparameter an. Wir werden diese später im Lernprogramm erklären. Deklariere diese Parameter zunächst wie unten gezeigt:
ALPHA = 0.5
DECAY_FUNC = 'linear_decay_to_zero'
SIGMA0 = 1.5
SIGMA_DECAY_FUNC = 'linear_decay_to_one'
NEIGHBORHOOD_FUNC = 'triangle'
DISTANCE_FUNC = 'euclidean'
TOPOLOGY = 'rectangular'
RANDOM_SEED = 123Erstelle ein SOM mit diesen Parametern:
som = MiniSom(
SOM_X_AXIS_NODES,
SOM_Y_AXIS_NODES,
SOM_N_VARIABLES,
sigma=SIGMA0,
learning_rate=ALPHA,
neighborhood_function=NEIGHBORHOOD_FUNC,
activation_distance=DISTANCE_FUNC,
topology=TOPOLOGY,
sigma_decay_function = SIGMA_DECAY_FUNC,
decay_function = DECAY_FUNC,
random_seed=RANDOM_SEED,
)Initialisierung der Neuronen
Der obige Befehl erstellt ein SOM mit zufälligen Gewichten für alle Neuronen. Die Initialisierung der Neuronen mit Gewichten aus den Daten (anstelle von Zufallszahlen) kann den Trainingsprozess effizienter machen.
Wenn du MiniSom verwendest, um eine selbstorganisierende Karte (SOM) zu erstellen, gibt es zwei Möglichkeiten, die Gewichte der Neuronen auf der Grundlage der Daten zu initialisieren:
- Zufällige Initialisierung: Die Anfangsgewichte der Neuronen werden zufällig aus den Eingabedaten gezogen. Wir tun dies, indem wir die Funktion
.random_weights_init()auf das SOM anwenden. - PCA-Initialisierung: Die Initialisierung der Hauptkomponentenanalyse (PCA) verwendetdie Hauptkomponenten dereingangsdaten, um die Gewichte zu initialisieren. Die Anfangsgewichte der Neuronen umfassen die ersten beiden Hauptkomponenten. Das führt oft zu einer schnelleren Konvergenz.
In diesem Leitfaden verwenden wir die PCA-Initialisierung. Um die PCA-Initialisierung auf die SOM-Gewichte anzuwenden, verwendest du die Funktion .pca_weights_init() wie unten gezeigt:
som.pca_weights_init(data)Training der SOM
Der Trainingsprozess aktualisiert die SOM-Gewichte, um den Abstand zwischen den Neuronen und den Datenpunkten zu minimieren.
Im Folgenden erklären wir den iterativen Ausbildungsprozess:
- Initialisierung: Die Gewichtsvektoren aller Neuronen werden initialisiert, normalerweise mit zufälligen Werten. Es ist auch möglich, die Gewichte durch Stichproben aus der Verteilung der Eingangsdaten zu initialisieren.
- Eingangsauswahl: Ein Eingangsvektor wird (zufällig) aus dem Trainingsdatensatz ausgewählt.
- BMU-Kennung: Das Neuron mit dem Gewichtsvektor, der dem Eingangsvektor am nächsten liegt, wird als BMU identifiziert.
- Update für die Nachbarschaft: Die BMU und ihre benachbarten Neuronen aktualisieren ihre Gewichtsvektoren. Die Lernrate und die Nachbarschaftsfunktion entscheiden, welche Neuronen aktualisiert werden und um wie viel. Bei dem Iterationsschritt twird der Eingangsvektor xder Gewichtsvektor des Neurons i als wi, die Lernrate (t)und die Nachbarschaftsfunktion hbi (diese Funktion quantifiziert das Ausmaß der Aktualisierung für das Neuron i angesichts des BMU-Neurons b), die Formel zur Aktualisierung der Gewichte für Neuron i wie folgt ausgedrückt:

- Abklingrate der Lernrate und Nachbarschaftsradius: Sowohl die Lernrate als auch der Nachbarschaftsradius nehmen mit der Zeit ab. In den früheren Iterationen nimmt der Trainingsprozess größere Anpassungen in einer größeren Nachbarschaft vor. Spätere Iterationen helfen bei der Feinabstimmung der Gewichte, indem sie kleinere Änderungen an den Gewichten benachbarter Neuronen vornehmen. So kann sich die Karte stabilisieren und konvergieren.
Um das SOM zu trainieren, präsentieren wir dem Modell die Eingabedaten. Dazu können wir zwischen zwei Ansätzen wählen:
- Ziehe Stichproben nach dem Zufallsprinzip aus den Eingabedaten. Die Funktion
.train_random()setzt diese Technik um . - Gehe nacheinander die Vektoren in den Eingabedaten durch. Dies geschieht mit der Funktion
.train_batch().
Diese Funktionen akzeptieren die Eingabedaten und die Anzahl der Iterationen als Parameter. In diesem Leitfaden verwenden wir die Funktion .train_random() . Deklariere die Anzahl der Iterationen als Konstante und übergebe sie an die Trainingsfunktion:
N_ITERATIONS = 5000
som.train_random(data, N_ITERATIONS, verbose=True) Nachdem das Skript ausgeführt und das Training abgeschlossen wurde, wird eine Meldung mit dem Quantisierungsfehler angezeigt:
quantization error: 0.05357240680504421 Der Quantisierungsfehler gibt an, wie viel Information verloren geht, wenn das SOM die Daten quantisiert (die Dimensionalität reduziert). Ein großer Quantisierungsfehler weist auf einen größeren Abstand zwischen den Neuronen und den Datenpunkten hin. Das bedeutet auch, dass das Clustering weniger zuverlässig ist.
SOM-Neuronen visualisieren
Wir haben jetzt ein trainiertes SOM-Modell. Um sie zu visualisieren, verwenden wir eine Distanzkarte (auch bekannt als U-Matrix). Die Distanzkarte zeigt die Neuronen des SOM als Gitter aus Zellen an. Die Farbe jeder Zelle steht für ihren Abstand zu den benachbarten Neuronen.
Die Distanzkarte ist ein Raster mit denselben Abmessungen wie das SOM. Jede Zelle in der Distanzkarte ist die normalisierte Summe der (euklidischen) Distanzen zwischen einem Neuron und seinen Nachbarn.
Rufe die Seite SOM distance map über die Funktion .distance_map() auf. Um die U-Matrix zu erstellen, gehen wir folgendermaßen vor:
- Verwende
pyplot, um eine Figur mit denselben Abmessungen wie das SOM zu erstellen. In diesem Beispiel sind die Abmessungen 8x8. - Zeichne die Entfernungskarte mit matplotlib und der Funktion
.pcolor(). In diesem Beispiel verwenden wirgist_yargals Farbschema. - Zeige den
colorbaran, einen Index, der verschiedene Farben verschiedenen Skalarwerten zuordnet. Da die Abstände in diesem Fall normalisiert sind, reichen die skalaren Abstandswerte von 0 bis 1.
Der folgende Code setzt diese Schritte um:
# create the grid
plt.figure(figsize=(8, 8))
#plot the distance map
plt.pcolor(som.distance_map().T, cmap='gist_yarg')
# show the color bar
plt.colorbar()
plt.show()In diesem Beispiel verwendet die U-Matrix ein monotones Farbschema. Sie kann mit Hilfe dieser Richtlinien verstanden werden:
- Hellere Schattierungen stehen für eng beieinander liegende Neuronen und dunklere Schattierungen für Neuronen, die weiter von anderen entfernt sind.
- Gruppen von helleren Schattierungen können als Cluster interpretiert werden. Dunkle Knoten zwischen den Clustern können als die Grenzen zwischen den Clustern interpretiert werden.
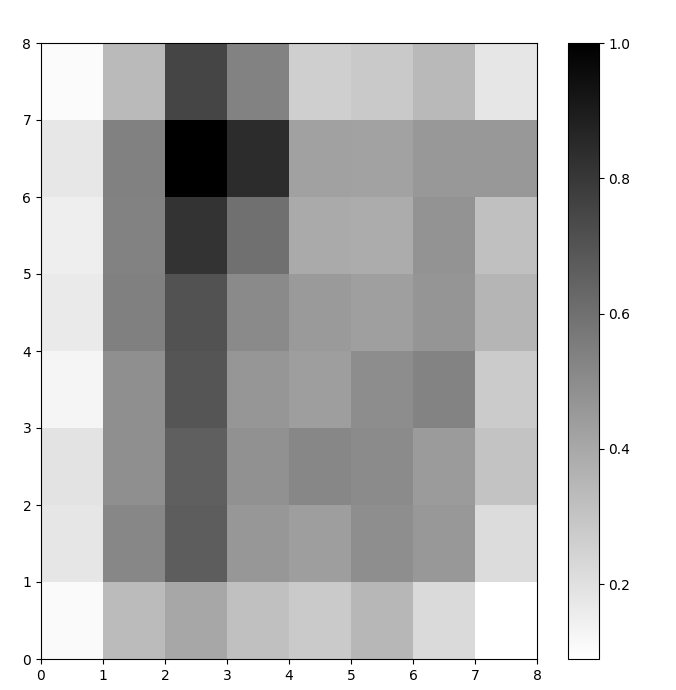
Abbildung 1: U-Matrix von SOM, trainiert mit dem Iris-Datensatz (Bild vom Autor)
Auswertung der SOM-Clustering-Ergebnisse
In der vorherigen Abbildung wurden die Neuronen des SOM grafisch dargestellt. In diesem Abschnitt zeigen wir, wie das SOM die Daten geclustert hat.
Identifizierung von Clustern
Wir überlagern die obige U-Matrix mit Markierungen, um zu kennzeichnen, welche Klasse der Iris-Pflanze jede Zelle (Neuron) repräsentiert. Um dies zu tun:
- Erstelle wie zuvor mit
pyploteine 8x8 Figur, zeichne die Entfernungskarte und zeige den Farbbalken . - Gib ein Array mit drei Matplotlib-Markernan, einen für jede Klasse der Iris-Pflanze.
- Gib ein Array mit drei Matplotlib-Farbcodes an, einen für jede Klasse der Iris-Pflanze.
- Zeichne iterativ das Gewinnerneuron für jeden Datenpunkt auf:
- Bestimme die (Koordinaten des) Gewinnerneurons für jeden Datenpunkt mithilfe der Funktion
.winner(). - Zeichne die Position jedes Gewinnerneurons in der Mitte jeder Zelle des Rasters ein.
w[0]undw[1]geben die X- bzw. Y-Koordinaten des Neurons an. Zu jeder Koordinate wird ein Wert von 0,5 hinzugefügt, damit sie in der Mitte der Zelle liegt.
Der folgende Code zeigt, wie man das macht:
# plot the distance map
plt.figure(figsize=(8, 8))
plt.pcolor(som.distance_map().T, cmap='gist_yarg')
plt.colorbar()
# create the markers and colors for each class
markers = ['o', 'x', '^']
colors = ['C0', 'C1', 'C2']
# plot the winning neuron for each data point
for count, datapoint in enumerate(data):
# get the winner
w = som.winner(datapoint)
# place a marker on the winning position for the sample data point
plt.plot(w[0]+.5, w[1]+.5, markers[target[count]-1], markerfacecolor='None',
markeredgecolor=colors[target[count]-1], markersize=12, markeredgewidth=2)
plt.show()Das resultierende Bild ist unten abgebildet:
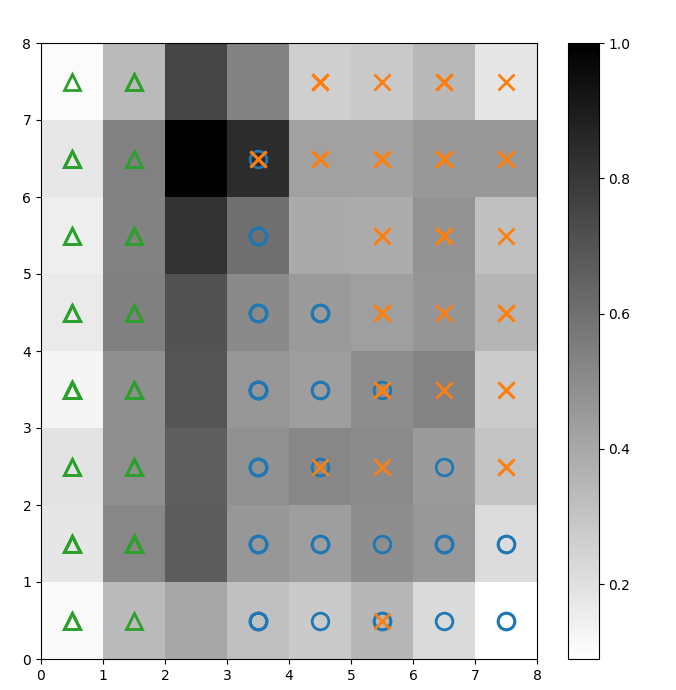
Abbildung 2: U-Matrix überlagert mit Klassenmarkern (Bild vom Autor)
In der Dokumentation des Iris-Datensatzes heißt es: "Eine Klasse ist linear von den anderen beiden trennbar; letztere sind nicht linear voneinander trennbar". In der U-Matrix oben werden diese drei Klassen durch drei Marker dargestellt - Dreieck, Kreis und Kreuz.
Beachte, dass es keine klare Grenze zwischen den blauen Kreisen und den orangefarbenen Kreuzen gibt. Außerdem überlagern sich in vielen Zellen zwei Klassen auf demselben Neuron. Das bedeutet, dass das Neuron von beiden Klassen gleich weit entfernt ist.
Visualisierung des Clustering-Ergebnisses
Ein SOM ist ein Clustermodell. Ähnliche Datenpunkte werden demselben Neuron zugeordnet. Datenpunkte der gleichen Klasse werden einem Cluster von benachbarten Neuronen zugeordnet. Wir stellen alle Datenpunkte auf dem SOM-Gitter dar, um das Clustering-Verhalten besser zu untersuchen.
Die folgenden Schritte beschreiben, wie du dieses Streudiagramm erstellst:
- Erhalte die X- und Y-Koordinaten des Gewinnerneurons für jeden Datenpunkt.
- Zeichne die Entfernungskarte, wie wir es für Abbildung 1.
- Verwende
plt.scatter(), um ein Streudiagramm mit allen gewinnenden Neuronen für jeden Datenpunkt zu erstellen. Füge zu jedem Punkt einen zufälligen Versatz hinzu, um Überschneidungen zwischen Datenpunkten innerhalb derselben Zelle zu vermeiden.
Wir setzen diese Schritte im folgenden Code um:
# get the X and Y coordinates of the winning neuron for each data pointw_x, w_y = zip(*[som.winner(d) for d in data])
w_x = np.array(w_x)
w_y = np.array(w_y)
# plot the distance map
plt.figure(figsize=(8, 8))
plt.pcolor(som.distance_map().T, cmap='gist_yarg', alpha=.2)
plt.colorbar()
# make a scatter plot of all the winning neurons for each data point
# add a random offset to each point to avoid overlaps
for c in np.unique(target):
idx_target = target==c
plt.scatter(w_x[idx_target]+.5+(np.random.rand(np.sum(idx_target))-.5)*.8,
w_y[idx_target]+.5+(np.random.rand(np.sum(idx_target))-.5)*.8,
s=50,
c=colors[c-1],
label=labels_iris[c+1]
)
plt.legend(loc='upper right')
plt.grid()
plt.show()Das folgende Diagramm zeigt die Streuung der Ergebnisse:
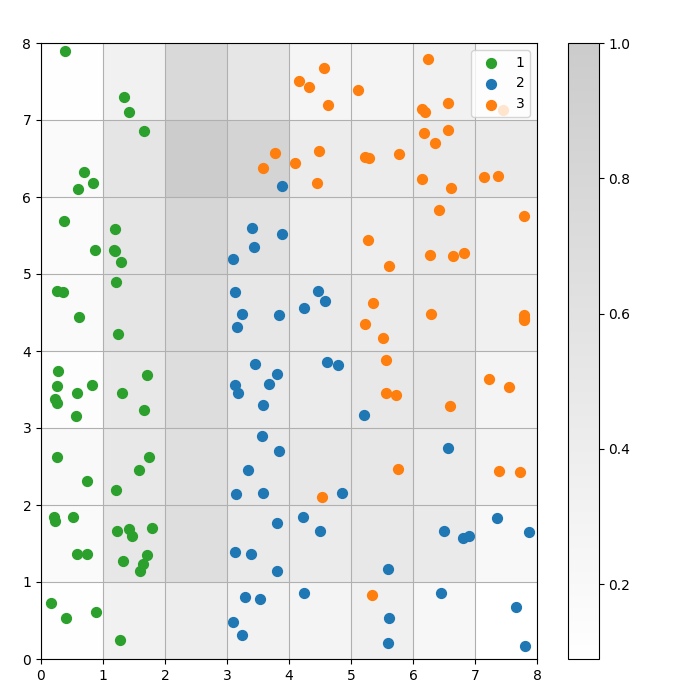 Abbildung 3: Streudiagramm von Datenpunkten innerhalb von Zellen (Bild vom Autor)
Abbildung 3: Streudiagramm von Datenpunkten innerhalb von Zellen (Bild vom Autor)
Im obigen Streudiagramm kannst du sehen, dass:
- Einige Zellen enthalten sowohl blaue als auch orangefarbene Punkte.
- Die grünen Punkte sind klar vom Rest der Daten getrennt, aber die blauen und orangefarbenen Punkte sind nicht sauber getrennt.
- Die obigen Beobachtungen stimmen mit der Tatsache überein, dass nur einer der drei Cluster im Iris-Datensatz eine klare Grenze hat.
- Unter Abbildung 1stimmen die dunklen Knoten zwischen den Clustern (die als die Grenzen zwischen den Clustern interpretiert werden können) mit den leeren Zellen im Streudiagramm überein.
Du kannst den kompletten Code auf diesem DataLab-Notebook aufrufen und ausführen.
Abstimmung des SOM-Modells
In den vorherigen Abschnitten wurde gezeigt, wie man ein SOM-Modell erstellt und trainiert und wie man die Ergebnisse visuell untersucht. In diesem Abschnitt erörtern wir, wie die Leistung von SOM-Modellen optimiert werden kann.
Wichtige Hyperparameter zum Abstimmen
Wie bei jedem maschinellen Lernmodell haben die Hyperparameter einen erheblichen Einfluss auf die Leistung des Modells.
Einige der Hyperparameter, die beim Training von SOMs wichtig sind, sind:
- Die Rastergröße bestimmt die Größe der Karte. Die Anzahl der Neuronen in einer Karte mit einer Gittergröße von AxB ist A*B.
- Die Lernrate bestimmt, wie stark die Gewichte in jeder Iteration verändert werden. Wir legen die anfängliche Lernrate fest, die mit der Zeit gemäß der Abklingfunktion abnimmt.
- Die Abklingfunktion bestimmt, wie stark die Lernrate in jeder nachfolgenden Iteration verringert wird.
- Die Nachbarschaftsfunktion ist eine mathematische Funktion, die angibt, welche Neuronen als Nachbarn der BMU betrachtet werden sollen.
- Die Standardabweichung gibt die Streuung der Nachbarschaftsfunktion an. Zum Beispiel hat eine Gaußsche Nachbarschaftsfunktion mit einer hohen Standardabweichung eine größere Nachbarschaft als die gleiche Funktion mit einer kleineren Standardabweichung. Wir legen die anfängliche Standardabweichung fest, die mit der Zeit gemäß der Sigma-Abklingfunktion abnimmt.
- Der sigma decay Funktion steuert, wie stark die Standardabweichung in jeder nachfolgenden Iteration verringert wird.
- Die Anzahl der Trainingswiederholungen bestimmt, wie oft die Gewichte aktualisiert werden. In jeder Trainingsiteration werden die Neuronengewichte einmal aktualisiert.
- Die Abstandsfunktion ist eine mathematische Funktion, die den Abstand zwischen Neuronen und Datenpunkten berechnet.
- Die Topologie bestimmt das Layout der Gitterstruktur. Die Neuronen im Gitter können in einem rechteckigen oder sechseckigen Muster angeordnet sein.
Im nächsten Abschnitt besprechen wir Richtlinien für die Festlegung der Werte dieser Hyperparameter.
Auswirkungen der Abstimmung der Hyperparameter
Die Hyperparameterwerte sollten auf der Grundlage des Modells und des Datensatzes festgelegt werden. Bis zu einem gewissen Grad ist die Bestimmung dieser Werte ein Prozess von Versuch und Irrtum. In diesem Abschnitt geben wir Richtlinien für die Abstimmung der einzelnen Hyperparameter an. Neben jedem Hyperparameter stehen (in Klammern) die jeweiligen Python-Konstanten, die im Beispielcode verwendet werden.
- Rastergröße (
SOM_X_AXIS_NODESundSOM_X_AXIS_NODES): Die Größe des Rasters hängt von der Größe des Datensatzes ab. Als Faustregel gilt, dass das Raster bei einem Datensatz der Größe N ungefähr 5*sqrt(N) Neuronen enthalten sollte. Wenn der Datensatz zum Beispiel 150 Stichproben hat, sollte das Raster 5*sqrt(150) = ca. 61 Neuronen enthalten. In diesem Lernprogramm hat der Iris-Datensatz 150 Zeilen und wir verwenden ein 8x8-Gitter. - Anfängliche Lernrate (
ALPHA): Eine höhere Rate beschleunigt die Konvergenz, während niedrigere Raten für feinere Anpassungen nach frühen Iterationen verwendet werden. Die anfängliche Lernrate sollte groß genug sein, um eine schnelle Anpassung zu ermöglichen, aber nicht so groß, dass sie über die optimalen Gewichtswerte hinausgeht. In diesem Artikel ist die anfängliche Lernrate 0,5. - Anfängliche Standardabweichung (
SIGMA0): Sie bestimmt die anfängliche Größe oder Ausbreitung des Viertels. Ein größerer Wert berücksichtigt mehr globale Muster. In diesem Beispiel gehen wir von einer Standardabweichung von 1,5 aus. - Für die Zerfallsrate (
DECAY_FUNC) und die Sigma-Zerfallsrate (SIGMA_DECAY_FUNC) können wir eine von drei Arten von Zerfallsfunktionen wählen : - Inverser Zerfall: Diese Funktion ist geeignet, wenn die Daten sowohl globale als auch lokale Muster aufweisen. In solchen Fällen brauchen wir eine längere Phase des breiten Lernens, bevor wir uns auf lokale Muster konzentrieren.
- Linearer Zerfall: Das ist gut für Datensätze, bei denen wir eine stetige und gleichmäßige Verringerung der Nachbarschaftsgröße oder Lernrate wünschen. Das ist nützlich, wenn die Daten nicht viel Feinschliff brauchen.
- Asymptotischer Zerfall: Diese Funktion ist nützlich, wenn die Daten komplex und hochdimensional sind. In solchen Fällen ist es besser, mehr Zeit auf die globale Erkundung zu verwenden, bevor man schrittweise zu feineren Details übergeht.
- Nachbarschaftsfunktion (
NEIGHBORHOOD_FUNC): Die Standardauswahl für die Nachbarschaftsfunktion ist die Gauß-Funktion. Andere Funktionen, wie unten erklärt, werden ebenfalls verwendet. - Gaussian (Standard): Dies ist eine glockenförmige Kurve. Das Ausmaß, in dem ein Neuron aktualisiert wird, nimmt mit zunehmendem Abstand zum Gewinnerneuron gleichmäßig ab. Sie sorgt für einen sanften und kontinuierlichen Übergang und bewahrt die Topologie der Daten. Aufgrund seines stabilen und vorhersehbaren Verhaltens ist es für die meisten allgemeinen Zwecke geeignet.
- Bubble: Diese Funktion erstellt eine Nachbarschaft mit fester Breite. Alle Neuronen innerhalb dieser Nachbarschaft werden gleichermaßen aktualisiert, und Neuronen außerhalb dieser Nachbarschaft werden (für einen bestimmten Datenpunkt) nicht aktualisiert. Sie ist rechnerisch günstiger und einfacher zu implementieren. Sie ist nützlich für kleinere Karten, bei denen scharfe Nachbarschaftsgrenzen eine effektive Clusterbildung nicht gefährden.
- Mexikanischer Hut: Er hat einen zentralen positiven Bereich, der von einem negativen Bereich umgeben ist. Neuronen, die sich in der Nähe der BMU befinden, werden aktualisiert, um sich dem Datenpunkt zu nähern, und Neuronen, die weiter entfernt sind, werden aktualisiert, um sich vom Datenpunkt zu entfernen. Diese Technik verstärkt den Kontrast und schärft die Merkmale auf der Karte. Da sie unterschiedliche Cluster hervorhebt, eignet sie sich gut für Aufgaben der Mustererkennung, bei denen eine klare Trennung von Clustern erwünscht ist.
- Dreieck: Diese Funktion definiert die Größe der Nachbarschaft als ein Dreieck, wobei das BMU den größten Einfluss hat. Sie nimmt linear mit der Entfernung von der BMU ab. Sie wird für das Clustern von Daten mit allmählichen Übergängen zwischen Clustern oder Merkmalen verwendet, z. B. bei Bild-, Sprach- oder Zeitreihendaten, bei denen benachbarte Datenpunkte ähnliche Merkmale aufweisen sollen.
- Distanzfunktion (
DISTANCE_FUNC): Um den Abstand zwischen Neuronen und Datenpunkten zu messen, können wir zwischen 4 Methoden wählen: - Euklidischer Abstand (Standardeinstellung): Nützlich, wenn die Daten kontinuierlich sind und wir eine geradlinige Entfernung messen wollen. Sie eignet sich für die meisten allgemeinen Aufgaben, vor allem wenn die Datenpunkte gleichmäßig verteilt sind und in einem räumlichen Zusammenhang stehen.
- Kosinusabstand: Eine gute Wahl für Text oder hochdimensionale spärliche Daten, bei denen der Winkel zwischen Vektoren wichtiger ist als der Betrag. Sie ist nützlich, um die Richtungsabhängigkeit von Daten zu vergleichen.
- Manhattan Entfernung: Ideal, wenn die Datenpunkte in einem Gitternetz liegen (z. B. Häuserblocks). Diese Methode ist weniger anfällig für Ausreißer als der euklidische Abstand.
- Tschebyscheff-Abstand: Geeignet für Situationen, in denen die Bewegung in jede Richtung erfolgen kann (z. B. bei Schachbrettabständen). Sie ist nützlich für diskrete Räume, in denen wir die maximale Achsendifferenz priorisieren wollen.
- Topologie (
TOPOLOGY): In einem Gitter können die Neuronen in einer sechseckigen oder rechteckigen Struktur angeordnet sein: - Rechteckig (Standard): Jedes Neuron hat 4 direkte Nachbarn. Das ist die richtige Wahl, wenn die Daten keinen klaren räumlichen Bezug haben. Sie ist auch rechnerisch einfacher.
- Hexagonal: Jedes Neuron hat 6 Nachbarn. Dies ist die bevorzugte Option, wenn die Daten räumliche Beziehungen aufweisen, die mit einem sechseckigen Gitter besser dargestellt werden können. Dies ist bei kreisförmigen oder winkelförmigen Datenverteilungen der Fall.
- Anzahl der Trainingswiederholungen (
N_ITERATIONS): Im Prinzip führen längere Trainingszeiten zu geringeren Fehlern und einer besseren Anpassung der Gewichte an die Eingabedaten. Die Leistung des Modells steigt jedoch asymptotisch mit der Anzahl der Iterationen. Nach einer bestimmten Anzahl von Iterationen ist die Leistungssteigerung durch nachfolgende Interaktionen also nur noch marginal. Um die richtige Anzahl von Iterationen zu bestimmen, musst du ein wenig experimentieren. In diesem Lernprogramm trainieren wir das Modell über 5000 Iterationen.
Um die richtige Konfiguration der Hyperparameter zu ermitteln, empfehlen wir, mit verschiedenen Optionen auf einer kleineren Teilmenge der Daten zu experimentieren.
Fazit
Selbstorganisierende Karten sind ein robustes Werkzeug für unüberwachtes Lernen. Sie werden für das Clustering, die Dimensionalitätsreduktion, die Erkennung von Anomalien und die Datenvisualisierung verwendet. Da sie die topologischen Eigenschaften von hochdimensionalen Daten bewahren und sie auf einem niedrigdimensionalen Raster darstellen, erleichtern SOMs die Visualisierung und Interpretation komplexer Datensätze.
In diesem Tutorium wurden die grundlegenden Prinzipien von SOMs besprochen und gezeigt, wie man ein SOM mit der MiniSom Python-Bibliothek implementiert. Außerdem wurde gezeigt, wie die Ergebnisse visuell analysiert werden können, und es wurden die wichtigen Hyperparameter erklärt, die zum Trainieren von SOMs und zur Feinabstimmung ihrer Leistung verwendet werden.
Werde ein ML-Wissenschaftler
FAQs
Beinhaltet das SOM-Training Backpropagation?
Ein SOM ist ein künstliches neuronales Netz, aber sein Training basiert nicht auf einer Fehlerkorrektur. Es verwendet also keine Backpropagation mit Gradientenabstieg. Stattdessen werden SOMs durch kompetitives Lernen trainiert.
Wozu braucht man die Abklingfunktionen?
Schon früh im Trainingsprozess muss das Modell die globale Landschaft der Daten erkunden. Die Lernrate muss also hoch sein und die Nachbarschaftsfunktion muss breit gestreut sein. Spätere Trainingsiterationen nehmen feinere Anpassungen der Gewichte auf der Grundlage der benachbarten Neuronen vor. Mit fortschreitendem Training reduzieren die Abklingfunktionen die Lernrate und den Nachbarschaftsradius.
Was ist der Unterschied zwischen der standardmäßigen (zufälligen) Initialisierung und der zufälligen Initialisierung auf der Grundlage der Eingabedaten?
In der Standardinitialisierung werden die Gewichte mit beliebigen Zufallszahlen initialisiert.
Bei der zufälligen Initialisierung auf Basis der Eingangsdaten werden die Gewichte mit zufällig ausgewählten Stichproben aus den Eingangsdaten initialisiert.
Können SOMs nicht-lineare Beziehungen in den Daten erfassen?
Ja, SOMs erfassen nicht-lineare Muster in den Daten, indem sie verschiedene Nachbarschaftsfunktionen verwenden. Ein einziger Datenpunkt kann die Werte vieler Neuronen (in der Nachbarschaft) beeinflussen. So können SOMs komplexe Beziehungen in den Daten erfassen.
Was ist der Unterschied zwischen SOM und traditionellen Clustermethoden?
SOMs bilden hochdimensionale Daten auf einem niedrigdimensionalen Gitter ab, wobei die räumlichen Beziehungen erhalten bleiben. Traditionelle Clustering-Verfahren wie K-Means hingegen erstellen Gruppen auf der Grundlage von Abstandsmetriken, ohne notwendigerweise die Datentopologie oder räumliche Beziehungen zu berücksichtigen.
In der Datenanalyse beziehen sich topologische oder räumliche Beziehungen darauf, wie die Datenpunkte in einem mehrdimensionalen Raum zueinander angeordnet sind.
Arun ist ein ehemaliger Startup-Gründer, der Spaß daran hat, neue Dinge zu entwickeln. Derzeit erforscht er die technischen und mathematischen Grundlagen der Künstlichen Intelligenz. Er liebt es, sein Wissen mit anderen zu teilen, also schreibt er darüber.
Neben dem DataCamp kannst du seine Veröffentlichungen auf Medium, Airbyte und Vultr lesen.
