Cours
Le Segment Anything Model 2 (SAM 2) de Meta est la dernière innovation en matière de technologie de segmentation. Il s'agit du premier modèle unifié de Meta capable de segmenter des objets dans des images et des vidéos en temps réel.
Mais pourquoi affiner SAM 2 s'il peut déjà tout segmenter ?
Bien que SAM 2 soit puissant, ses performances sur des tâches rares ou spécifiques à un domaine ne sont pas toujours à la hauteur des attentes. Le réglage fin vous permet d'adapter SAM2 à vos besoins spécifiques, en améliorant sa précision et son efficacité pour votre cas d'utilisation particulier.
Dans cet article, je vous guiderai pas à pas dans le processus de mise au point de SAM 2.
Développer des applications d'IA
Qu'est-ce que SAM2 ?
SAM2 est un modèle de base développé par Meta pour la segmentation visuelle rapide dans les images et les vidéos. Contrairement à son prédécesseur, SAMqui se concentrait principalement sur les images statiques, SAM2 est conçu pour traiter également les complexités de la segmentation vidéo.
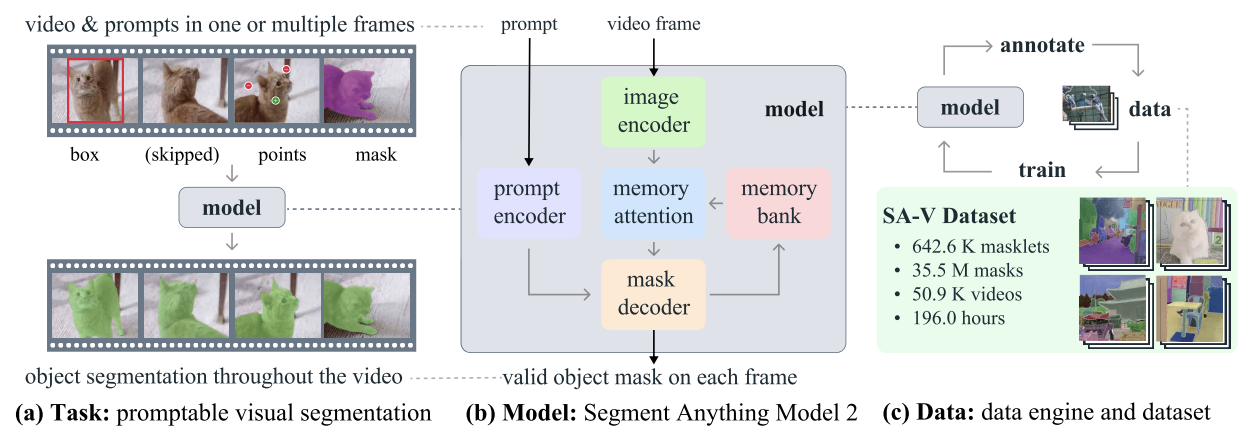
SAM2 - Tâche, modèle et données (Source : Ravi et al, 2024)
Il utilise une architecture de transformateur avec une mémoire en continu, permettant un traitement vidéo en temps réel. L'entraînement de SAM 2 a porté sur un ensemble de données vaste et varié comprenant le nouvel ensemble de données SA-V, qui comprend plus de 600 000 annotations de masques couvrant 51 000 vidéos.
Son moteur de données, qui permet la collecte interactive de données et l'amélioration du modèle, donne au modèle la capacité de segmenter tout ce qui est possible. Ce moteur permet à SAM 2 d'apprendre et de s'adapter en permanence, ce qui le rend plus efficace pour traiter des données nouvelles et difficiles. Toutefois, pour les tâches spécifiques à un domaine ou les objets rares, un réglage fin est essentiel pour obtenir des performances optimales.
Pourquoi affiner SAM2 ?
Dans le contexte de SAM 2, le réglage fin est le processus d'entraînement supplémentaire du modèle SAM 2 pré-entraîné sur un ensemble de données spécifique afin d'améliorer ses performances pour une tâche ou un domaine particulier. Bien que SAM 2 soit un outil puissant formé sur un ensemble de données large et diversifié, sa nature polyvalente ne permet pas toujours d'obtenir des résultats optimaux pour des tâches spécialisées ou rares.
Par exemple, si vous travaillez sur un projet d'imagerie médicale projet d'imagerie médicale qui nécessite l'identification de types de tumeurs spécifiques, les performances du modèle risquent d'être insuffisantes en raison de sa formation généralisée.
Le processus de mise au point
Le réglage fin de SAM 2 répond à cette limitation en vous permettant d'adapter le modèle à votre ensemble de données spécifique. Ce processus permet d'améliorer la précision du modèle et le rend plus efficace pour votre cas d'utilisation unique.
Voici les principaux avantages de la mise au point de SAM 2 :
- Amélioration de la précision: En affinant le modèle sur votre ensemble de données spécifique, vous pouvez améliorer de manière significative sa précision, garantissant ainsi de meilleures performances dans votre application ciblée.
- Segmentation spécialisée: Le réglage fin permet au modèle de devenir expert dans la segmentation de types d'objets spécifiques, de styles visuels ou d'environnements pertinents pour votre projet, fournissant ainsi des résultats sur mesure qu'un modèle polyvalent ne pourrait pas atteindre.
- Efficacité: Le réglage fin est souvent plus efficace que la formation d'un modèle à partir de zéro. Elle nécessite généralement moins de données et de temps, ce qui en fait une solution pratique pour adapter rapidement le modèle à des tâches nouvelles ou spécialisées.
Pour commencer à affiner les réglages de SAM 2 : Conditions préalables
Pour commencer à peaufiner SAM 2, vous devez disposer des conditions préalables suivantes :
- Accès au modèle SAM 2 et à la base de code: Avoir accès au modèle SAM 2 et à sa base de code. Vous pouvez télécharger le modèle SAM 2 pré-entraîné à partir du dépôt GitHub de le dépôt GitHub de Meta.
- Un ensemble de données approprié: Vous aurez besoin d'un ensemble de données comprenant des masques de segmentation de vérité terrain. Pour ce tutoriel, nous utiliserons le jeu de données Jeu de données de segmentation de la tomodensitométrie thoraciqueque vous pouvez télécharger et préparer pour l'entraînement.
- Ressources informatiques: Le réglage fin de SAM 2 nécessite un matériel doté d'une puissance de calcul suffisante. Les GPU sont fortement recommandés pour garantir un processus efficace et gérable, en particulier lorsque vous travaillez avec de grands ensembles de données ou des modèles complexes. Dans cet exemple, un GPU A100 sur Google Colab est utilisé.
Logiciels et autres exigences :
- Python 3.11 ou plus
- PyTorch
- OpenCV: Installez-le en utilisant !pip install opencv-python
Préparation de l'ensemble de données pour le réglage fin SAM 2
La qualité de votre jeu de données est cruciale pour affiner le modèle SAM 2. Des images ou des vidéos annotées de haute qualité avec des masques de segmentation précis sont essentiels pour obtenir des performances optimales. Des annotations précises permettent au modèle d'apprendre les caractéristiques correctes, ce qui améliore la précision de la segmentation et la robustesse des applications dans le monde réel.
1. Acquisition de données
La première étape consiste à acquérir l'ensemble de données, qui constitue l'épine dorsale du processus de formation. Nos données proviennent de Kaggleune plateforme fiable qui fournit un large éventail d'ensembles de données. À l'aide de l'API Kaggle, nous avons téléchargé les données dans le format requis, en veillant à ce que les images et les masques de segmentation correspondants soient facilement disponibles pour un traitement ultérieur.
2. Extraction et nettoyage des données
Après avoir téléchargé les ensembles de données, nous avons procédé aux étapes suivantes :
- Dézipper et nettoyer : Extrayez les données des fichiers zip téléchargés et supprimez les fichiers inutiles pour économiser de l'espace disque.
- Extraction de l'ID : Des identifiants uniques (ID) pour les images et les masques sont extraits afin d'assurer une correspondance correcte entre eux pendant la formation.
- Suppression des fichiers inutiles : Supprimez tous les fichiers bruyants ou non pertinents, tels que certaines images présentant des problèmes connus, afin de préserver l'intégrité de l'ensemble de données.
3. Conversion en formats utilisables
Le modèle SAM2 exigeant des données dans des formats spécifiques, nous avons converti les données comme suit :
- DICOM vers NumPy: Les images DICOM ont été lues et stockées sous forme de tableaux NumPy, qui ont ensuite été redimensionnés à une dimension standard de 512x512 pixels.
- NRRD à NumPy pour les masques : De même, les fichiers NRRD contenant des masques pour les poumons, le cœur et la trachée ont été traités et enregistrés sous forme de tableaux NumPy. Ces masques ont ensuite été remodelés pour correspondre aux images correspondantes.
- Conversion en JPG/PNG : Pour une meilleure visualisation et compatibilité, les tableaux NumPy ont été convertis aux formats JPG/PNG. Cette étape a consisté à normaliser les valeurs d'intensité de l'image et à s'assurer que les masques étaient correctement orientés.
4. Sauvegarde et organisation des données
Les images et les masques traités sont ensuite organisés dans des dossiers respectifs afin de faciliter l'accès au processus d'affinage. En outre, les chemins d'accès à ces images et masques sont inscrits dans un fichier CSV (train.csv ) pour faciliter le chargement des données pendant la formation.
5. Visualisation et validation
La dernière étape a consisté à valider l'ensemble des données afin d'en garantir l'exactitude :
- Visualisation: Nous avons visualisé les paires image-masque en superposant les masques aux images. Cela nous a permis de vérifier l'alignement et la précision des masques.
- Inspection: En inspectant quelques échantillons, nous avons pu confirmer que l'ensemble de données était correctement préparé et prêt à être utilisé pour le réglage fin.
Voici est un carnet de notes rapide qui vous guidera à travers le code de création d'un jeu de données. Vous pouvez soit suivre ce parcours de création de données, soit utiliser directement un jeu de données disponible en ligne dans le même format que celui mentionné dans les prérequis.
Mise au point de SAM2
Le segment Tout Le modèle 2 contient plusieurs composants, mais pour un réglage fin plus rapide, il suffit d'entraîner uniquement les composants légers, tels que le décodeur de masque et le codeur d'invite, plutôt que l'ensemble du modèle. Les étapes pour affiner ce modèle sont les suivantes :
Étape 1 : Installer SAM-2
Pour commencer le processus de mise au point, nous devons installer la bibliothèque SAM-2, qui est essentielle pour le modèle Segment Anything (SAM2). Ce modèle est conçu pour traiter efficacement diverses tâches de segmentation. L'installation consiste à cloner le dépôt SAM-2 depuis GitHub et à installer les dépendances nécessaires.
!git clone https://github.com/facebookresearch/segment-anything-2
%cd /content/segment-anything-2
!pip install -q -e .Cet extrait de code garantit que la bibliothèque SAM2 est correctement installée et prête à être utilisée dans notre flux de travail de réglage fin.
Étape 2 : Téléchargez l'ensemble des données
Une fois la bibliothèque SAM-2 installée, l'étape suivante consiste à acquérir le jeu de données que nous utiliserons pour le réglage fin. Nous utilisons un ensemble de données disponible sur Kaggle, en particulier un ensemble de données de segmentation de tomodensitométrie thoracique contenant des images et des masques des poumons, du cœur et de la trachée.
L'ensemble de données contient :
- images.zip: Images au format RVB
- masks.zip: Masques de segmentation au format RVB
- train.csv: Fichier CSV avec les noms des images
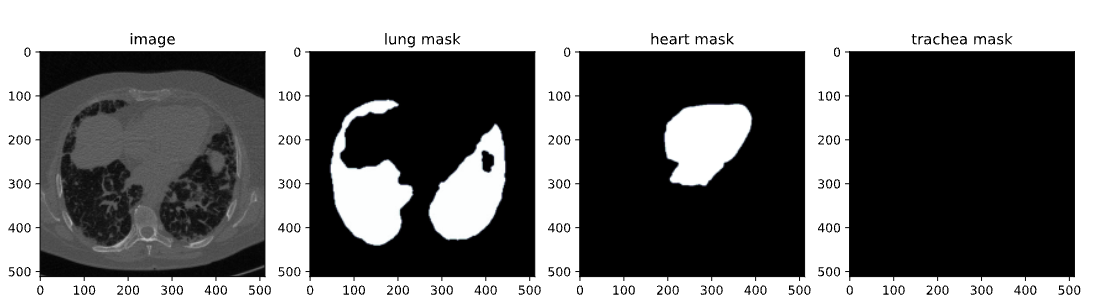
Image de l'ensemble de données du scanner
Dans ce blog, nous n'utiliserons que des images et des masques de poumons pour la segmentation. L'API Kaggle nous permet de télécharger des ensembles de données directement dans notre environnement. Nous commençons par télécharger le fichier kaggle.json à partir de Kaggle pour accéder facilement à n'importe quel ensemble de données.
Pour obtenir kaggle.json, allez dans l'onglet Paramètres sous votre profil d'utilisateur et sélectionnez Créer un nouveau jeton. Cela déclenchera le téléchargement Kaggle. json, un fichier contenant vos identifiants API.
# get dataset from Kaggle
from google.colab import files
files.upload() # This will prompt you to upload the kaggle.json file
!mkdir -p ~/.kaggle
!mv kaggle.json ~/.kaggle/
!chmod 600 ~/.kaggle/kaggle.json
!kaggle datasets download -d polomarco/chest-ct-segmentationDécompressez les données :
!unzip chest-ct-segmentation.zip -d chest-ct-segmentationL'ensemble de données étant prêt, commençons le processus d'affinage. Comme je l'ai déjà mentionné, l'essentiel est de ne régler que les composants légers de SAM2, tels que le décodeur de masque et l'encodeur d'invite, plutôt que l'ensemble du modèle. Cette approche est plus efficace et nécessite moins de ressources.
Étape 3 : Télécharger les points de contrôle SAM-2
Pour le processus de réglage fin, nous devons commencer par des poids de modèle SAM2 pré-entraînés. Ces poids, appelés "points de contrôle", constituent le point de départ de la formation ultérieure. Les points de contrôle ont été formés sur un large éventail d'images et, en les affinant sur notre ensemble de données spécifique, nous pouvons obtenir de meilleures performances sur nos tâches cibles.
!wget -O sam2_hiera_tiny.pt "https://dl.fbaipublicfiles.com/segment_anything_2/072824/sam2_hiera_tiny.pt"
!wget -O sam2_hiera_small.pt "https://dl.fbaipublicfiles.com/segment_anything_2/072824/sam2_hiera_small.pt"
!wget -O sam2_hiera_base_plus.pt "https://dl.fbaipublicfiles.com/segment_anything_2/072824/sam2_hiera_base_plus.pt"
!wget -O sam2_hiera_large.pt "https://dl.fbaipublicfiles.com/segment_anything_2/072824/sam2_hiera_large.pt"Au cours de cette étape, nous téléchargeons différents points de contrôle SAM-2 correspondant à différentes tailles de modèles (par exemple, tiny, small, base_plus, large). Le choix du point de contrôle peut être ajusté en fonction des ressources informatiques disponibles et de la tâche spécifique à accomplir.
Étape 4 : Préparation des données
Une fois l'ensemble de données téléchargé, l'étape suivante consiste à le préparer pour la formation. Il s'agit de diviser l'ensemble de données en ensembles de formation et de test et de créer des structures de données qui peuvent être introduites dans le modèle SAM 2 lors de l'ajustement.
%cd /content/segment-anything-2
import os
import pandas as pd
import cv2
import torch
import torch.nn.utils
import numpy as np
import matplotlib.pyplot as plt
import matplotlib.colors as mcolors
from sklearn.model_selection import train_test_split
from sam2.build_sam import build_sam2
from sam2.sam2_image_predictor import SAM2ImagePredictor
# Path to the chest-ct-segmentation dataset folder
data_dir = "/content/segment-anything-2/chest-ct-segmentation"
images_dir = os.path.join(data_dir, "images/images")
masks_dir = os.path.join(data_dir, "masks/masks")
# Load the train.csv file
train_df = pd.read_csv(os.path.join(data_dir, "train.csv"))
# Split the data into two halves: one for training and one for testing
train_df, test_df = train_test_split(train_df, test_size=0.2, random_state=42)
# Prepare the training data list
train_data = []
for index, row in train_df.iterrows():
image_name = row['ImageId']
mask_name = row['MaskId']
# Append image and corresponding mask paths
train_data.append({
"image": os.path.join(images_dir, image_name),
"annotation": os.path.join(masks_dir, mask_name)
})
# Prepare the testing data list (if needed for inference or evaluation later)
test_data = []
for index, row in test_df.iterrows():
image_name = row['ImageId']
mask_name = row['MaskId']
# Append image and corresponding mask paths
test_data.append({
"image": os.path.join(images_dir, image_name),
"annotation": os.path.join(masks_dir, mask_name)
})
Nous avons divisé l'ensemble de données en un ensemble de formation (80 %) et un ensemble de test (20 %) afin de pouvoir évaluer les performances du modèle après la formation. Les données d'entraînement seront utilisées pour affiner le modèle SAM 2, tandis que les données de test seront utilisées pour l'inférence et l'évaluation.
Après avoir divisé votre ensemble de données en ensembles de formation et de test, l'étape suivante consiste à créer des masques binaires, à sélectionner des points clés au sein de ces masques et à visualiser ces éléments pour s'assurer que les données sont correctement traitées.
1. Lecture et redimensionnement des images : Le processus commence par la sélection aléatoire d'une image et de son masque correspondant dans l'ensemble de données. L'image est convertie du format BGR au format RGB, qui est le format de couleur standard pour la plupart des modèles d'apprentissage profond. L'annotation correspondante (masque) est lue en niveaux de gris. Ensuite, l'image et le masque d'annotation sont redimensionnés à une dimension maximale de 1024 pixels, en conservant le rapport hauteur/largeur pour garantir que les données correspondent aux exigences d'entrée du modèle et réduisent la charge de calcul.
def read_batch(data, visualize_data=False):
# Select a random entry
ent = data[np.random.randint(len(data))]
# Get full paths
Img = cv2.imread(ent["image"])[..., ::-1] # Convert BGR to RGB
ann_map = cv2.imread(ent["annotation"], cv2.IMREAD_GRAYSCALE) # Read annotation as grayscale
if Img is None or ann_map is None:
print(f"Error: Could not read image or mask from path {ent['image']} or {ent['annotation']}")
return None, None, None, 0
# Resize image and mask
r = np.min([1024 / Img.shape[1], 1024 / Img.shape[0]]) # Scaling factor
Img = cv2.resize(Img, (int(Img.shape[1] * r), int(Img.shape[0] * r)))
ann_map = cv2.resize(ann_map, (int(ann_map.shape[1] * r), int(ann_map.shape[0] * r)), interpolation=cv2.INTER_NEAREST)
2. Binarisation des masques de segmentation : Le masque d'annotation multi-classes (qui peut comporter plusieurs classes d'objets étiquetées avec des valeurs de pixels différentes) est converti en un masque binaire. Ce masque met en évidence toutes les régions d'intérêt de l'image, simplifiant ainsi la tâche de segmentation en un problème de classification binaire : objet ou arrière-plan. Le masque binaire est ensuite érodé à l'aide d'un noyau 5x5.
L'érosion réduit légèrement la taille du masque, ce qui permet d'éviter les effets de bord lors de la sélection des points. Cela permet de s'assurer que les points sélectionnés se trouvent bien à l'intérieur de l'objet plutôt qu'à proximité de ses bords, qui peuvent être bruyants ou ambigus.
Les points clés sont sélectionnés à l'intérieur du masque érodé. Ces points agissent comme des invites au cours du processus de mise au point, en guidant le modèle sur les points sur lesquels il doit porter son attention. Les points sont sélectionnés au hasard à l'intérieur des objets afin de garantir qu'ils sont représentatifs et qu'ils ne sont pas influencés par des frontières bruyantes.
### Continuation of read_batch() ###
# Initialize a single binary mask
binary_mask = np.zeros_like(ann_map, dtype=np.uint8)
points = []
# Get binary masks and combine them into a single mask
inds = np.unique(ann_map)[1:] # Skip the background (index 0)
for ind in inds:
mask = (ann_map == ind).astype(np.uint8) # Create binary mask for each unique index
binary_mask = np.maximum(binary_mask, mask) # Combine with the existing binary mask
# Erode the combined binary mask to avoid boundary points
eroded_mask = cv2.erode(binary_mask, np.ones((5, 5), np.uint8), iterations=1)
# Get all coordinates inside the eroded mask and choose a random point
coords = np.argwhere(eroded_mask > 0)
if len(coords) > 0:
for _ in inds: # Select as many points as there are unique labels
yx = np.array(coords[np.random.randint(len(coords))])
points.append([yx[1], yx[0]])
points = np.array(points)
3. Visualisation : Cette étape est cruciale pour vérifier que les étapes de prétraitement des données ont été exécutées correctement. En inspectant visuellement les points du masque binarisé, vous pouvez vous assurer que le modèle recevra les données appropriées pendant la formation. Enfin, le masque binaire est remodelé et formaté correctement (avec des dimensions adaptées à l'entrée du modèle), et les points sont également remodelés en vue d'une utilisation ultérieure dans le processus de formation. La fonction renvoie l'image traitée, le masque binaire, les points sélectionnés et le nombre de masques trouvés.
### Continuation of read_batch() ###
if visualize_data:
# Plotting the images and points
plt.figure(figsize=(15, 5))
# Original Image
plt.subplot(1, 3, 1)
plt.title('Original Image')
plt.imshow(img)
plt.axis('off')
# Segmentation Mask (binary_mask)
plt.subplot(1, 3, 2)
plt.title('Binarized Mask')
plt.imshow(binary_mask, cmap='gray')
plt.axis('off')
# Mask with Points in Different Colors
plt.subplot(1, 3, 3)
plt.title('Binarized Mask with Points')
plt.imshow(binary_mask, cmap='gray')
# Plot points in different colors
colors = list(mcolors.TABLEAU_COLORS.values())
for i, point in enumerate(points):
plt.scatter(point[0], point[1], c=colors[i % len(colors)], s=100, label=f'Point {i+1}') # Corrected to plot y, x order
# plt.legend()
plt.axis('off')
plt.tight_layout()
plt.show()
binary_mask = np.expand_dims(binary_mask, axis=-1) # Now shape is (1024, 1024, 1)
binary_mask = binary_mask.transpose((2, 0, 1))
points = np.expand_dims(points, axis=1)
# Return the image, binarized mask, points, and number of masks
return img, binary_mask, points, len(inds)
# Visualize the data
Img1, masks1, points1, num_masks = read_batch(train_data, visualize_data=True)
Le code ci-dessus renvoie la figure suivante contenant l'image originale de l'ensemble de données ainsi que son masque binarisé et son masque binarisé avec des points.

Image originale, masque binarisé et masque binarisé avec points pour l'ensemble de données.
Étape 5 : Affiner le modèle SAM2
La mise au point du modèle SAM2 comporte plusieurs étapes, notamment le chargement du modèle, la configuration de l'optimiseur et du planificateur, et la mise à jour itérative des poids du modèle sur la base des données d'apprentissage.
Chargez les points de contrôle du modèle :
sam2_checkpoint = "sam2_hiera_small.pt" # @param ["sam2_hiera_tiny.pt", "sam2_hiera_small.pt", "sam2_hiera_base_plus.pt", "sam2_hiera_large.pt"]
model_cfg = "sam2_hiera_s.yaml" # @param ["sam2_hiera_t.yaml", "sam2_hiera_s.yaml", "sam2_hiera_b+.yaml", "sam2_hiera_l.yaml"]
sam2_model = build_sam2(model_cfg, sam2_checkpoint, device="cuda")
predictor = SAM2ImagePredictor(sam2_model)
Nous commençons par construire le modèle SAM2 en utilisant les points de contrôle pré-entraînés. Le modèle est ensuite intégré dans une classe de prédicteurs, ce qui simplifie le processus de définition des images, d'encodage des messages-guides et de décodage des masques.
Configurer les hyperparamètres
Nous configurons plusieurs hyperparamètres pour nous assurer que le modèle apprend efficacement, tels que le taux d'apprentissage, la décroissance des poids et les étapes d'accumulation du gradient. Ces hyperparamètres contrôlent le processus d'apprentissage, notamment la vitesse à laquelle le modèle met à jour ses poids et la manière dont il évite le surajustement. N'hésitez pas à jouer avec ces éléments.
# Train mask decoder.
predictor.model.sam_mask_decoder.train(True)
# Train prompt encoder.
predictor.model.sam_prompt_encoder.train(True)
# Configure optimizer.
optimizer=torch.optim.AdamW(params=predictor.model.parameters(),lr=0.0001,weight_decay=1e-4) #1e-5, weight_decay = 4e-5
# Mix precision.
scaler = torch.cuda.amp.GradScaler()
# No. of steps to train the model.
NO_OF_STEPS = 3000 # @param
# Fine-tuned model name.
FINE_TUNED_MODEL_NAME = "fine_tuned_sam2"
L'optimiseur est responsable de la mise à jour des poids du modèle, tandis que le planificateur ajuste le taux d'apprentissage pendant la formation afin d'améliorer la convergence. En affinant ces paramètres, nous pouvons obtenir une meilleure précision de segmentation.
Commencer la formation
Le processus d'affinage proprement dit est itératif : à chaque étape, un lot d'images et de masques pour les poumons uniquement est transmis au modèle, et la perte est calculée et utilisée pour mettre à jour les poids du modèle.
# Initialize scheduler
scheduler = torch.optim.lr_scheduler.StepLR(optimizer, step_size=500, gamma=0.2) # 500 , 250, gamma = 0.1
accumulation_steps = 4 # Number of steps to accumulate gradients before updating
for step in range(1, NO_OF_STEPS + 1):
with torch.cuda.amp.autocast():
image, mask, input_point, num_masks = read_batch(train_data, visualize_data=False)
if image is None or mask is None or num_masks == 0:
continue
input_label = np.ones((num_masks, 1))
if not isinstance(input_point, np.ndarray) or not isinstance(input_label, np.ndarray):
continue
if input_point.size == 0 or input_label.size == 0:
continue
predictor.set_image(image)
mask_input, unnorm_coords, labels, unnorm_box = predictor._prep_prompts(input_point, input_label, box=None, mask_logits=None, normalize_coords=True)
if unnorm_coords is None or labels is None or unnorm_coords.shape[0] == 0 or labels.shape[0] == 0:
continue
sparse_embeddings, dense_embeddings = predictor.model.sam_prompt_encoder(
points=(unnorm_coords, labels), boxes=None, masks=None,
)
batched_mode = unnorm_coords.shape[0] > 1
high_res_features = [feat_level[-1].unsqueeze(0) for feat_level in predictor._features["high_res_feats"]]
low_res_masks, prd_scores, _, _ = predictor.model.sam_mask_decoder(
image_embeddings=predictor._features["image_embed"][-1].unsqueeze(0),
image_pe=predictor.model.sam_prompt_encoder.get_dense_pe(),
sparse_prompt_embeddings=sparse_embeddings,
dense_prompt_embeddings=dense_embeddings,
multimask_output=True,
repeat_image=batched_mode,
high_res_features=high_res_features,
)
prd_masks = predictor._transforms.postprocess_masks(low_res_masks, predictor._orig_hw[-1])
gt_mask = torch.tensor(mask.astype(np.float32)).cuda()
prd_mask = torch.sigmoid(prd_masks[:, 0])
seg_loss = (-gt_mask * torch.log(prd_mask + 0.000001) - (1 - gt_mask) * torch.log((1 - prd_mask) + 0.00001)).mean()
inter = (gt_mask * (prd_mask > 0.5)).sum(1).sum(1)
iou = inter / (gt_mask.sum(1).sum(1) + (prd_mask > 0.5).sum(1).sum(1) - inter)
score_loss = torch.abs(prd_scores[:, 0] - iou).mean()
loss = seg_loss + score_loss * 0.05
# Apply gradient accumulation
loss = loss / accumulation_steps
scaler.scale(loss).backward()
# Clip gradients
torch.nn.utils.clip_grad_norm_(predictor.model.parameters(), max_norm=1.0)
if step % accumulation_steps == 0:
scaler.step(optimizer)
scaler.update()
predictor.model.zero_grad()
# Update scheduler
scheduler.step()
if step % 500 == 0:
FINE_TUNED_MODEL = FINE_TUNED_MODEL_NAME + "_" + str(step) + ".torch"
torch.save(predictor.model.state_dict(), FINE_TUNED_MODEL)
if step == 1:
mean_iou = 0
mean_iou = mean_iou * 0.99 + 0.01 * np.mean(iou.cpu().detach().numpy())
if step % 100 == 0:
print("Step " + str(step) + ":\t", "Accuracy (IoU) = ", mean_iou)
À chaque itération, le modèle traite un lot d'images, calcule les masques de segmentation et les compare à la vérité terrain pour calculer la perte. Cette perte est ensuite utilisée pour ajuster les poids du modèle, ce qui permet d'améliorer progressivement les performances du modèle. Après une formation d'environ 3000 époques, nous obtenons une précision (IoU - Intersection over Union) d'environ 72%.
Étape 6 : Inférence avec le modèle affiné
Le modèle peut ensuite être utilisé pour l'inférence, où il prédit les masques de segmentation sur de nouvelles images inédites. Commencez par lesfonctions d'aide read_images et get_points pour obtenir l'image d'inférence et son masque ainsi que les points clés.
def read_image(image_path, mask_path): # read and resize image and mask
img = cv2.imread(image_path)[..., ::-1] # Convert BGR to RGB
mask = cv2.imread(mask_path, 0)
r = np.min([1024 / img.shape[1], 1024 / img.shape[0]])
img = cv2.resize(img, (int(img.shape[1] * r), int(img.shape[0] * r)))
mask = cv2.resize(mask, (int(mask.shape[1] * r), int(mask.shape[0] * r)), interpolation=cv2.INTER_NEAREST)
return img, mask
def get_points(mask, num_points): # Sample points inside the input mask
points = []
coords = np.argwhere(mask > 0)
for i in range(num_points):
yx = np.array(coords[np.random.randint(len(coords))])
points.append([[yx[1], yx[0]]])
return np.array(points)Chargez ensuite les échantillons d'images que vous souhaitez utiliser pour l'inférence, ainsi que les poids nouvellement ajustés, et effectuez l'inférence en paramétrant la fonction torch.no_grad().
# Randomly select a test image from the test_data
selected_entry = random.choice(test_data)
image_path = selected_entry['image']
mask_path = selected_entry['annotation']
# Load the selected image and mask
image, mask = read_image(image_path, mask_path)
# Generate random points for the input
num_samples = 30 # Number of points per segment to sample
input_points = get_points(mask, num_samples)
# Load the fine-tuned model
FINE_TUNED_MODEL_WEIGHTS = "fine_tuned_sam2_1000.torch"
sam2_model = build_sam2(model_cfg, sam2_checkpoint, device="cuda")
# Build net and load weights
predictor = SAM2ImagePredictor(sam2_model)
predictor.model.load_state_dict(torch.load(FINE_TUNED_MODEL_WEIGHTS))
# Perform inference and predict masks
with torch.no_grad():
predictor.set_image(image)
masks, scores, logits = predictor.predict(
point_coords=input_points,
point_labels=np.ones([input_points.shape[0], 1])
)
# Process the predicted masks and sort by scores
np_masks = np.array(masks[:, 0])
np_scores = scores[:, 0]
sorted_masks = np_masks[np.argsort(np_scores)][::-1]
# Initialize segmentation map and occupancy mask
seg_map = np.zeros_like(sorted_masks[0], dtype=np.uint8)
occupancy_mask = np.zeros_like(sorted_masks[0], dtype=bool)
# Combine masks to create the final segmentation map
for i in range(sorted_masks.shape[0]):
mask = sorted_masks[i]
if (mask * occupancy_mask).sum() / mask.sum() > 0.15:
continue
mask_bool = mask.astype(bool)
mask_bool[occupancy_mask] = False # Set overlapping areas to False in the mask
seg_map[mask_bool] = i + 1 # Use boolean mask to index seg_map
occupancy_mask[mask_bool] = True # Update occupancy_mask
# Visualization: Show the original image, mask, and final segmentation side by side
plt.figure(figsize=(18, 6))
plt.subplot(1, 3, 1)
plt.title('Test Image')
plt.imshow(image)
plt.axis('off')
plt.subplot(1, 3, 2)
plt.title('Original Mask')
plt.imshow(mask, cmap='gray')
plt.axis('off')
plt.subplot(1, 3, 3)
plt.title('Final Segmentation')
plt.imshow(seg_map, cmap='jet')
plt.axis('off')
plt.tight_layout()
plt.show()Dans cette étape, nous utilisons le modèle affiné pour générer des masques de segmentation pour les images de test. Les masques prédits sont ensuite visualisés avec les images originales et les masques de vérité terrain pour évaluer la performance du modèle.

Image de segmentation finale sur les données de test
Conclusion
Le réglage fin de SAM2 offre un moyen pratique d'améliorer ses capacités pour des tâches spécifiques. Que vous travailliez sur l'imagerie médicale, les véhicules autonomes ou le montage vidéo, le réglage fin vous permet d'utiliser SAM2 pour vos besoins spécifiques. En suivant ce guide, vous pouvez adapter SAM2 à vos projets et obtenir des résultats de segmentation de pointe.
Pour des cas d'utilisation plus avancés, envisagez d'affiner des composants supplémentaires de SAM2, tels que l'encodeur d'images. Bien que cela nécessite plus de ressources, cela offre une plus grande flexibilité et des améliorations de performance.
Obtenez une certification de haut niveau en matière d'IA

Je suis experte Google Developers en ML (Gen AI), triple experte Kaggle et ambassadrice Women Techmakers, avec plus de trois ans d’expérience dans la tech. J’ai cofondé une startup dans le domaine de la santé en 2020 et je poursuis actuellement un master en informatique à Georgia Tech, avec une spécialisation en apprentissage automatique.
