Modelos basados en árboles en R
La partición recursiva es una herramienta fundamental en la minería de datos. Nos ayuda a explorar la estructura de un conjunto de datos, a la vez que desarrollamos reglas de decisión fáciles de visualizar para predecir un resultado categórico (árbol de clasificación) o continuo (árbol de regresión). Esta sección describe brevemente el modelado CART, los árboles de inferencia condicional y los bosques aleatorios.
Modelización CART mediante rpart
Se pueden generar árboles de clasificación y regresión (como los descritos por Brieman, Freidman, Olshen y Stone) mediante el paquete rpart. Encontrarás información detallada sobre rpart en Introducción al Particionado Recursivo mediante las Rutinas RPART</a >. A continuación se indican los pasos generales, seguidos de dos ejemplos.
1. Haz crecer el árbol
Para hacer crecer un árbol,userpart(fórmula, datos=, método=,control=) donde
| formula | tiene el formatoresultado ~ predictor1+predictor2+predictor3+ect . |
| data= | especifica el marco de datos |
| method= | " clase" para un árbol de clasificación"anova"</strong > para un árbol de regresión |
| control= | parámetros opcionales para controlar el crecimiento de los árboles. Por ejemplo, control=rpart.control(minsplit=30, cp=0,001) exige que el número mínimo de observaciones en un nodo sea 30 antes de intentar una división y que una división debe disminuir la falta de ajuste global en un factor de 0,001 (factor de complejidad del coste) antes de intentarse. |
2. Examina los resultados
Las siguientes funciones nos ayudan a examinar los resultados.
| printcp(encajar) | mostrar tabla cp |
| trazarcp(ajustar) | trazar los resultados de la validación cruzada |
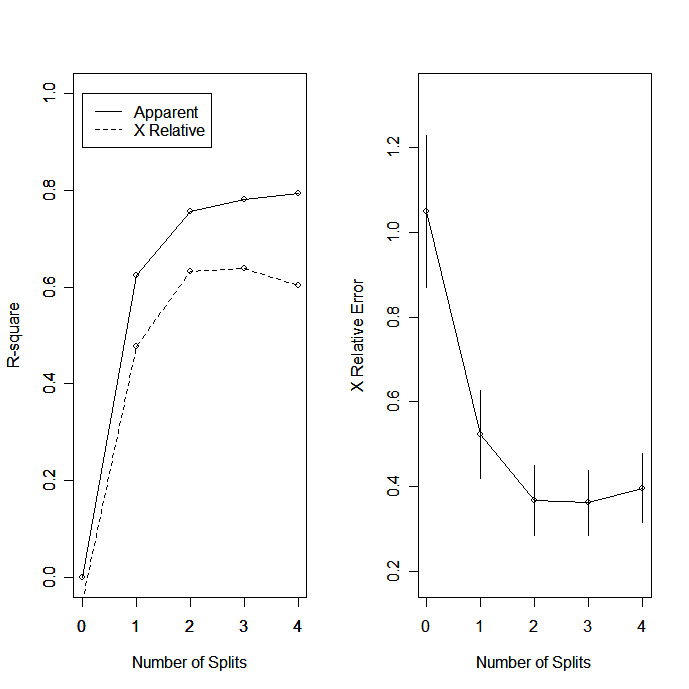
| rsq.rpart(fit) | traza el R-cuadrado aproximado y el error relativo para diferentes divisiones (2 trazados). las etiquetas sólo son apropiadas para el método "anova". |
| imprimir(ajustar) | imprimir resultados |
| summary(fit) | resultados detallados, incluidas las divisiones sustitutivas |
| trazar(ajustar) | trazar árbol de decisión |
| text(fit) | etiqueta el diagrama del árbol de decisión |
| post(fit, file=) | crear un gráfico postscript del árbol de decisión |
En los árboles creados por rpart( ), pasa a la rama IZQUIERDA cuando la condición indicada es verdadera (ver los gráficos de abajo).
3. podar el árbol
Poda el árbol para evitar el sobreajuste de los datos. Normalmente, querrás seleccionar un tamaño de árbol que minimice el error de validación cruzada, la columna xerror impresa por printcp( ).
Poda el árbol al tamaño deseado utilizandoprune(fit, cp= )
En concreto, utiliza printcp ( ) para examinar los resultados de error de validación cruzada, selecciona el parámetro de complejidad asociado al error mínimo y colócalo en la función prune( ). Como alternativa, puedes utilizar el fragmento de código
fit$cptable[which.min(fit$cptable[, "xerror"]), "CP"]</strong >
para seleccionar automáticamente el parámetro de complejidad asociado al menor error de validación cruzada. Gracias a HSAUR por esta idea.
Ejemplo de árbol de clasificación
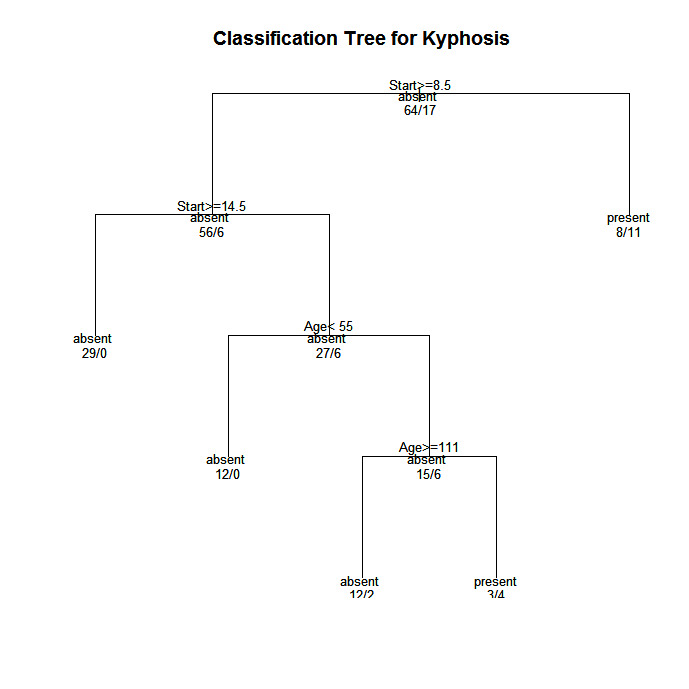
Utilicemos el marco de datos cifosis para predecir un tipo de deformación (cifosis) tras la cirugía, a partir de la edad en meses (Edad), el número de vértebras implicadas (Número) y la vértebra más alta operada (Inicio).
# Classification Tree with rpart
library(rpart)
# grow tree
fit <- rpart(Kyphosis ~ Age + Number + Start,
method="class", data=kyphosis)
printcp(fit) # display the results
plotcp(fit) # visualize cross-validation results
summary(fit) # detailed summary of splits
# plot tree
plot(fit, uniform=TRUE,
main="Classification Tree for Kyphosis")
text(fit, use.n=TRUE, all=TRUE, cex=.8)
# create attractive postscript plot of tree
post(fit, file = "c:/tree.ps",
title = "Classification Tree for Kyphosis")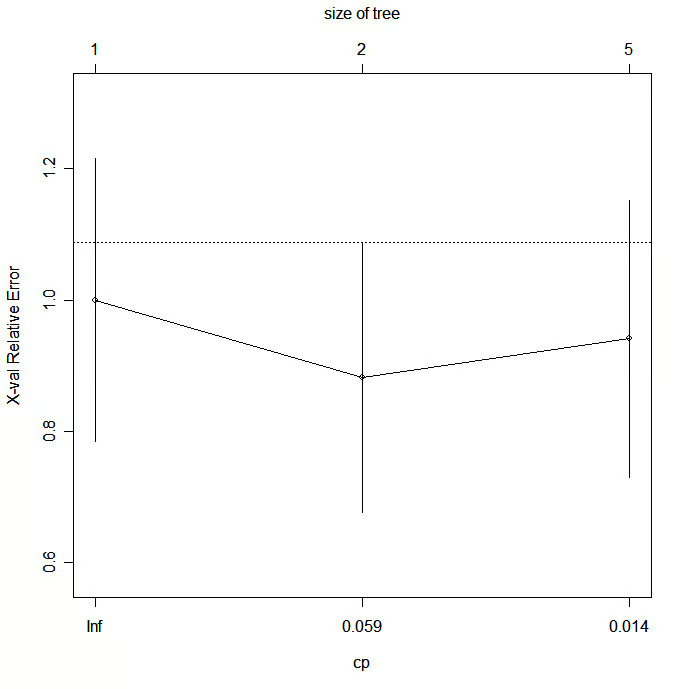
# prune the tree
pfit<- prune(fit, cp=
fit$cptable[which.min(fit$cptable[,"xerror"]),"CP"])
# plot the pruned tree
plot(pfit, uniform=TRUE,
main="Pruned Classification Tree for Kyphosis")
text(pfit, use.n=TRUE, all=TRUE, cex=.8)
post(pfit, file = "c:/ptree.ps",
title = "Pruned Classification Tree for Kyphosis")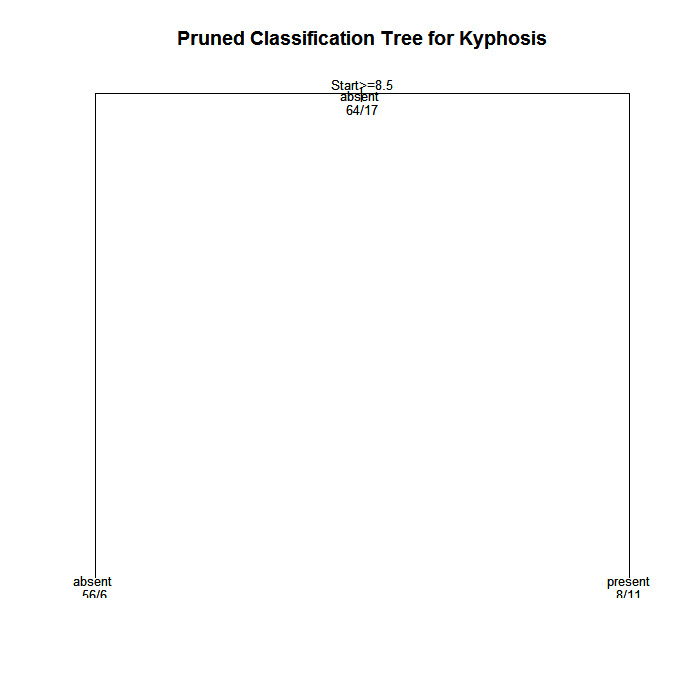

Ejemplo de árbol de regresión
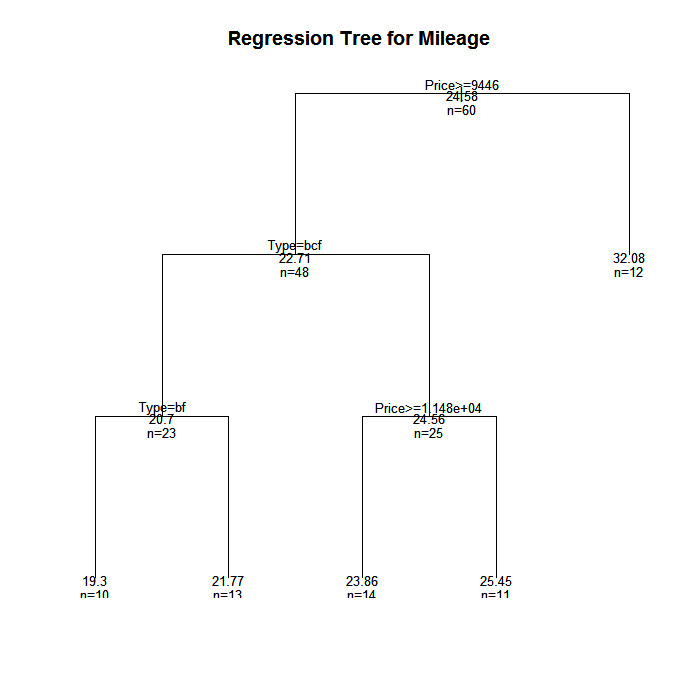
En este ejemplo predeciremos el kilometraje de un coche a partir del precio, el país, la fiabilidad y el tipo de coche. El marco de datos es cu.resumen.
# Regression Tree Example
library(rpart)
# grow tree
fit <- rpart(Mileage~Price + Country + Reliability + Type,
method="anova", data=cu.summary)
printcp(fit) # display the results
plotcp(fit) # visualize cross-validation results
summary(fit) # detailed summary of splits
# create additional plots
par(mfrow=c(1,2)) # two plots on one page
rsq.rpart(fit) # visualize cross-validation results
# plot tree
plot(fit, uniform=TRUE,
main="Regression Tree for Mileage ")
text(fit, use.n=TRUE, all=TRUE, cex=.8)
# create attractive postcript plot of tree
post(fit, file = "c:/tree2.ps",
title = "Regression Tree for Mileage ")
# prune the tree
pfit<- prune(fit, cp=0.01160389) # from cptable
# plot the pruned tree
plot(pfit, uniform=TRUE,
main="Pruned Regression Tree for Mileage")
text(pfit, use.n=TRUE, all=TRUE, cex=.8)
post(pfit, file = "c:/ptree2.ps",
title = "Pruned Regression Tree for Mileage")Resulta que esto produce el mismo árbol que el original.
Árboles de inferencia condicional mediante parte
El paquete party proporciona árboles de regresión no paramétricos para respuestas nominales, ordinales, numéricas, censuradas y multivariantes. party: Un laboratorio para la partición recursiva</a >, proporciona detalles.
Puedes crear un árbol de regresión o de clasificación mediante la función
ctree(fórmula, datos=)El tipo de árbol creado dependerá de la variable resultado (factor nominal, factor ordenado, numérico, etc.). El crecimiento del árbol se basa en reglas estadísticas de parada, por lo que no debería ser necesaria la poda.
A continuación se vuelven a analizar los dos ejemplos anteriores.
# Conditional Inference Tree for Kyphosis
library(party)
fit <- ctree(Kyphosis ~ Age + Number + Start,
data=kyphosis)
plot(fit, main="Conditional Inference Tree for Kyphosis")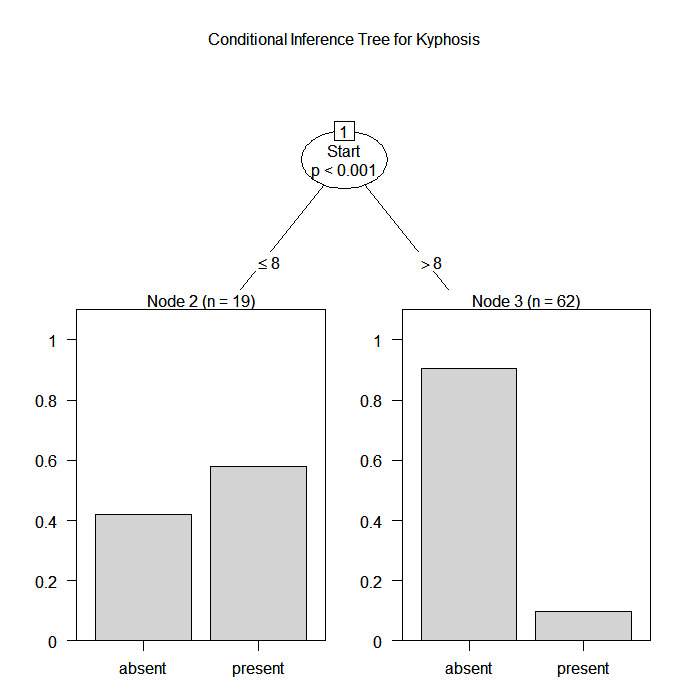
# Conditional Inference Tree for Mileage
library(party)
fit2 <- ctree(Mileage~Price + Country + Reliability + Type,
data=na.omit(cu.summary))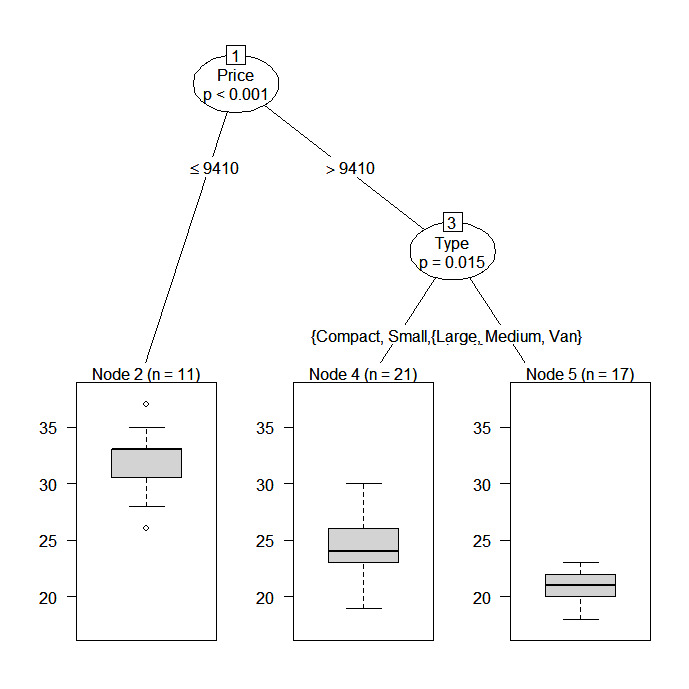
Bosques aleatorios
Los bosques aleatorios mejoran la precisión predictiva generando un gran número de árboles bootstrapped (basados en muestras aleatorias de variables), clasificando un caso utilizando cada árbol de este nuevo "bosque", y decidiendo un resultado predictivo final combinando los resultados de todos los árboles (una media en regresión, un voto mayoritario en clasificación). El enfoque de bosque aleatorio de Breiman y Cutler se implanta mediante el paquete randomForest</a > .
He aquí un ejemplo.
# Random Forest prediction of Kyphosis data
library(randomForest)
fit <- randomForest(Kyphosis ~ Age + Number + Start, data=kyphosis)
print(fit) # view results
importance(fit) # importance of each predictorPara más detalles, consulta el sitio web completo de Random Forest</a > .
Ir más lejos
En esta sección sólo se han mencionado las opciones disponibles. Para saber más, consulta la Visión de Tareas de CRAN sobre Aprendizaje Automático y Estadístico</a > .
Practicar
Prueba el curso Aprendizaje Supervisado en R</a > que incluye un ejercicio con Bosques Aleatorios .