Baum-basierte Modelle in R
Die rekursive Partitionierung ist ein grundlegendes Werkzeug im Data Mining. Er hilft uns, die Struktur eines Datensatzes zu erforschen und gleichzeitig einfach zu visualisierende Entscheidungsregeln für die Vorhersage eines kategorischen (Klassifikationsbaum) oder kontinuierlichen (Regressionsbaum) Ergebnisses zu entwickeln. Dieser Abschnitt beschreibt kurz die CART-Modellierung, bedingte Inferenzbäume und Zufallswälder.
CART-Modellierung über rpart
Klassifizierungs- und Regressionsbäume (wie von Brieman, Freidman, Olshen und Stone beschrieben) können mit dem Paket rpart erstellt werden. Ausführliche Informationen zu rpart findest du in An Introduction to Recursive Partitioning Using the RPART Routines</a >. Im Folgenden findest du die allgemeinen Schritte, gefolgt von zwei Beispielen.
1. Den Baum wachsen lassen
Um einen Baum wachsen zu lassen,userpart(formula, data=, method=,control=) wo
| formula | hat das FormatErgebnis ~ Prädiktor1+Prädiktor2+Prädiktor3+Effekt. |
| data= | gibt den Datenrahmen an |
| method= | "Klasse" für einen Klassifikationsbaum"anova"</strong > für einen Regressionsbaum |
| control= | optionale Parameter zur Steuerung des Baumwachstums. Zum Beispiel verlangt control=rpart.control(minsplit=30, cp=0.001), dass die Mindestanzahl der Beobachtungen in einem Knoten 30 beträgt, bevor ein Split versucht wird, und dass ein Split die Gesamtunzulänglichkeit um den Faktor 0.001 (Kostenkomplexitätsfaktor) verringern muss, bevor er versucht wird. |
2. Untersuche die Ergebnisse
Die folgenden Funktionen helfen uns, die Ergebnisse zu untersuchen.
| printcp(fit) | cp-Tabelle anzeigen |
| plotcp(fit) | Kreuzvalidierungsergebnisse darstellen |
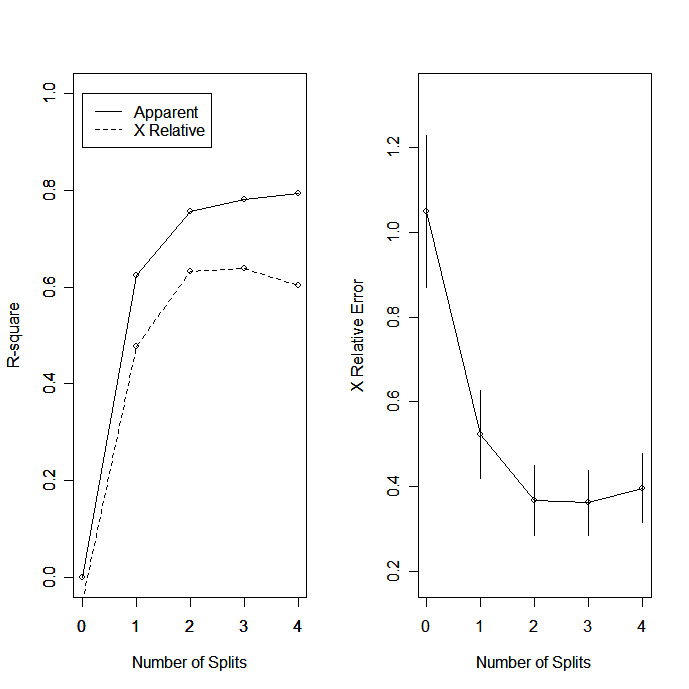
| rsq.rpart(fit) | Stelle das ungefähre R-Quadrat und den relativen Fehler für verschiedene Splits dar (2 Plots). Die Beschriftungen sind nur für die "Anova"-Methode geeignet. |
| drucken(fit) | Druckergebnisse |
| Zusammenfassung(fit) | Detaillierte Ergebnisse einschließlich Surrogat-Splits |
| plot(fit) | Entscheidungsbaum erstellen |
| text(fit) | beschrifte den Entscheidungsbaumplot |
| post(fit, file=) | Postscript-Diagramm des Entscheidungsbaums erstellen |
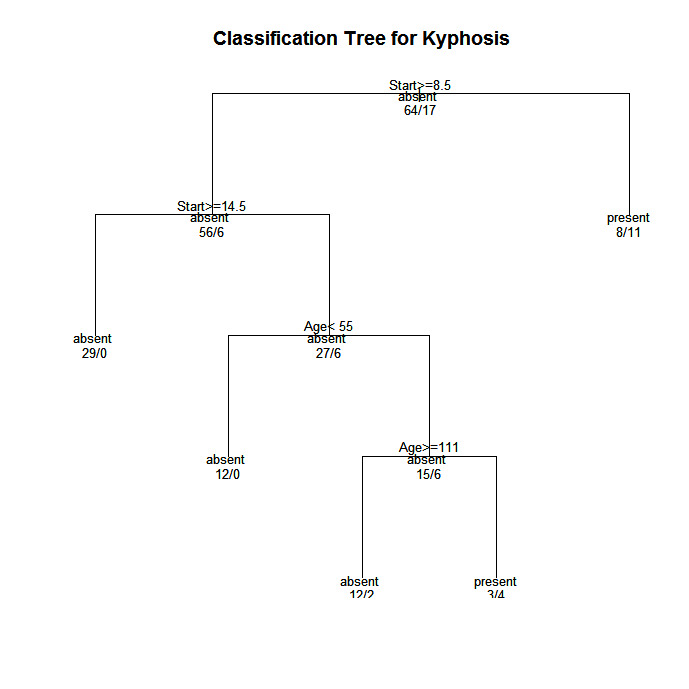
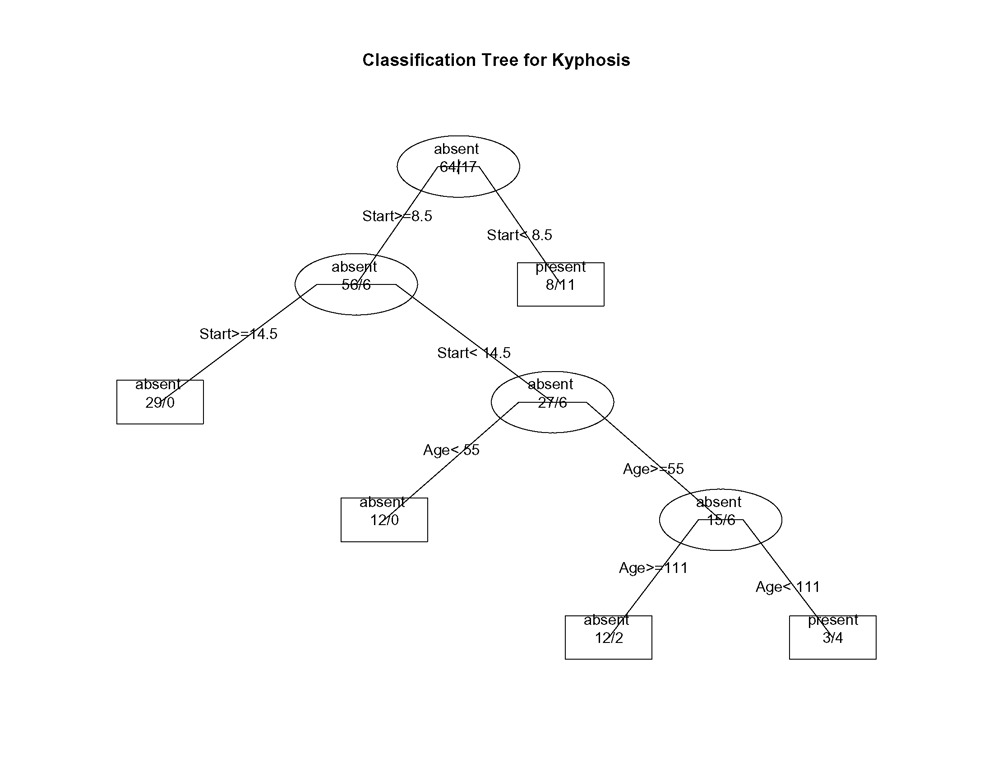
In Bäumen, die mit rpart( ) erstellt wurden, wird zum LINKEN Zweig gewechselt, wenn die angegebene Bedingung erfüllt ist (siehe die Grafiken unten).
3. Baum beschneiden
Schneide den Baum zurück, um eine Überanpassung der Daten zu vermeiden. Normalerweise solltest du eine Baumgröße wählen, die den kreuzvalidierten Fehler minimiert, also die xerror-Spalte, die von printcp( ) ausgegeben wird.
Beschneide den Baum auf die gewünschte Größe mitprune(fit, cp= )
Verwende printcp( ), um die kreuzvalidierten Fehlerergebnisse zu untersuchen, wähle den Komplexitätsparameter aus, der mit dem geringsten Fehler verbunden ist, und gib ihn in die Funktion prune( ) ein. Alternativ kannst du das folgende Codefragment verwenden
fit$cptable[which.min(fit$cptable[, "xerror"]), "CP"]</strong >
um automatisch den Komplexitätsparameter auszuwählen, der den kleinsten kreuzvalidierten Fehler aufweist. Danke an HSAUR für diese Idee.
Beispiel für einen Klassifikationsbaum
Verwenden wir den Datenrahmen Kyphose, um eine Art der Verformung (Kyphose) nach einer Operation vorherzusagen, und zwar anhand des Alters in Monaten (Alter), der Anzahl der betroffenen Wirbel (Anzahl) und des höchsten operierten Wirbels (Start).
# Classification Tree with rpart
library(rpart)
# grow tree
fit <- rpart(Kyphosis ~ Age + Number + Start,
method="class", data=kyphosis)
printcp(fit) # display the results
plotcp(fit) # visualize cross-validation results
summary(fit) # detailed summary of splits
# plot tree
plot(fit, uniform=TRUE,
main="Classification Tree for Kyphosis")
text(fit, use.n=TRUE, all=TRUE, cex=.8)
# create attractive postscript plot of tree
post(fit, file = "c:/tree.ps",
title = "Classification Tree for Kyphosis")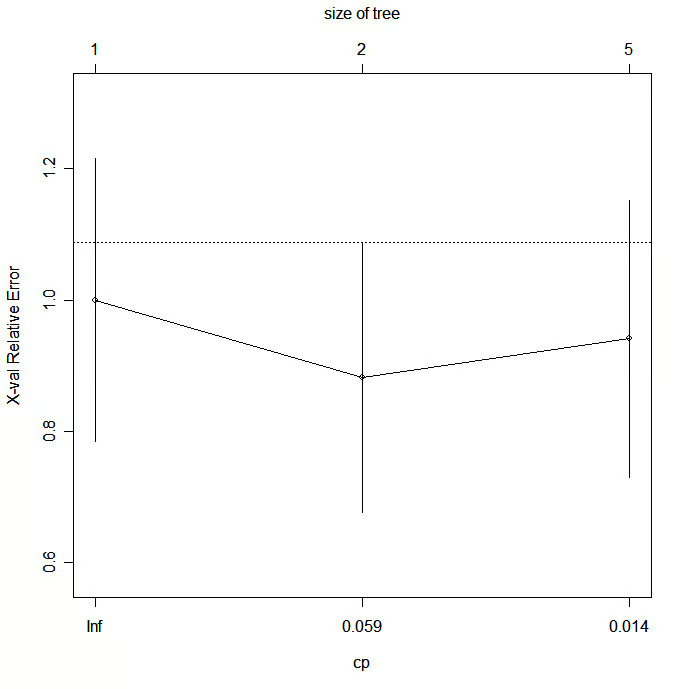
# prune the tree
pfit<- prune(fit, cp=
fit$cptable[which.min(fit$cptable[,"xerror"]),"CP"])
# plot the pruned tree
plot(pfit, uniform=TRUE,
main="Pruned Classification Tree for Kyphosis")
text(pfit, use.n=TRUE, all=TRUE, cex=.8)
post(pfit, file = "c:/ptree.ps",
title = "Pruned Classification Tree for Kyphosis")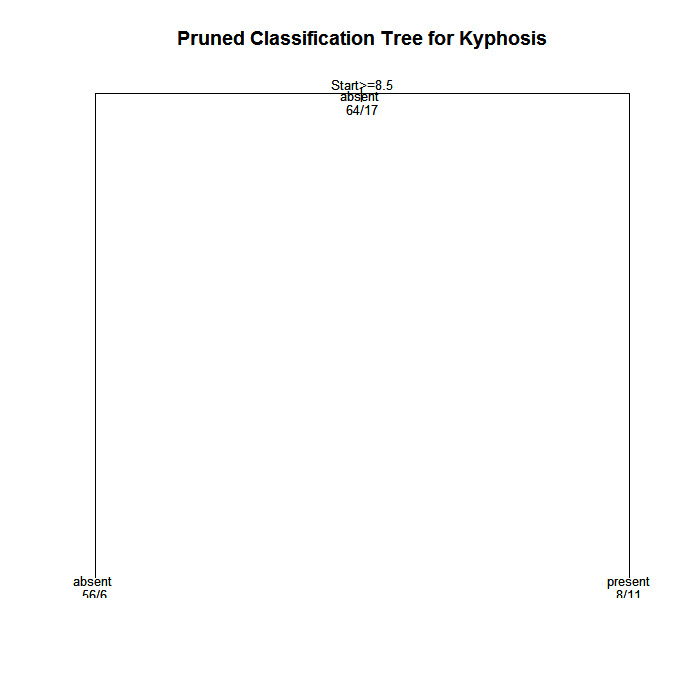

Beispiel für einen Regressionsbaum
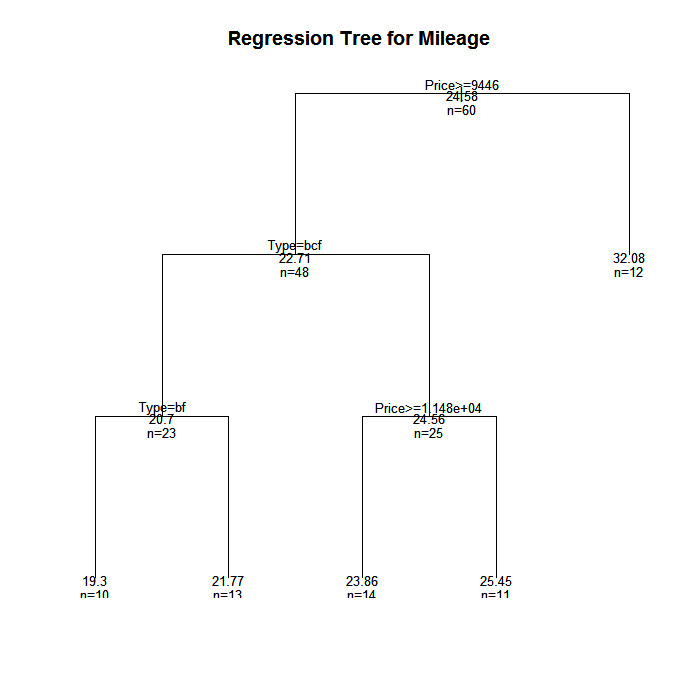
In diesem Beispiel werden wir den Kilometerstand eines Autos anhand des Preises, des Landes, der Zuverlässigkeit und des Fahrzeugtyps vorhersagen. Der Datenrahmen ist cu.summary.
# Regression Tree Example
library(rpart)
# grow tree
fit <- rpart(Mileage~Price + Country + Reliability + Type,
method="anova", data=cu.summary)
printcp(fit) # display the results
plotcp(fit) # visualize cross-validation results
summary(fit) # detailed summary of splits
# create additional plots
par(mfrow=c(1,2)) # two plots on one page
rsq.rpart(fit) # visualize cross-validation results
# plot tree
plot(fit, uniform=TRUE,
main="Regression Tree for Mileage ")
text(fit, use.n=TRUE, all=TRUE, cex=.8)
# create attractive postcript plot of tree
post(fit, file = "c:/tree2.ps",
title = "Regression Tree for Mileage ")
# prune the tree
pfit<- prune(fit, cp=0.01160389) # from cptable
# plot the pruned tree
plot(pfit, uniform=TRUE,
main="Pruned Regression Tree for Mileage")
text(pfit, use.n=TRUE, all=TRUE, cex=.8)
post(pfit, file = "c:/ptree2.ps",
title = "Pruned Regression Tree for Mileage")Es stellt sich heraus, dass dies denselben Baum ergibt wie das Original.
Bedingte Inferenzbäume über die Partei
Das Party-Paket bietet nichtparametrische Regressionsbäume für nominale, ordinale, numerische, zensierte und multivariate Antworten. party: Ein Labor für rekursive Partitionierung</a >, liefert Details.
Du kannst einen Regressions- oder Klassifikationsbaum über die Funktion
ctree(formula, data=)Die Art des erstellten Baums hängt von der Ergebnisvariable ab (nominaler Faktor, geordneter Faktor, numerisch, etc.). Das Wachstum der Bäume basiert auf statistischen Stoppregeln, sodass ein Beschneiden nicht erforderlich sein sollte.
Die beiden vorherigen Beispiele werden im Folgenden erneut analysiert.
# Conditional Inference Tree for Kyphosis
library(party)
fit <- ctree(Kyphosis ~ Age + Number + Start,
data=kyphosis)
plot(fit, main="Conditional Inference Tree for Kyphosis")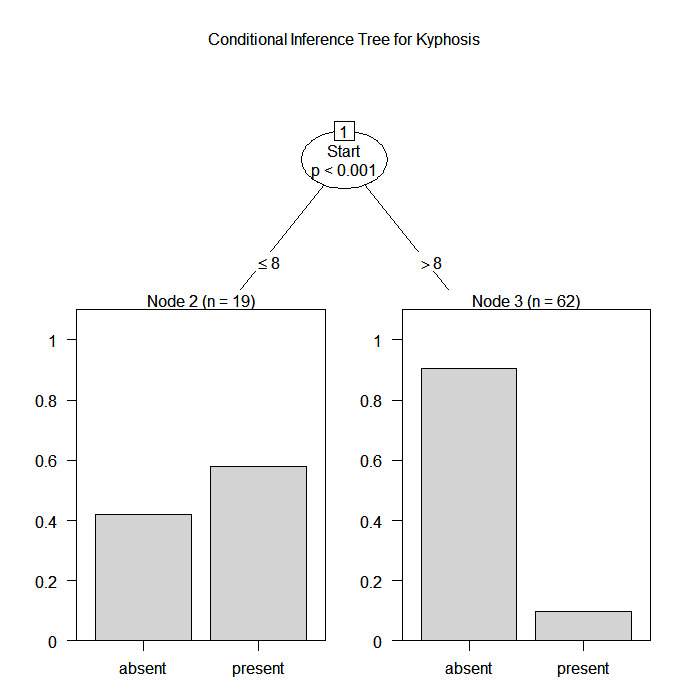
# Conditional Inference Tree for Mileage
library(party)
fit2 <- ctree(Mileage~Price + Country + Reliability + Type,
data=na.omit(cu.summary))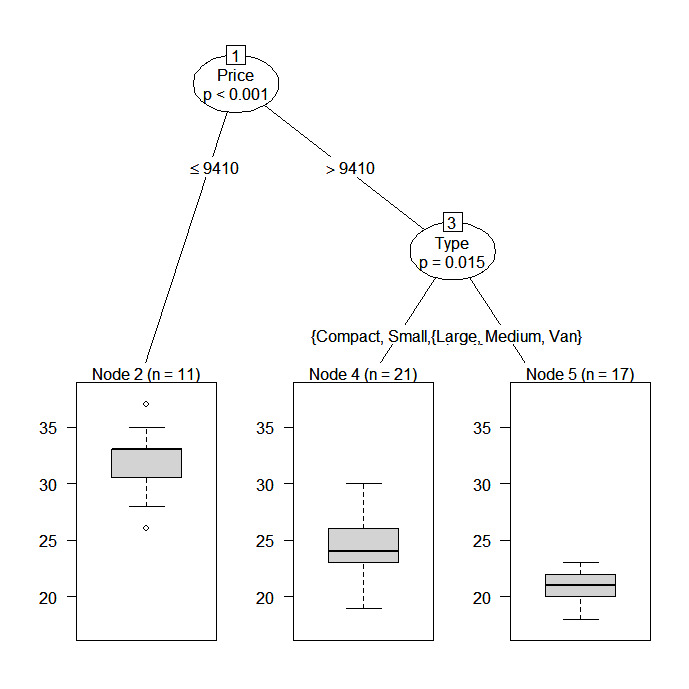
Zufallsforsten
Zufallswälder verbessern die Vorhersagegenauigkeit, indem sie eine große Anzahl von Bootstrap-Bäumen (basierend auf Zufallsstichproben von Variablen) erzeugen, einen Fall mit jedem Baum in diesem neuen "Wald" klassifizieren und ein endgültiges Vorhersageergebnis durch die Kombination der Ergebnisse aller Bäume bestimmen (ein Durchschnitt bei der Regression, ein Mehrheitsvotum bei der Klassifizierung). Der Random-Forest-Ansatz von Breiman und Cutler wird mit dem Paket randomForest</a> umgesetzt.
Hier ist ein Beispiel.
# Random Forest prediction of Kyphosis data
library(randomForest)
fit <- randomForest(Kyphosis ~ Age + Number + Start, data=kyphosis)
print(fit) # view results
importance(fit) # importance of each predictorWeitere Informationen findest du auf der umfassenden Random Forest Website</a > .
Weiter gehen
In diesem Abschnitt wurden die verfügbaren Optionen nur angerissen. Weitere Informationen findest du in der CRAN Task View on Machine & Statistical Learning</a > .
Zum Üben
Probiere den Kurs Überwachtes Lernen in R</a > aus, der eine Übung mit Random Forests enthält .