Modelos baseados em árvores no R
O particionamento recursivo é uma ferramenta fundamental na mineração de dados. Ele nos ajuda a explorar a estrutura de um conjunto de dados e a desenvolver regras de decisão fáceis de visualizar para prever um resultado categórico (árvore de classificação) ou contínuo (árvore de regressão). Esta seção descreve brevemente a modelagem CART, as árvores de inferência condicional e as florestas aleatórias.
Modelagem CART via rpart
As árvores de classificação e regressão (conforme descrito por Brieman, Freidman, Olshen e Stone) podem ser geradas por meio do pacote rpart. Informações detalhadas sobre o rpart estão disponíveis em An Introduction to Recursive Partitioning Using the RPART Routines</a >. As etapas gerais são fornecidas abaixo, seguidas de dois exemplos.
1. Cultivar a árvore
Para desenvolver uma árvore,userpart(formula, data=, method=,control=) where
| fórmula | está no formatooutcome ~ predictor1+predictor2+predictor3+ect. |
| data= | especifica o quadro de dados |
| método= | "class" para uma árvore de classificação"anova"</strong > para uma árvore de regressão |
| control= | parâmetros opcionais para controlar o crescimento das árvores. Por exemplo, control=rpart.control(minsplit=30, cp=0,001) exige que o número mínimo de observações em um nó seja 30 antes de tentar uma divisão e que uma divisão diminua a falta de ajuste geral por um fator de 0,001 (fator de complexidade de custo) antes de ser tentada. |
2. Examine os resultados
As funções a seguir nos ajudam a examinar os resultados.
| printcp(fit) | exibir tabela cp |
| plotcp(fit) | plotar resultados de validação cruzada |
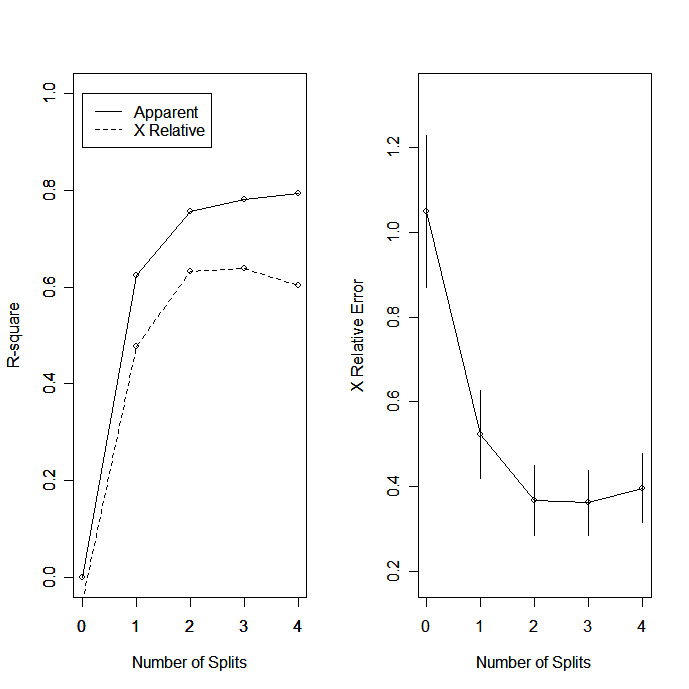
| rsq.rpart(fit) | traçar o R-quadrado aproximado e o erro relativo para diferentes divisões (2 gráficos). os rótulos são apropriados apenas para o método "anova". |
| print(fit) | imprimir resultados |
| resumo(fit) | resultados detalhados, incluindo divisões substitutas |
| plotar(fit) | plotar árvore de decisão |
| text(fit) | rotular o gráfico da árvore de decisão |
| post(fit, file=) | Criar um gráfico postscript da árvore de decisão |
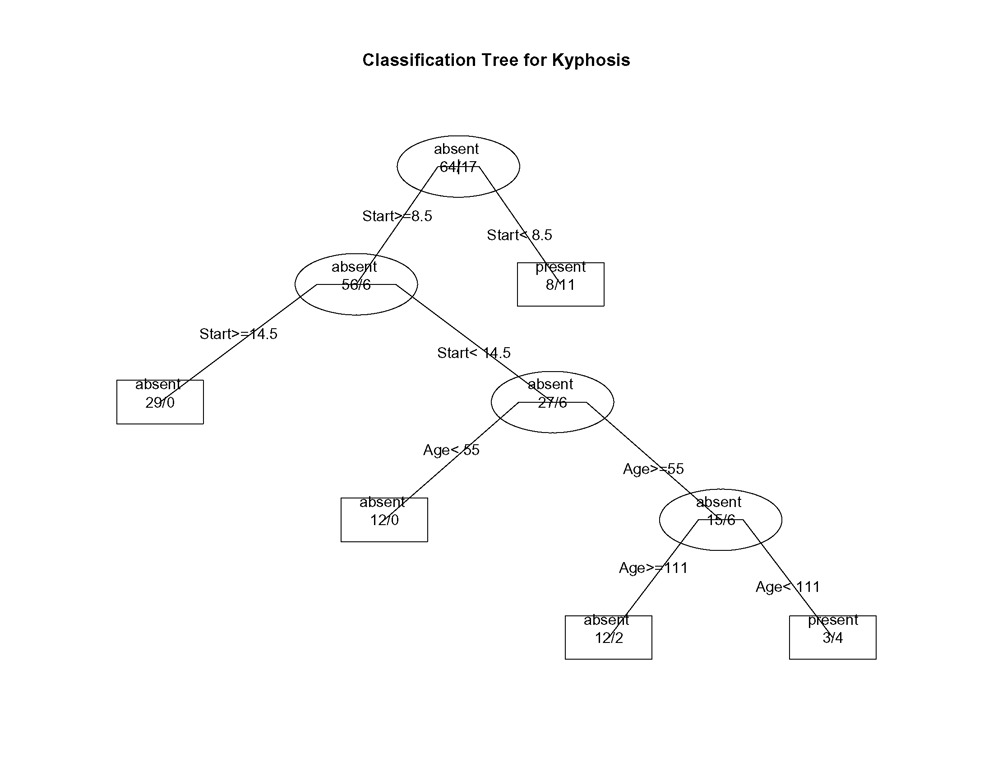
Em árvores criadas por rpart( ), vá para o ramo LEFT quando a condição declarada for verdadeira (veja os gráficos abaixo).
3. podar a árvore
Podar a árvore para evitar o ajuste excessivo dos dados. Normalmente, você desejará selecionar um tamanho de árvore que minimize o erro de validação cruzada, a coluna xerror impressa por printcp( ).
Podar a árvore para o tamanho desejado usandoprune(fit, cp= )
Especificamente, use printcp( ) para examinar os resultados de erro com validação cruzada, selecione o parâmetro de complexidade associado ao erro mínimo e coloque-o na função prune( ). Como alternativa, você pode usar o fragmento de código
fit$cptable[which.min(fit$cptable[, "xerror"]), "CP"]</strong >
para selecionar automaticamente o parâmetro de complexidade associado ao menor erro de validação cruzada. Agradecemos à HSAUR por essa ideia.
Exemplo de árvore de classificação
Vamos usar o quadro de dados cifose para prever um tipo de deformação (cifose) após a cirurgia, com base na idade em meses (Age), no número de vértebras envolvidas (Number) e na vértebra mais alta operada (Start).
# Classification Tree with rpart
library(rpart)
# grow tree
fit <- rpart(Kyphosis ~ Age + Number + Start,
method="class", data=kyphosis)
printcp(fit) # display the results
plotcp(fit) # visualize cross-validation results
summary(fit) # detailed summary of splits
# plot tree
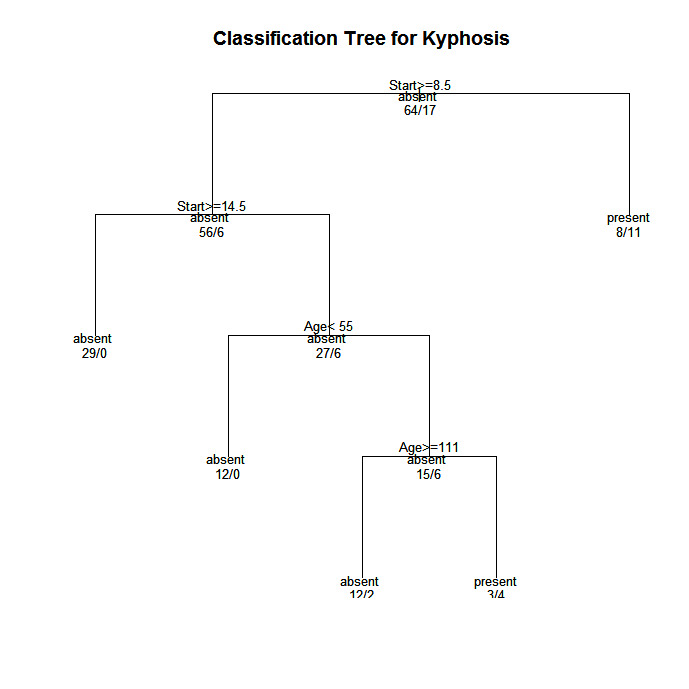
plot(fit, uniform=TRUE,
main="Classification Tree for Kyphosis")
text(fit, use.n=TRUE, all=TRUE, cex=.8)
# create attractive postscript plot of tree
post(fit, file = "c:/tree.ps",
title = "Classification Tree for Kyphosis")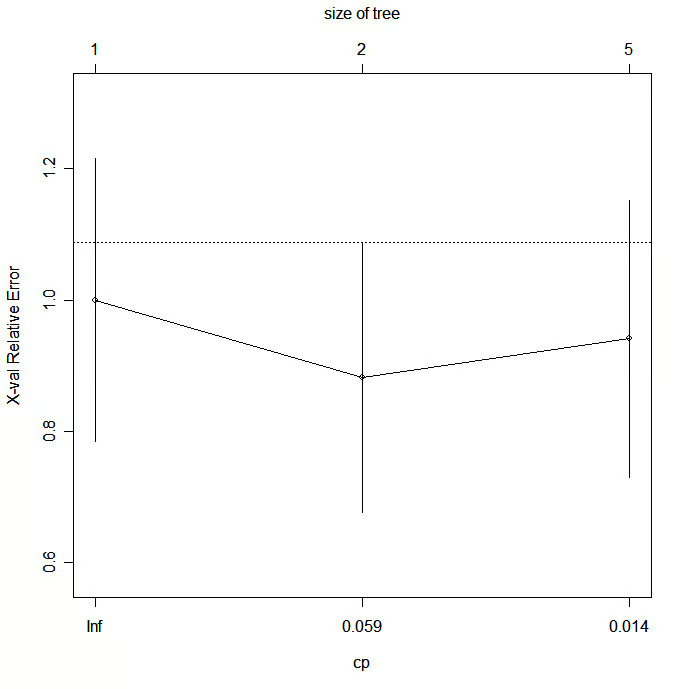
# prune the tree
pfit<- prune(fit, cp=
fit$cptable[which.min(fit$cptable[,"xerror"]),"CP"])
# plot the pruned tree
plot(pfit, uniform=TRUE,
main="Pruned Classification Tree for Kyphosis")
text(pfit, use.n=TRUE, all=TRUE, cex=.8)
post(pfit, file = "c:/ptree.ps",
title = "Pruned Classification Tree for Kyphosis")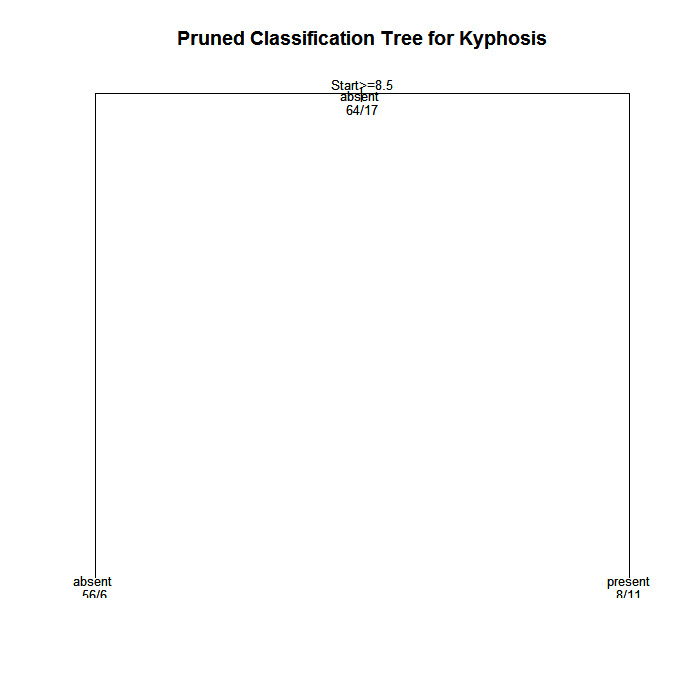

Exemplo de árvore de regressão
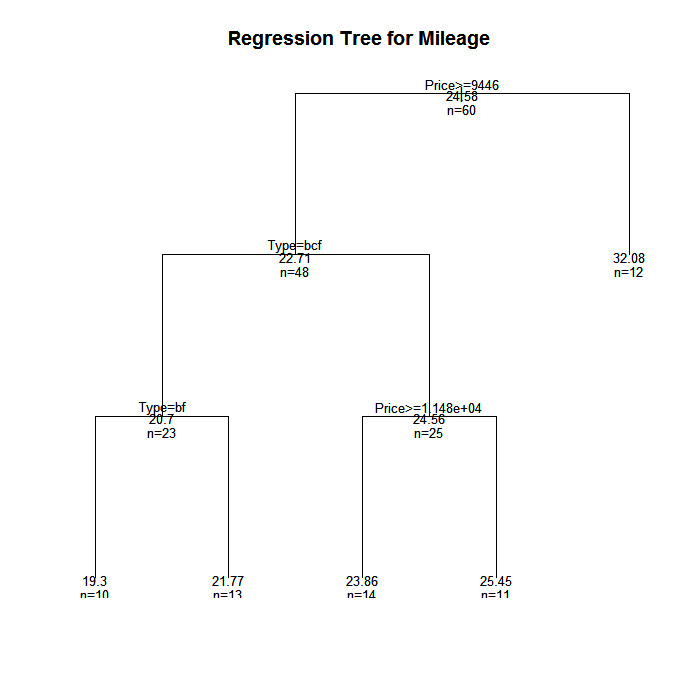
Neste exemplo, vamos prever a quilometragem do carro com base no preço, no país, na confiabilidade e no tipo de carro. O quadro de dados é cu.summary.
# Regression Tree Example
library(rpart)
# grow tree
fit <- rpart(Mileage~Price + Country + Reliability + Type,
method="anova", data=cu.summary)
printcp(fit) # display the results
plotcp(fit) # visualize cross-validation results
summary(fit) # detailed summary of splits
# create additional plots
par(mfrow=c(1,2)) # two plots on one page
rsq.rpart(fit) # visualize cross-validation results
# plot tree
plot(fit, uniform=TRUE,
main="Regression Tree for Mileage ")
text(fit, use.n=TRUE, all=TRUE, cex=.8)
# create attractive postcript plot of tree
post(fit, file = "c:/tree2.ps",
title = "Regression Tree for Mileage ")
# prune the tree
pfit<- prune(fit, cp=0.01160389) # from cptable
# plot the pruned tree
plot(pfit, uniform=TRUE,
main="Pruned Regression Tree for Mileage")
text(pfit, use.n=TRUE, all=TRUE, cex=.8)
post(pfit, file = "c:/ptree2.ps",
title = "Pruned Regression Tree for Mileage")Acontece que isso produz a mesma árvore que a original.
Árvores de inferência condicional via parte
O pacote party fornece árvores de regressão não paramétricas para respostas nominais, ordinais, numéricas, censuradas e multivariadas. party: Um laboratório para particionamento recursivo</a >, fornece detalhes.
Você pode criar uma árvore de regressão ou classificação por meio da função
ctree(formula, data=)O tipo de árvore criada dependerá da variável de resultado (fator nominal, fator ordenado, numérico etc.). O crescimento da árvore é baseado em regras de parada estatística, portanto, a poda não deve ser necessária.
Os dois exemplos anteriores são analisados novamente a seguir.
# Conditional Inference Tree for Kyphosis
library(party)
fit <- ctree(Kyphosis ~ Age + Number + Start,
data=kyphosis)
plot(fit, main="Conditional Inference Tree for Kyphosis")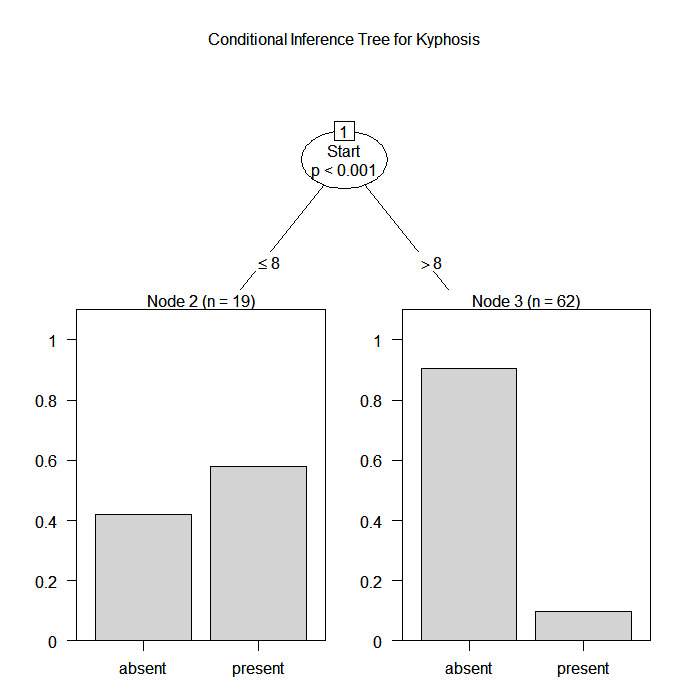
# Conditional Inference Tree for Mileage
library(party)
fit2 <- ctree(Mileage~Price + Country + Reliability + Type,
data=na.omit(cu.summary))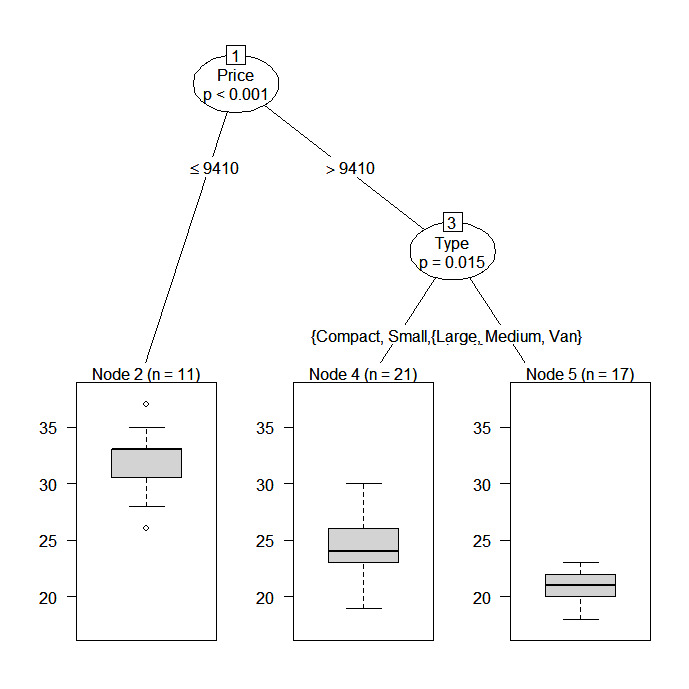
Florestas aleatórias
As florestas aleatórias melhoram a precisão da previsão gerando um grande número de árvores bootstrapped (com base em amostras aleatórias de variáveis), classificando um caso usando cada árvore nessa nova "floresta" e decidindo um resultado final previsto combinando os resultados de todas as árvores (uma média na regressão, um voto majoritário na classificação). A abordagem de floresta aleatória de Breiman e Cutler é implementada por meio do pacote randomForest</a > .
Aqui está um exemplo.
# Random Forest prediction of Kyphosis data
library(randomForest)
fit <- randomForest(Kyphosis ~ Age + Number + Start, data=kyphosis)
print(fit) # view results
importance(fit) # importance of each predictorPara obter mais detalhes, consulte o site abrangente do Random Forest</a > .
Indo além
Esta seção abordou apenas as opções disponíveis. Para saber mais, consulte o CRAN Task View on Machine & Statistical Learning</a > .
Para praticar
Experimente o curso Supervised Learning in R</a > que inclui um exercício com Random Forests .