Modèles arborescents en R
Le partitionnement récursif est un outil fondamental dans l'exploration des données. Il nous aide à explorer la structure d'un ensemble de données, tout en développant des règles de décision faciles à visualiser pour prédire un résultat catégorique (arbre de classification) ou continu (arbre de régression). Cette section décrit brièvement la modélisation CART, les arbres d'inférence conditionnelle et les forêts aléatoires.
Modélisation CART via rpart
Les arbres de classification et de régression (tels que décrits par Brieman, Freidman, Olshen et Stone) peuvent être générés à l'aide du logiciel rpart. Vous trouverez des informations détaillées sur rpart à l'adresse An Introduction to Recursive Partitioning Using the RPART Routines</a >. Les étapes générales sont présentées ci-dessous, suivies de deux exemples.
1. Faites pousser l'arbre
Pour faire pousser un arbre,userpart(formula, data=, method=,control=) où
| formule | est au format résultat ~ prédicteur1+prédicteur2+prédicteur3+ect. |
| data= | spécifie le cadre de données |
| method= | "classe" pour un arbre de classification"anova"</strong > pour un arbre de régression |
| control= | paramètres optionnels pour le contrôle de la croissance des arbres. Par exemple, control=rpart.control(minsplit=30, cp=0.001) exige que le nombre minimum d'observations dans un nœud soit de 30 avant de tenter un fractionnement et qu'un fractionnement doit diminuer l'inadéquation globale d'un facteur de 0.001 (facteur de complexité des coûts) avant d'être tenté. |
2. Examiner les résultats
Les fonctions suivantes nous aident à examiner les résultats.
| printcp(fit) | afficher le tableau des cp |
| plotcp(fit) | tracer les résultats de la validation croisée |
| rsq.rpart(fit) | tracer le R-carré approximatif et l'erreur relative pour différentes divisions (2 tracés). les étiquettes ne sont appropriées que pour la méthode "anova". |
| print(fit) | imprimer les résultats |
| résumé(fit) | des résultats détaillés, y compris des splits de substitution |
| tracer(fit) | tracer l'arbre de décision |
| text(fit) | étiqueter le diagramme de l'arbre de décision |
| post(fit, file=) | créer un graphique postscript de l'arbre de décision |
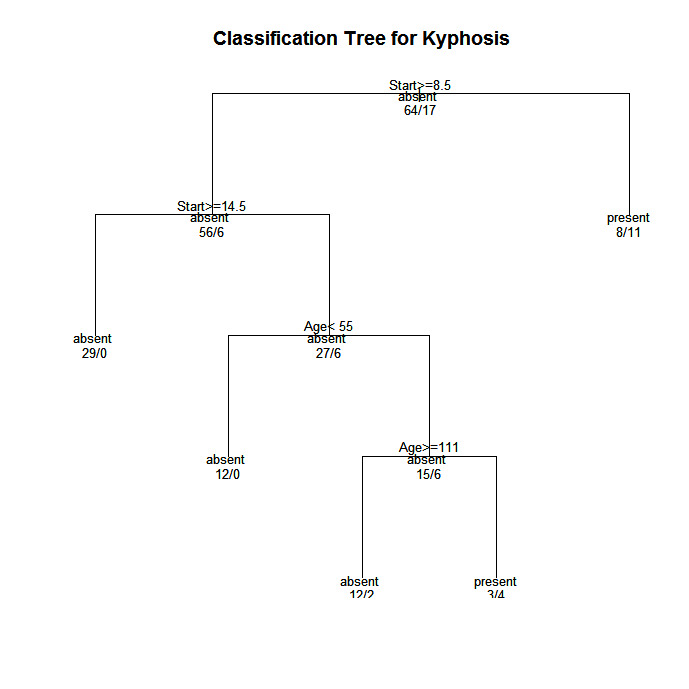
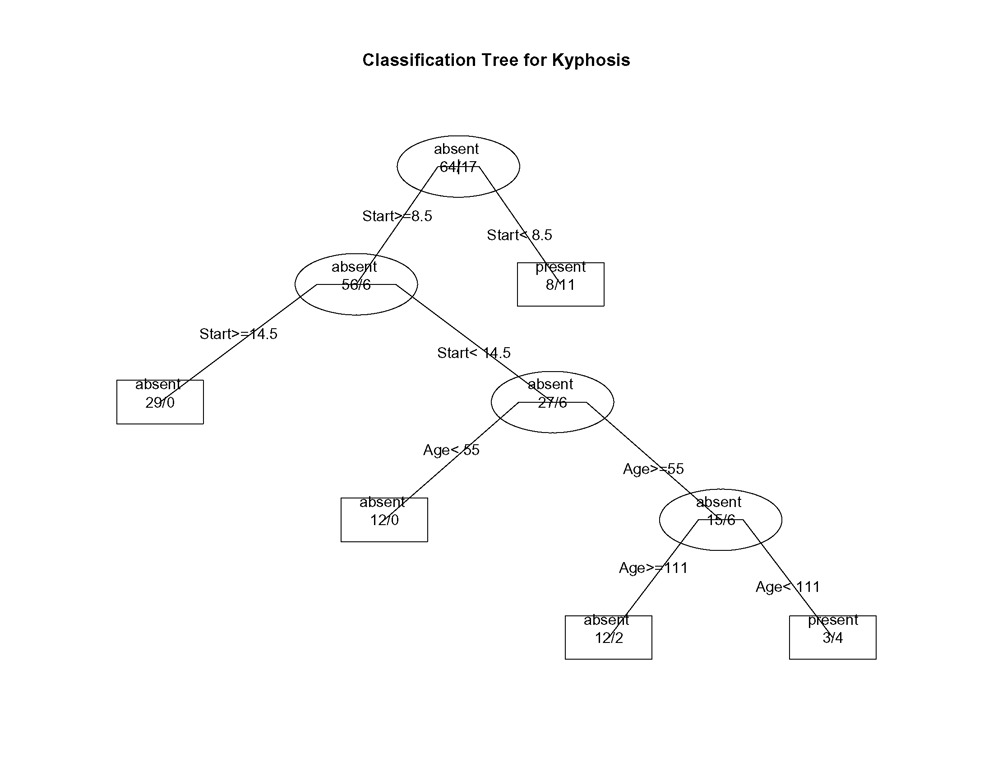
Dans les arbres créés par rpart( ), vous passez à la branche GAUCHE lorsque la condition énoncée est vraie (voir les graphiques ci-dessous).
3. élaguer l'arbre
Élaguez l'arbre pour éviter qu'il ne s'adapte trop aux données. En règle générale, vous voudrez sélectionner une taille d'arbre qui minimise l'erreur de validation croisée, la colonne xerror imprimée par printcp( ).
Élaguez l'arbre à la taille souhaitée en utilisantprune(fit, cp= )
Plus précisément, utilisez printcp( ) pour examiner les résultats de la validation croisée, sélectionnez le paramètre de complexité associé à l'erreur minimale et placez-le dans la fonction prune( ). Vous pouvez également utiliser le fragment de code suivant
fit$cptable[which.min(fit$cptable[, "xerror"]), "CP"]</strong >
pour sélectionner automatiquement le paramètre de complexité associé à la plus petite erreur de validation croisée. Merci à HSAUR pour cette idée.
Exemple d'arbre de classification
Utilisons le cadre de données cyphose pour prédire un type de déformation (cyphose) après une intervention chirurgicale, à partir de l'âge en mois (Age), du nombre de vertèbres concernées (Number), et de la vertèbre la plus haute opérée (Start).
# Classification Tree with rpart
library(rpart)
# grow tree
fit <- rpart(Kyphosis ~ Age + Number + Start,
method="class", data=kyphosis)
printcp(fit) # display the results
plotcp(fit) # visualize cross-validation results
summary(fit) # detailed summary of splits
# plot tree
plot(fit, uniform=TRUE,
main="Classification Tree for Kyphosis")
text(fit, use.n=TRUE, all=TRUE, cex=.8)
# create attractive postscript plot of tree
post(fit, file = "c:/tree.ps",
title = "Classification Tree for Kyphosis")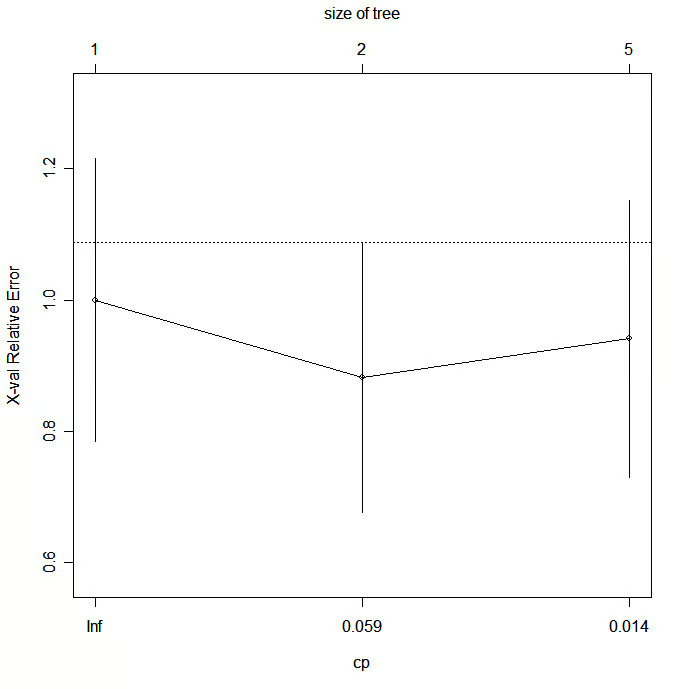
# prune the tree
pfit<- prune(fit, cp=
fit$cptable[which.min(fit$cptable[,"xerror"]),"CP"])
# plot the pruned tree
plot(pfit, uniform=TRUE,
main="Pruned Classification Tree for Kyphosis")
text(pfit, use.n=TRUE, all=TRUE, cex=.8)
post(pfit, file = "c:/ptree.ps",
title = "Pruned Classification Tree for Kyphosis")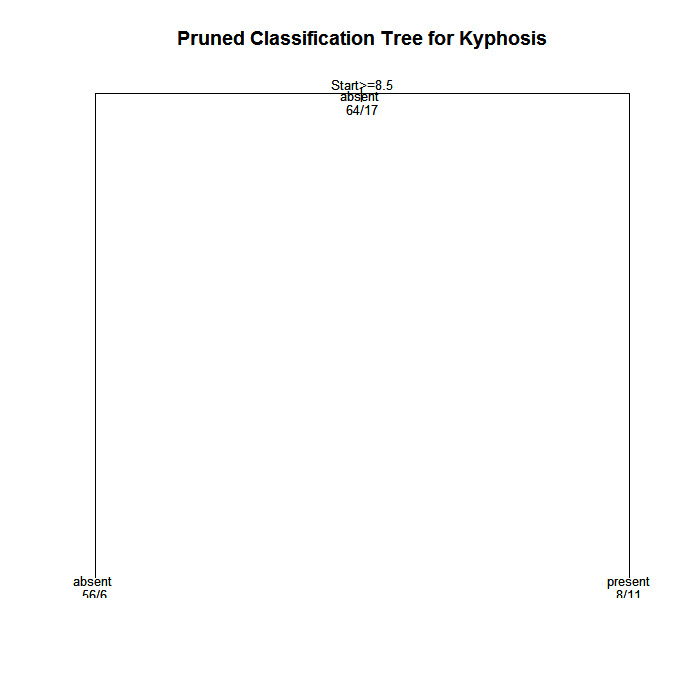

Exemple d'arbre de régression
Dans cet exemple, nous allons prédire le kilométrage d'une voiture à partir du prix, du pays, de la fiabilité et du type de voiture. La base de données est cu.summary.
# Regression Tree Example
library(rpart)
# grow tree
fit <- rpart(Mileage~Price + Country + Reliability + Type,
method="anova", data=cu.summary)
printcp(fit) # display the results
plotcp(fit) # visualize cross-validation results
summary(fit) # detailed summary of splits
# create additional plots
par(mfrow=c(1,2)) # two plots on one page
rsq.rpart(fit) # visualize cross-validation results
# plot tree
plot(fit, uniform=TRUE,
main="Regression Tree for Mileage ")
text(fit, use.n=TRUE, all=TRUE, cex=.8)
# create attractive postcript plot of tree
post(fit, file = "c:/tree2.ps",
title = "Regression Tree for Mileage ")
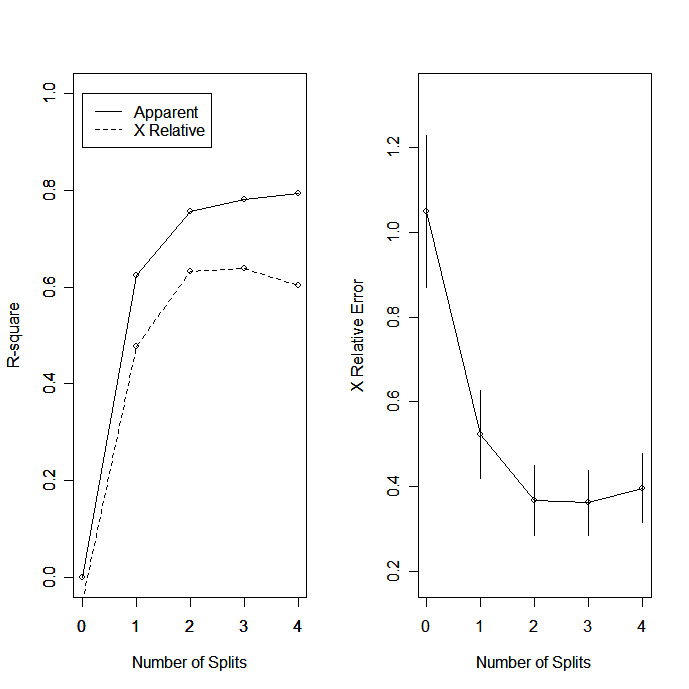
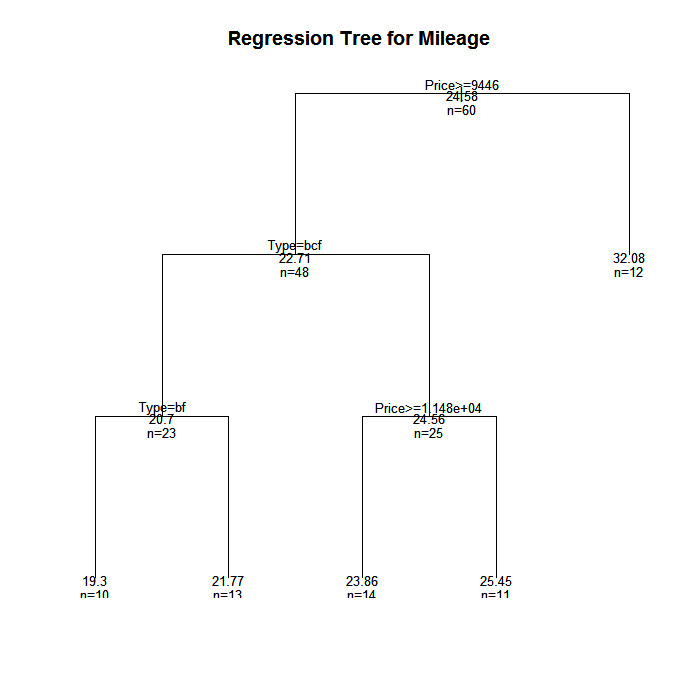
# prune the tree
pfit<- prune(fit, cp=0.01160389) # from cptable
# plot the pruned tree
plot(pfit, uniform=TRUE,
main="Pruned Regression Tree for Mileage")
text(pfit, use.n=TRUE, all=TRUE, cex=.8)
post(pfit, file = "c:/ptree2.ps",
title = "Pruned Regression Tree for Mileage")Il s'avère que cela produit le même arbre que l'original.
Arbres d'inférence conditionnelle par partie
Le paquetage party fournit des arbres de régression non paramétriques pour des réponses nominales, ordinales, numériques, censurées et multivariées. party : Un laboratoire pour le partitionnement récursif</a >, fournit des détails.
Vous pouvez créer un arbre de régression ou de classification à l'aide de la fonction
ctree(formula, data=)Le type d'arbre créé dépend de la variable de résultat (facteur nominal, facteur ordonné, numérique, etc.). La croissance des arbres est basée sur des règles d'arrêt statistique, de sorte que l'élagage ne devrait pas être nécessaire.
Les deux exemples précédents sont analysés à nouveau ci-dessous.
# Conditional Inference Tree for Kyphosis
library(party)
fit <- ctree(Kyphosis ~ Age + Number + Start,
data=kyphosis)
plot(fit, main="Conditional Inference Tree for Kyphosis")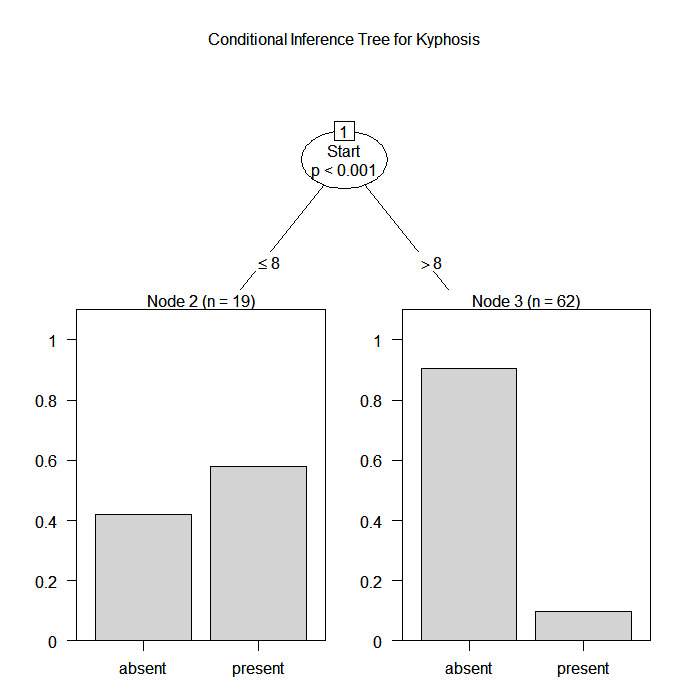
# Conditional Inference Tree for Mileage
library(party)
fit2 <- ctree(Mileage~Price + Country + Reliability + Type,
data=na.omit(cu.summary))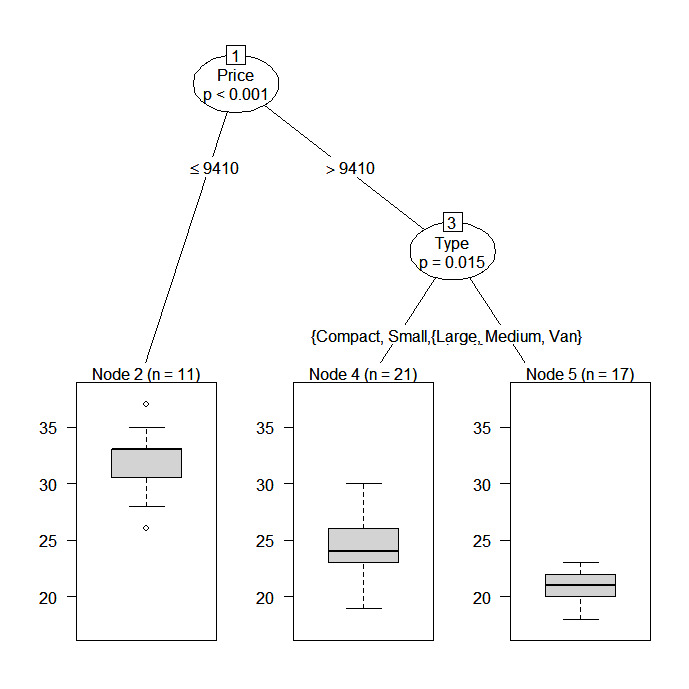
Forêts aléatoires
Les forêts aléatoires améliorent la précision des prédictions en générant un grand nombre d'arbres bootstrapés (basés sur des échantillons aléatoires de variables), en classant un cas à l'aide de chaque arbre de cette nouvelle "forêt" et en décidant d'un résultat final prédit en combinant les résultats de tous les arbres (une moyenne dans la régression, un vote majoritaire dans la classification). L'approche de la forêt aléatoire de Breiman et Cutler est mise en œuvre via le paquet randomForest</a >.
En voici un exemple.
# Random Forest prediction of Kyphosis data
library(randomForest)
fit <- randomForest(Kyphosis ~ Age + Number + Start, data=kyphosis)
print(fit) # view results
importance(fit) # importance of each predictorPour plus de détails, consultez le site Web complet de Random Forest</a >.
Aller plus loin
Cette section n'a fait qu'effleurer les options disponibles. Pour en savoir plus, consultez le CRAN Task View on Machine & Statistical Learning</a >.
Pratiquer
Essayez le cours Apprentissage supervisé en R</a > qui comprend un exercice sur les forêts aléatoires .