Analyse des fonctions discriminantes en R
Le progiciel MASS contient des fonctions permettant d'effectuer des analyses de fonctions discriminantes linéaires et quadratiques. À moins que des probabilités préalables ne soient spécifiées, chacun suppose des probabilités préalables proportionnelles (c'est-à-dire que les probabilités préalables sont basées sur la taille des échantillons). Dans les exemples ci-dessous, les lettres minuscules sont des variables numériques et les lettres majuscules sont des facteurs catégoriels.
Fonction discriminante linéaire
# Linear Discriminant Analysis with Jacknifed Prediction
library(MASS)
fit <- lda(G ~ x1 + x2 + x3, data=mydata,
na.action="na.omit", CV=TRUE)
fit # show resultsLe code ci-dessus effectue une analyse linéaire des données, en supprimant les données manquantes dans le sens de la liste. CV=TRUE génère des prédictions jacknifiées (c.-à-d. sans tenir compte d'un seul élément). Le code ci-dessous évalue la précision de la prédiction.
# Assess the accuracy of the prediction
# percent correct for each category of G
ct <- table(mydata$G, fit$class)
diag(prop.table(ct, 1))
# total percent correct
sum(diag(prop.table(ct)))lda() imprime les fonctions discriminantes basées sur des variables centrées (non standardisées). La "proportion de trace" qui est imprimée est la proportion de variance entre les classes qui est expliquée par les fonctions discriminantes successives. Aucun test de signification n'est produit. Reportez-vous à la section sur la MANOVA pour de tels tests.
Fonction discriminante quadratique
Pour obtenir une fonction discriminante quadratique, utilisez qda( ) au lieu de lda( ). La fonction discriminante quadratique ne suppose pas l'homogénéité des matrices de variance-covariance.
# Quadratic Discriminant Analysis with 3 groups applying
#
resubstitution prediction and equal prior probabilities.
library(MASS)
fit <- qda(G ~ x1 + x2 + x3 + x4, data=na.omit(mydata),
prior=c(1,1,1)/3))Notez l'autre façon de spécifier la suppression par liste des données manquantes. La resubstitution (utilisation des mêmes données pour dériver les fonctions et évaluer la précision de leur prédiction) est la méthode par défaut, sauf si CV=TRUE est spécifié. La resubstitution sera trop optimiste.
Visualisation des résultats
Vous pouvez représenter chaque observation dans l'espace des deux premières fonctions discriminantes linéaires à l'aide du code suivant. Les points sont identifiés par l'ID du groupe.
# Scatter plot using the 1st two discriminant dimensions
plot(fit) # fit from lda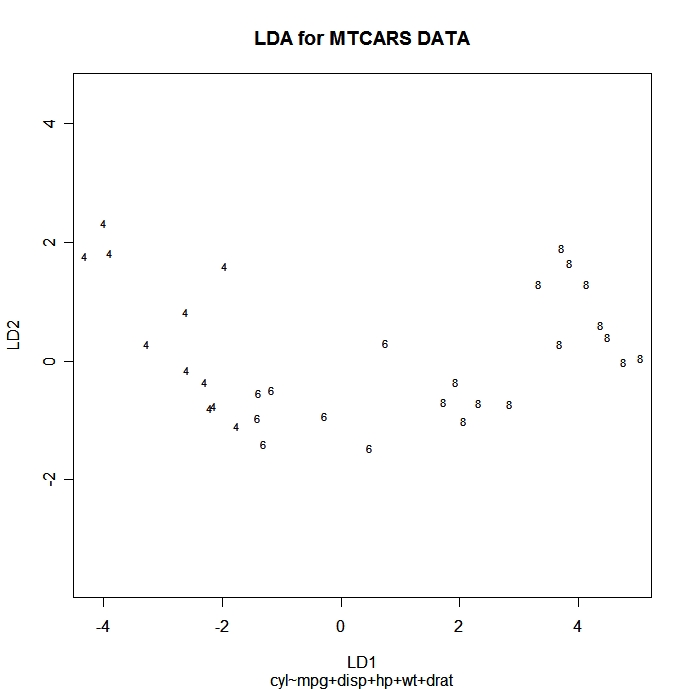
Le code suivant affiche des histogrammes et des diagrammes de densité pour les observations de chaque groupe sur la première dimension du discriminant linéaire. Il y a un panneau pour chaque groupe et ils apparaissent tous alignés sur le même graphique.
# Panels of histograms and overlayed density plots
# for 1st discriminant function
plot(fit, dimen=1, type="both") # fit from lda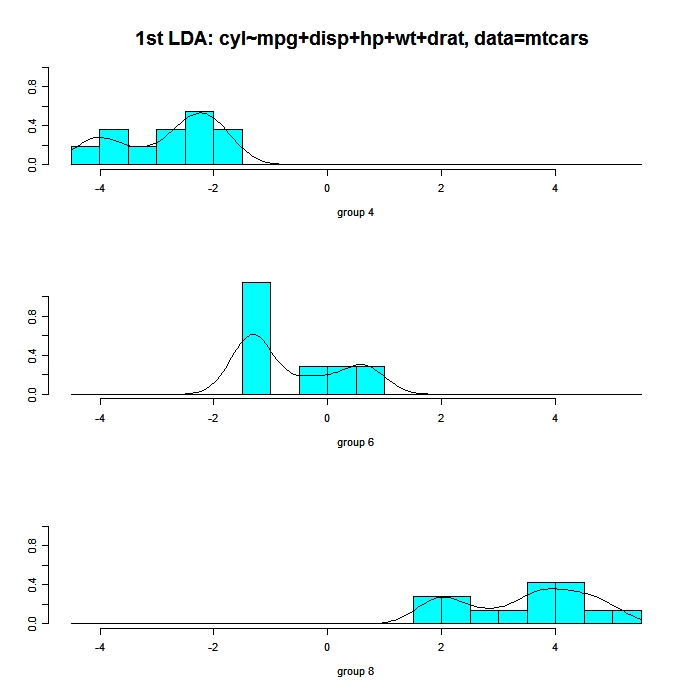
La fonction partimat( ) du package klaR permet d'afficher les résultats d'une classification linéaire ou quadratique 2 variables à la fois.
# Exploratory Graph for LDA or QDA
library(klaR)
partimat(G~x1+x2+x3,data=mydata,method="lda")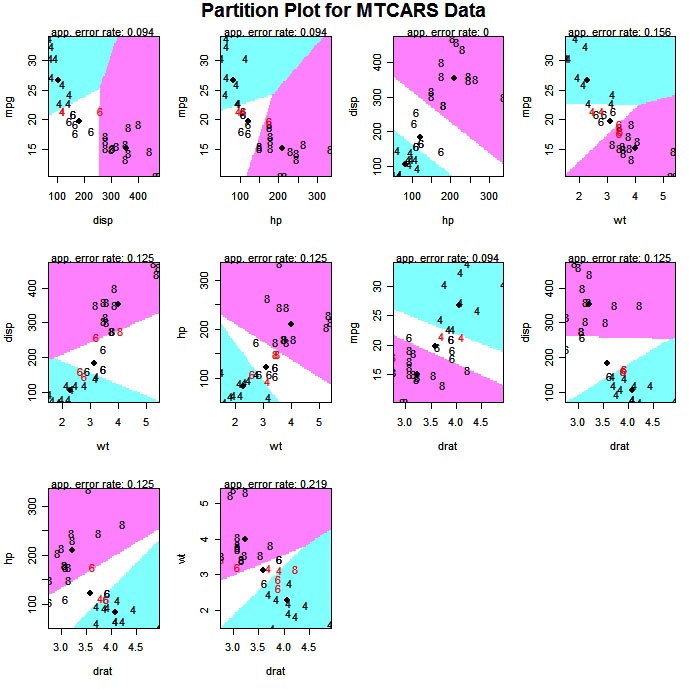
Vous pouvez également produire une matrice de nuage de points avec un code couleur par groupe.
# Scatterplot for 3 Group Problem
pairs(mydata[c("x1","x2","x3")], main="My Title ", pch=22,
bg=c("red", "yellow", "blue")[unclass(mydata$G)])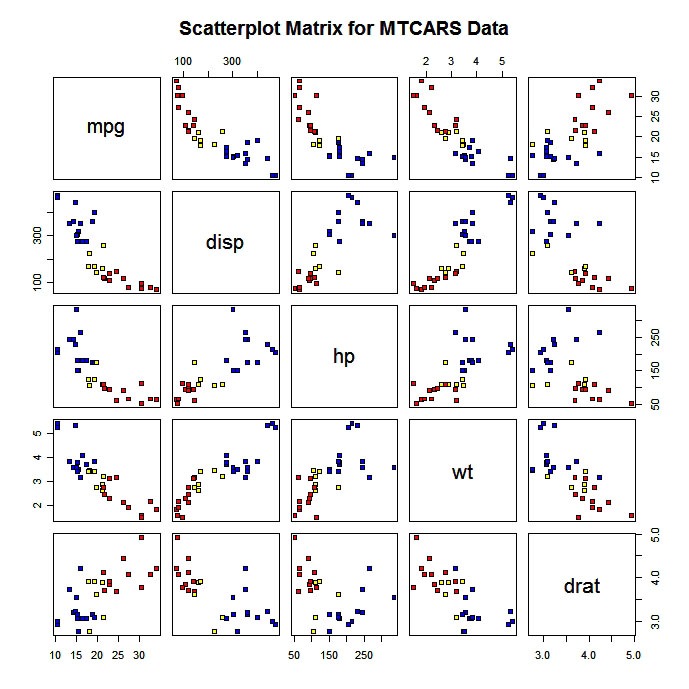
Hypothèses de test
Voir (M)ANOVA Assumptionspour lesméthodes d'évaluation de la normalité multivariée et de l'homogénéité des matrices de covariance.
Pratiquer
Pour vous entraîner à améliorer les prédictions, essayez le cours Apprentissage supervisé en R</a >