Évaluer les hypothèses des tests classiques dans R
Dans les procédures paramétriques classiques, nous supposons souvent la normalité et une variance constante pour le terme d'erreur du modèle. Les méthodes d'exploration de ces hypothèses dans un cadre ANOVA/ANCOVA/MANOVA sont discutées ici. Les diagnostics de régression sont couverts par la régression linéaire multiple.
Valeurs aberrantes
Les valeurs aberrantes pouvant affecter gravement la normalité et l'homogénéité de la variance, les méthodes de détection des observations disparates sont décrites en premier lieu.
# Detect Outliers in the MTCARS Data
library(mvoutlier)
outliers <-
aq.plot(mtcars[c("mpg","disp","hp","drat","wt","qsec")])
outliers # show list of outliers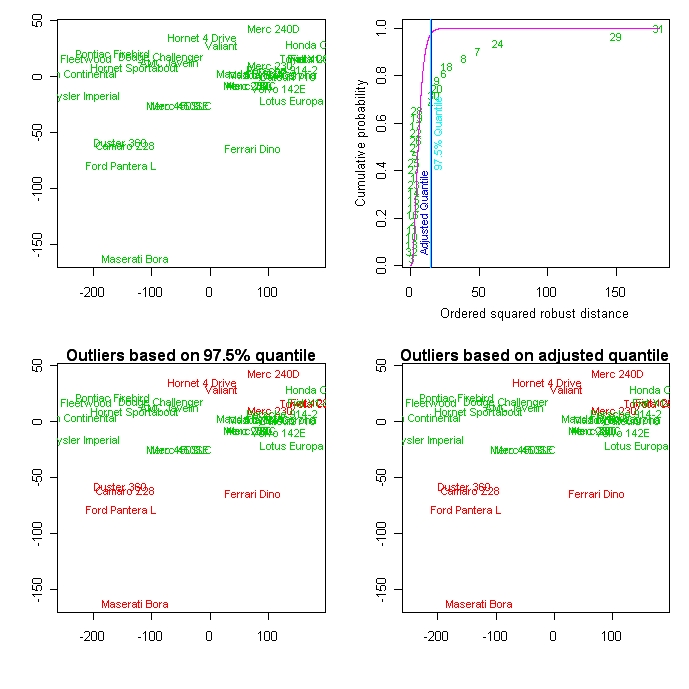
Normalité univariée
Vous pouvez évaluer la normalité d'une variable à l'aide d'un graphique Q-Q.
# Q-Q Plot for variable MPG
attach(mtcars)
qqnorm(mpg)
qqline(mpg)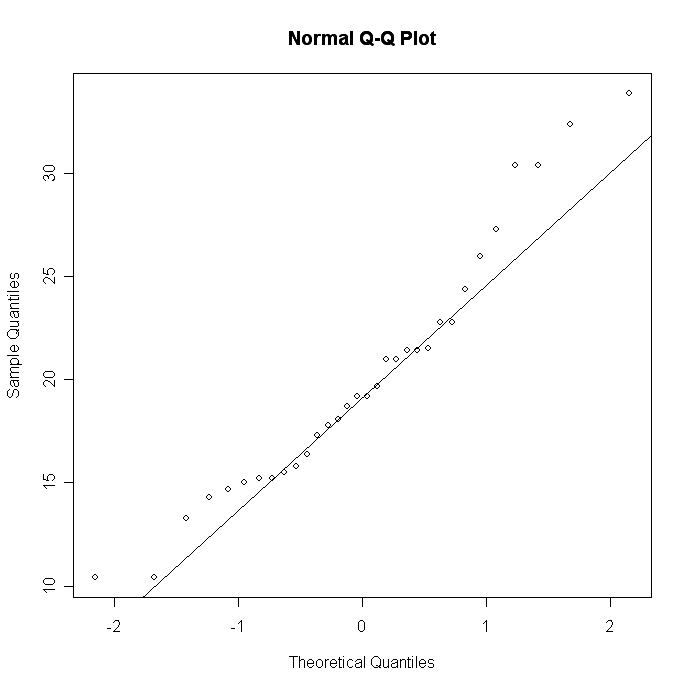
Des écarts importants par rapport à la ligne suggèrent des violations de la normalité.
Vous pouvez également effectuer un test de normalité de Shapiro-Wilk avec la fonction shapiro.test(x), où x est un tableau numérique. Des fonctions supplémentaires pour tester la normalité sont disponibles dans le paquet nortest</a >.
Normalité multivariée
La MANOVA suppose une normalité multivariée. La fonction mshapiro.test( ) du paquet mvnormtest</a > produit le test de Shapiro-Wilk pour la normalité multivariée. L'entrée doit être une matrice numérique.
# Test Multivariate Normality
mshapiro.test(M)Si nous avons un vecteur aléatoire normal multivarié p x 1, la distance de Mahalanobis au carré entre x et μ sera distribuée en chi-carré avec p degrés de liberté. Nous pouvons utiliser ce fait pour construire un graphique Q-Q afin d'évaluer la normalité multivariée.
# Graphical Assessment of Multivariate Normality
x <- as.matrix(mydata) # n x p numeric matrix
center <- colMeans(x) # centroid
n <- nrow(x); p <- ncol(x); cov <- cov(x);
d <-
mahalanobis(x,center,cov) # distances
qqplot(qchisq(ppoints(n),df=p),d,
main="QQ Plot Assessing Multivariate Normality",
ylab="Mahalanobis D2")
abline(a=0,b=1)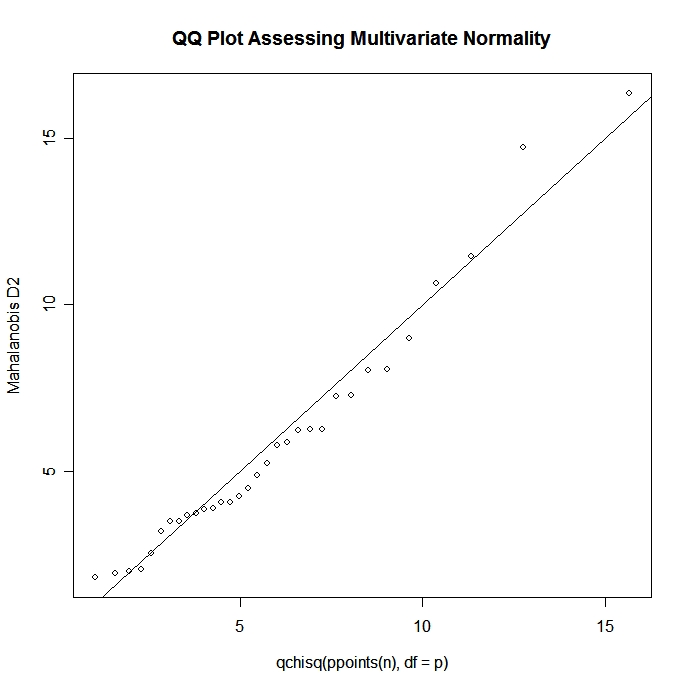
Homogénéité des variances
La fonction bartlett.test( ) fournit un test paramétrique d'échantillon K de l'égalité des variances. La fonction fligner.test( ) permet d'effectuer un test non paramétrique de la même manière. Dans les exemples suivants, y est une variable numérique et G est la variable de regroupement.
# Bartlett Test of Homogeneity of Variances
bartlett.test(y~G, data=mydata)
# Figner-Killeen Test of Homogeneity of Variances
fligner.test(y~G, data=mydata)La fonction hovPlot( ) du paquet HH fournit un test graphique de l'homogénéité des variances basé sur Brown-Forsyth. Dans l'exemple suivant, y est numérique et G est un facteur de regroupement. Notez que G doit être de type facteur.
# Homogeneity of Variance Plot
library(HH)
hov(y~G, data=mydata)
hovPlot(y~G,data=mydata)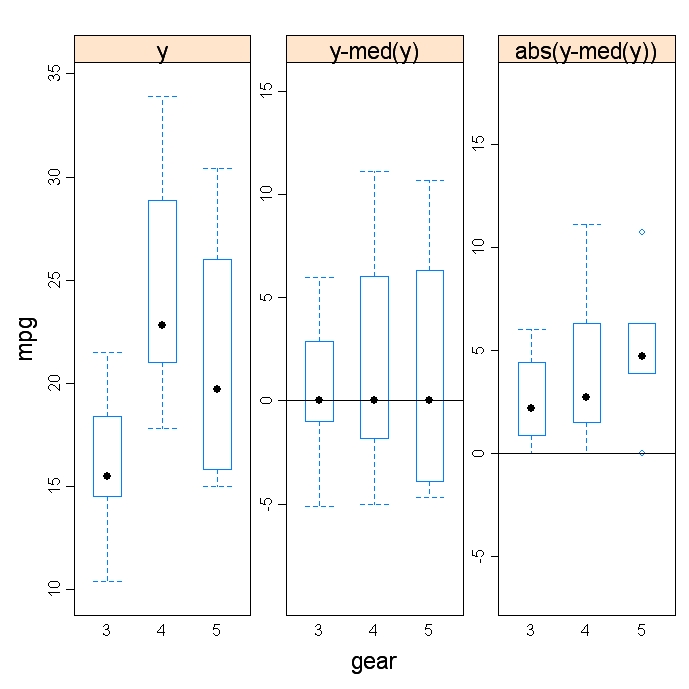
Homogénéité des matrices de covariance
MANOVA et LDF supposent l'homogénéité des matrices de variance-covariance. L'hypothèse est généralement testée avec le M de Box. Malheureusement, le test est très sensible aux violations de la normalité, ce qui conduit à un rejet dans la plupart des cas typiques. Le M de Box est disponible via la fonction boxM dans le paquet biotools</a >.