Régression multiple (linéaire) dans R
R offre un support complet pour la régression linéaire multiple. Les thèmes ci-dessous sont présentés par ordre de complexité croissante.
Ajustement du modèle
# Multiple Linear Regression Example
fit <- lm(y ~ x1 + x2 + x3, data=mydata)
summary(fit) # show results# Other useful functions
coefficients(fit) # model coefficients
confint(fit, level=0.95) # CIs for model parameters
fitted(fit) # predicted values
residuals(fit) # residuals
anova(fit) # anova table
vcov(fit) # covariance matrix for model parameters
influence(fit) # regression diagnosticsPlots de diagnostic
Les graphiques de diagnostic permettent de vérifier l'hétéroscédasticité, la normalité et les observations influentes.
# diagnostic plots
layout(matrix(c(1,2,3,4),2,2)) # optional 4 graphs/page
plot(fit)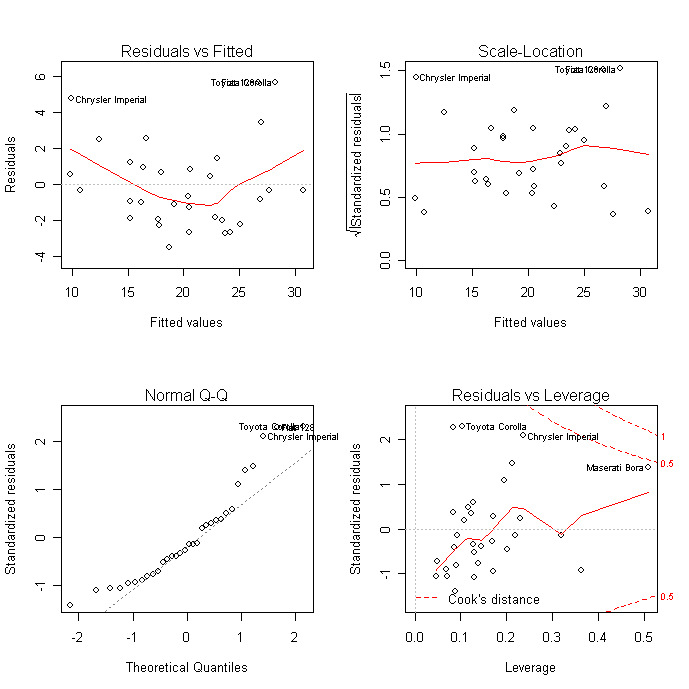
Pour une évaluation plus complète de l'adéquation du modèle, consultez les diagnostics de régression ou les exercices de ce cours interactif</a > sur l'apprentissage supervisé en R : Régression.
Comparaison des modèles
Vous pouvez comparer des modèles imbriqués à l'aide de la fonction anova( ). Le code suivant permet de tester simultanément que x3 et x4 ajoutent à la prédiction linéaire en plus de x1 et x2.
# compare models
fit1 <- lm(y ~ x1 + x2 + x3 + x4, data=mydata)
fit2 <- lm(y ~ x1 + x2)
anova(fit1, fit2)Validation croisée
Vous pouvez effectuer une validation croisée K-Fold</a > en utilisant la fonction cv.lm( ) dans le package DAAG.
# K-fold cross-validation
library(DAAG)
cv.lm(df=mydata, fit, m=3) # 3 fold cross-validationAdditionnez l'EQM pour chaque pli, divisez-la par le nombre d'observations et prenez la racine carrée pour obtenir l'erreur standard d'estimation validée croisée.
Vous pouvez évaluer le rétrécissement de R2 au moyen de la validation croisée K-fold. En utilisant la fonction crossval() du paquetage bootstrap</a ></strong >, procédez comme suit :
# Assessing R2 shrinkage using 10-Fold Cross-Validation
fit <- lm(y~x1+x2+x3,data=mydata)
library(bootstrap)
# define functions
theta.fit <- function(x,y){lsfit(x,y)}
theta.predict <- function(fit,x){cbind(1,x)%*%fit$coef}
# matrix of predictors
X
<- as.matrix(mydata[c("x1","x2","x3")])
# vector of predicted values
y <- as.matrix(mydata[c("y")])
results <- crossval(X,y,theta.fit,theta.predict,ngroup=10)
cor(y, fit$fitted.values)**2 # raw R2
cor(y,results$cv.fit)**2 # cross-validated R2Sélection des variables
La sélection d'un sous-ensemble de variables prédictives à partir d'un ensemble plus large (par exemple, la sélection par étapes) est un sujet controversé. Vous pouvez effectuer une sélection par étapes (vers l'avant, vers l'arrière, les deux) à l'aide de la fonction stepAIC( ) du progiciel MASS. stepAIC( ) effectue une sélection de modèle par étapes par AIC exact.
# Stepwise Regression
library(MASS)
fit <- lm(y~x1+x2+x3,data=mydata)
step <- stepAIC(fit, direction="both")
step$anova # display resultsVous pouvez également effectuer une régression sur tous les sous-ensembles à l'aide de la fonction leaps( ) du package leaps. Dans le code suivant, nbest indique le nombre de sous-ensembles de chaque taille à déclarer. Ici, les dix meilleurs modèles seront présentés pour chaque taille de sous-ensemble (1 prédicteur, 2 prédicteurs, etc.).
# All Subsets Regression
library(leaps)
attach(mydata)
leaps<-regsubsets(y~x1+x2+x3+x4,data=mydata,nbest=10)
# view results
summary(leaps)
# plot a table of models showing variables in each model.
#
models are ordered by the selection statistic.
plot(leaps,scale="r2")
# plot statistic by subset size
library(car)
subsets(leaps, statistic="rsq")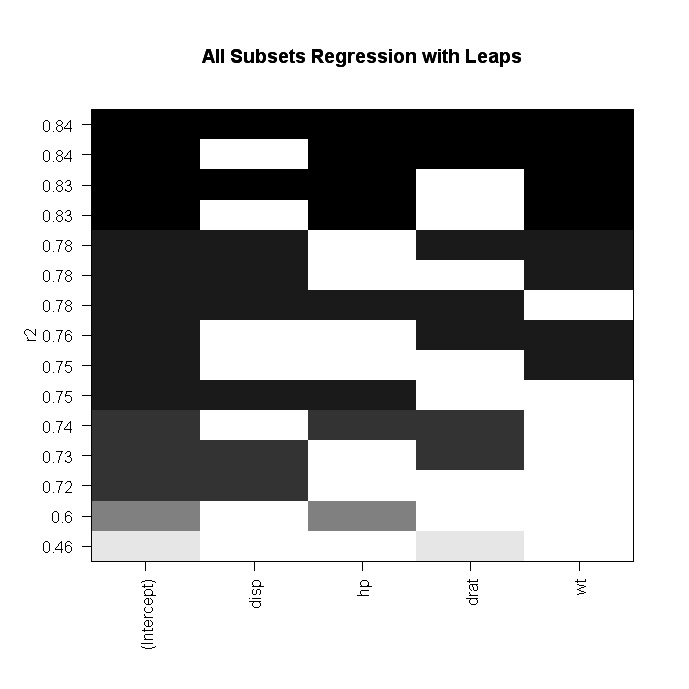
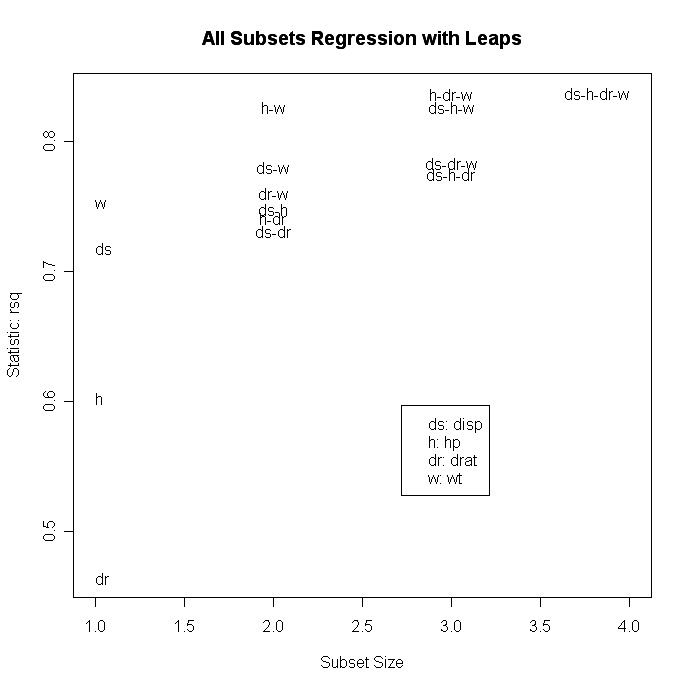
Les autres options de plot( ) sont bic, Cp et adjr2. Les autres options de tracé avecsubset( ) sont bic, cp, adjr2 et rss.
Importance relative
# Calculate Relative Importance for Each Predictor
library(relaimpo)
calc.relimp(fit,type=c("lmg","last","first","pratt"),
rela=TRUE)
# Bootstrap Measures of Relative Importance (1000 samples)
boot <- boot.relimp(fit, b = 1000, type = c("lmg",
"last", "first", "pratt"), rank = TRUE,
diff = TRUE, rela = TRUE)
booteval.relimp(boot) # print result
plot(booteval.relimp(boot,sort=TRUE)) # plot result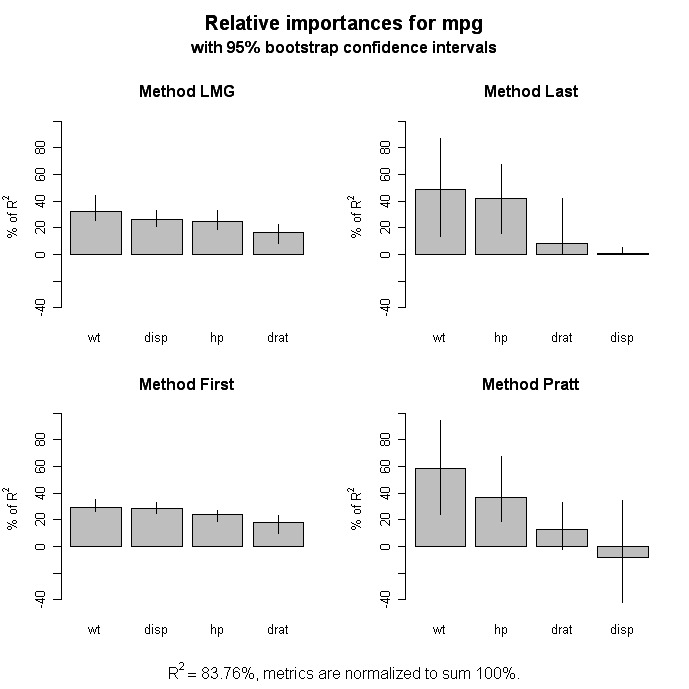
Améliorations graphiques
Le package car offre une grande variété de tracés pour la régression, y compris des tracés à variables ajoutées, et des diagrammes de diagnostic et de dispersion améliorés.
Aller plus loin
Régression non linéaire
Le paquet nls fournit des fonctions pour la régression non linéaire. Voir le site de John Fox Nonlinear Regression and Nonlinear Least Squares</a > pour une vue d'ensemble. Huet et ses collègues Statistical Tools for Nonlinear Regression (Outils statistiques pour la régression non linéaire) : A Practical Guide with S-PLUS and R Examples</a > est un ouvrage de référence précieux.
Régression robuste
Il existe de nombreuses fonctions dans R pour faciliter la régression robuste. Par exemple, vous pouvez effectuer une régression robuste avec la fonction rlm( ) du package MASS. John Fox (qui d'autre ?) Robust Regression</a > offre une bonne vue d'ensemble. Le site Web de l'UCLA Statistical Computing propose Robust Regression Examples</a >.
Le package robust fournit une bibliothèque complète de méthodes robustes, y compris la régression. Le paquet robustbase</a > fournit également des statistiques robustes de base, y compris des méthodes de sélection de modèles. Et David Olive a fourni un examen en ligne détaillé de Applied Robust Statistics</a > avec un exemple de code R.