Analyse des clusters en R
R offre un large éventail de fonctions pour cluster analysis</a >, y compris des approches agglomératives hiérarchiques, de partitionnement et basées sur des modèles. Bien qu'il n'existe pas de solution définitive pour déterminer le nombre optimal de grappes à extraire, plusieurs approches sont disponibles.
Préparation des données
Avant de regrouper les données, vous pouvez supprimer ou estimer les données manquantes et rééchelonner les variables pour les rendre comparables.
# Prepare Data
mydata <- na.omit(mydata) # listwise deletion of missing
mydata <- scale(mydata) # standardize variablesCloisonnement
Le regroupement par K-moyennes est la méthode de partitionnement la plus répandue. L'analyste doit spécifier le nombre de grappes à extraire. Un graphique de la somme des carrés à l'intérieur des groupes en fonction du nombre de grappes extraites peut aider à déterminer le nombre approprié de grappes. L'analyste recherche une courbure dans le tracé, similaire à un test d'échantillonnage (scree test) dans l'analyse factorielle.
# Determine number of clusters
wss <- (nrow(mydata)-1)*sum(apply(mydata,2,var))
for (i in 2:15) wss[i] <- sum(kmeans(mydata,
centers=i)$withinss)
plot(1:15, wss, type="b", xlab="Number of Clusters",
ylab="Within groups sum of squares")# K-Means Cluster Analysis
fit <- kmeans(mydata, 5) # 5 cluster solution
# get cluster means
aggregate(mydata,by=list(fit$cluster),FUN=mean)
# append cluster assignment
mydata <- data.frame(mydata, fit$cluster)Une version robuste de K-means basée sur les médiodes peut être invoquée en utilisant pam( ) au lieu de kmeans( ). La fonction pamk( ) du paquet fpc</a ></strong > est une enveloppe pour pam qui imprime également le nombre de grappes suggéré sur la base de la largeur moyenne optimale de la silhouette.
Hiérarchique Agglomératif
Il existe un large éventail d'approches de regroupement hiérarchique. J'ai eu de la chance avec la méthode de Ward décrite ci-dessous.
# Ward Hierarchical Clustering
d <- dist(mydata,
method = "euclidean") # distance matrix
fit <- hclust(d, method="ward")
plot(fit) # display dendogram
groups <- cutree(fit, k=5) # cut tree into 5 clusters
# draw dendogram with red borders around the 5 clusters
rect.hclust(fit, k=5, border="red")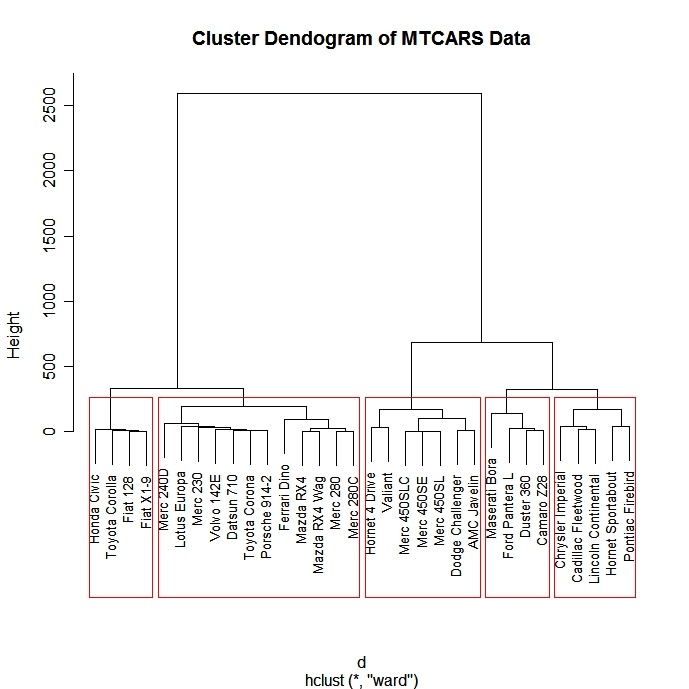
La fonction pvclust( ) du paquet pvclust</a > fournit des valeurs p pour le regroupement hiérarchique basé sur un rééchantillonnage bootstrap à plusieurs échelles. Les groupes qui sont fortement soutenus par les données auront des valeurs p élevées. Sachez que pvclust</a > regroupe les colonnes et non les lignes. Transposez vos données avant de les utiliser.
# Ward Hierarchical Clustering with Bootstrapped p values
library(pvclust)
fit <-
pvclust(mydata, method.hclust="ward",
method.dist="euclidean")
plot(fit) # dendogram with p values
# add rectangles around groups highly supported by the data
pvrect(fit, alpha=.95)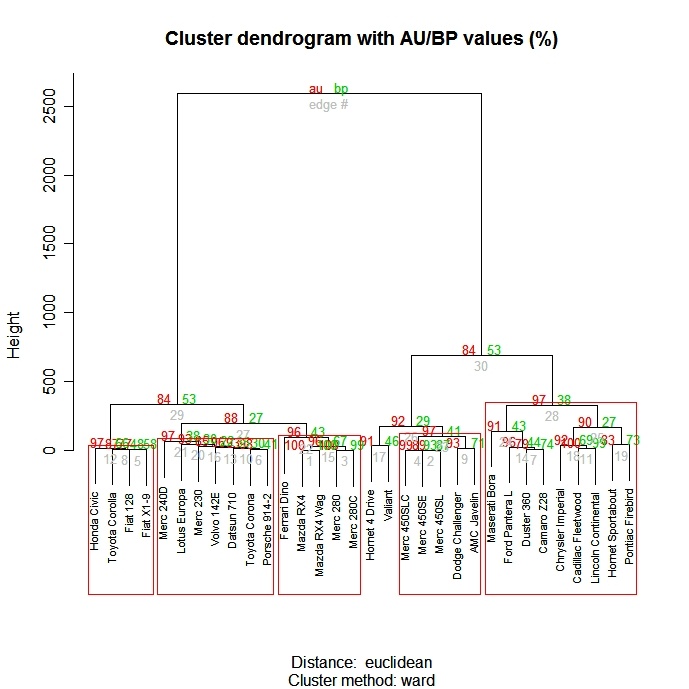
Sur la base d'un modèle
Les approches basées sur des modèles supposent une variété de modèles de données et appliquent l'estimation du maximum de vraisemblance et les critères de Bayes pour identifier le modèle le plus probable et le nombre de grappes. Plus précisément, la fonction Mclust( ) du package mclust sélectionne le modèle optimal en fonction du BIC pour EM initialisé par clustering hiérarchique pour les modèles de mélanges gaussiens paramétrés. (ouf !). On choisit le modèle et le nombre de grappes avec le plus grand BIC. Voir help(mclustModelNames)</a > pour plus de détails sur le modèle choisi comme étant le meilleur.
# Model Based Clustering
library(mclust)
fit <- Mclust(mydata)
plot(fit) # plot results
summary(fit) # display the best model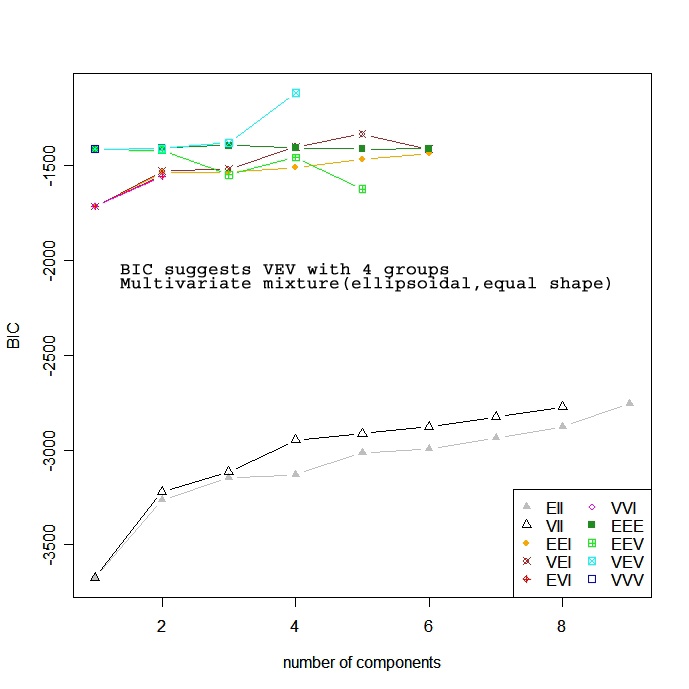
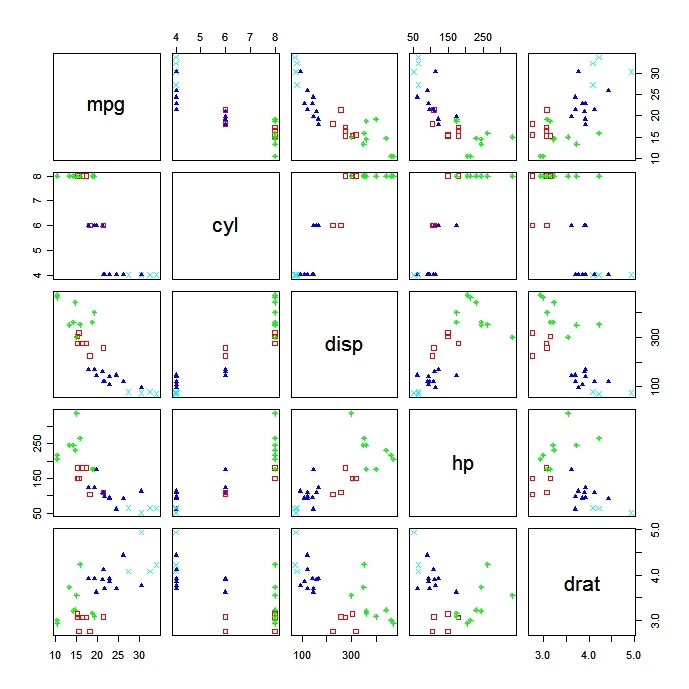
Tracer les solutions des grappes
Il est toujours utile d'examiner les résultats des grappes d'entreprises.
# K-Means Clustering with 5 clusters
fit <- kmeans(mydata, 5)
# Cluster Plot against 1st 2 principal components
# vary parameters for most readable graph
library(cluster)
clusplot(mydata, fit$cluster, color=TRUE, shade=TRUE,
labels=2, lines=0)
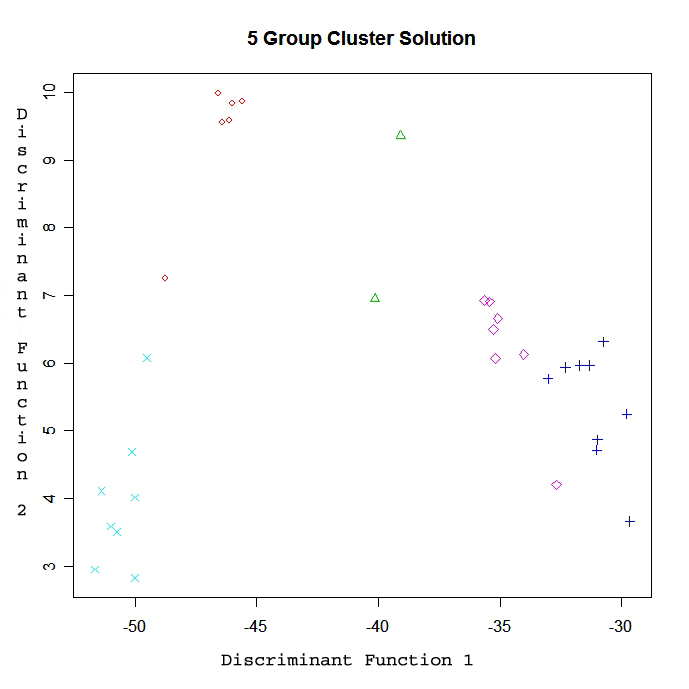
# Centroid Plot against 1st 2 discriminant functions
library(fpc)
plotcluster(mydata, fit$cluster)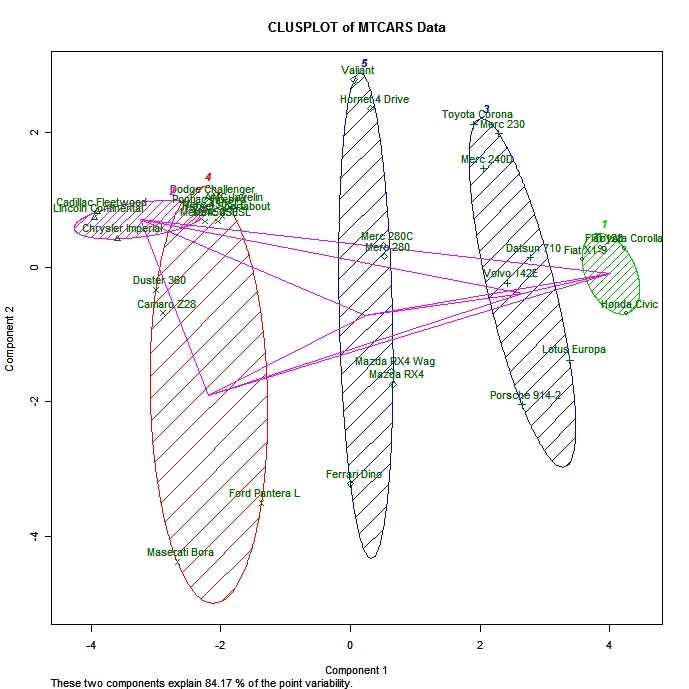
Valider les solutions en grappe
La fonction cluster.stats() du paquetage fpc fournit un mécanisme de comparaison de la similarité de deux solutions de cluster en utilisant une variété de critères de validation (le coefficient gamma de Hubert, l'indice de Dunn et l'indice rand corrigé)
# comparing 2 cluster solutions
library(fpc)
cluster.stats(d, fit1$cluster, fit2$cluster)où d est une matrice de distance entre les objets, et fit1$cluster et fit$cluste r sont des vecteurs entiers contenant les résultats de classification de deux regroupements différents des mêmes données.
Pratiquer
Essayez l'exercice de clustering dans ce cours d'introduction à l'apprentissage automatique.