Análise de cluster em R
O R oferece uma ampla variedade de funções para análise de cluster</a >, incluindo abordagens hierárquicas aglomerativas, de particionamento e baseadas em modelos. Embora não haja uma solução definitiva para determinar o número ideal de clusters a serem extraídos, há várias abordagens disponíveis.
Preparação de dados
Antes de agrupar os dados, talvez você queira remover ou estimar os dados ausentes e redimensionar as variáveis para fins de comparabilidade.
# Prepare Data
mydata <- na.omit(mydata) # listwise deletion of missing
mydata <- scale(mydata) # standardize variablesParticionamento
O clustering K-means é o método de particionamento mais popular. Ele exige que o analista especifique o número de clusters a serem extraídos. Um gráfico da soma de quadrados dentro dos grupos por número de clusters extraídos pode ajudar a determinar o número apropriado de clusters. O analista procura uma curva no gráfico semelhante a um teste scree na análise de fatores.
# Determine number of clusters
wss <- (nrow(mydata)-1)*sum(apply(mydata,2,var))
for (i in 2:15) wss[i] <- sum(kmeans(mydata,
centers=i)$withinss)
plot(1:15, wss, type="b", xlab="Number of Clusters",
ylab="Within groups sum of squares")# K-Means Cluster Analysis
fit <- kmeans(mydata, 5) # 5 cluster solution
# get cluster means
aggregate(mydata,by=list(fit$cluster),FUN=mean)
# append cluster assignment
mydata <- data.frame(mydata, fit$cluster)Uma versão robusta do K-means baseada em mediods pode ser invocada usando pam( ) em vez de kmeans( ). A função pamk( ) no pacote fpc</a ></strong > é um wrapper para pam que também imprime o número sugerido de clusters com base na largura média ideal da silhueta.
Aglomerativo hierárquico
Há uma grande variedade de abordagens de agrupamento hierárquico. Tive boa sorte com o método de Ward descrito abaixo.
# Ward Hierarchical Clustering
d <- dist(mydata,
method = "euclidean") # distance matrix
fit <- hclust(d, method="ward")
plot(fit) # display dendogram
groups <- cutree(fit, k=5) # cut tree into 5 clusters
# draw dendogram with red borders around the 5 clusters
rect.hclust(fit, k=5, border="red")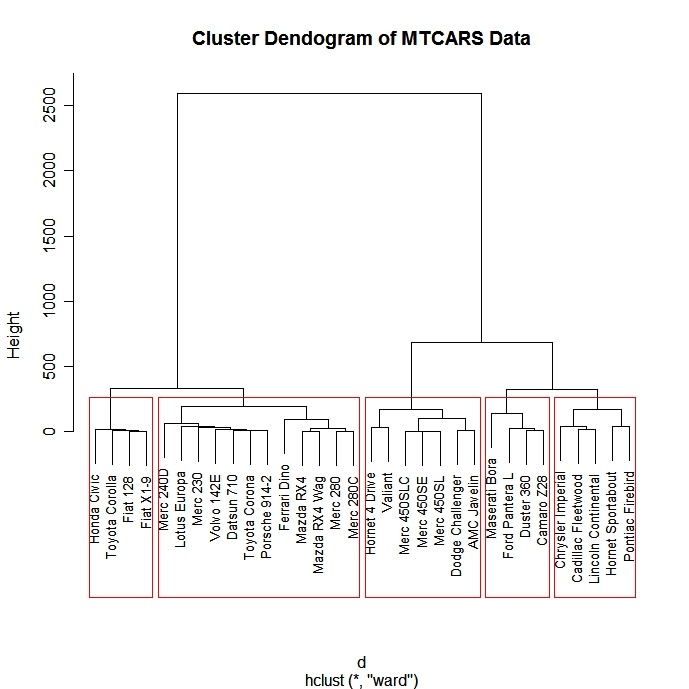
A função pvclust( ) do pacote pvclust</a > fornece valores de p para agrupamento hierárquico com base em reamostragem bootstrap multiescala. Os clusters que são altamente suportados pelos dados terão valores p grandes. Lembre-se de que pvclust</a > agrupa colunas, não linhas. Transponha seus dados antes de usá-los.
# Ward Hierarchical Clustering with Bootstrapped p values
library(pvclust)
fit <-
pvclust(mydata, method.hclust="ward",
method.dist="euclidean")
plot(fit) # dendogram with p values
# add rectangles around groups highly supported by the data
pvrect(fit, alpha=.95)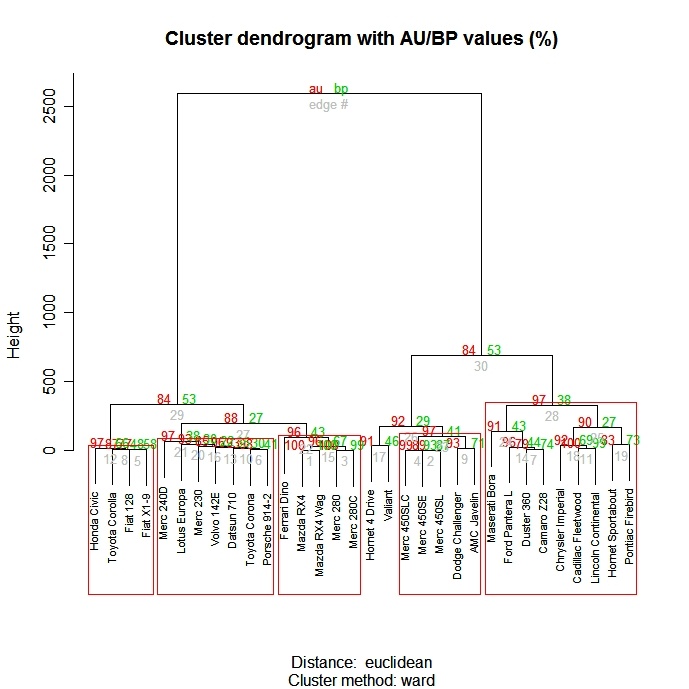
Baseado em modelos
As abordagens baseadas em modelos assumem uma variedade de modelos de dados e aplicam a estimativa de máxima verossimilhança e os critérios de Bayes para identificar o modelo mais provável e o número de clusters. Especificamente, a função Mclust( ) no pacote mclust seleciona o modelo ideal de acordo com o BIC para EM inicializado por agrupamento hierárquico para modelos de mistura gaussiana parametrizados. (ufa!). Você escolhe o modelo e o número de clusters com o maior BIC. Consulte help(mclustModelNames)</a > para obter detalhes sobre o modelo escolhido como o melhor.
# Model Based Clustering
library(mclust)
fit <- Mclust(mydata)
plot(fit) # plot results
summary(fit) # display the best model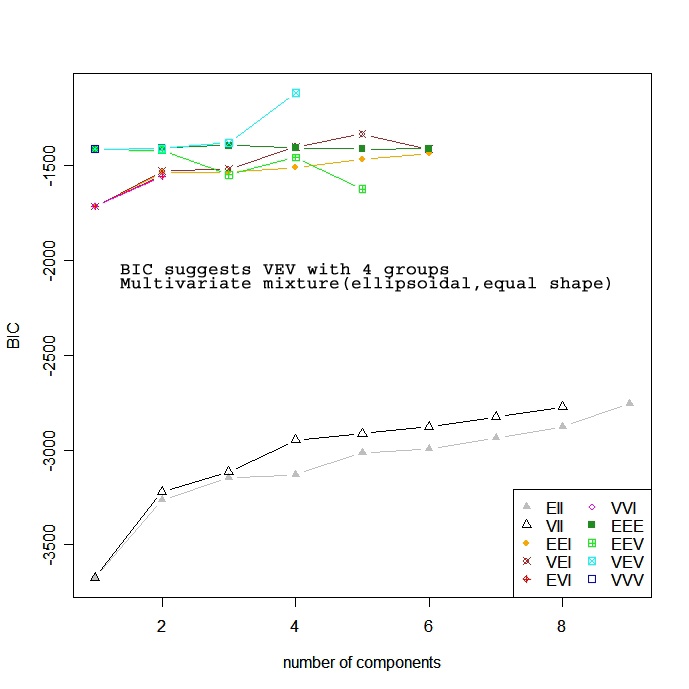
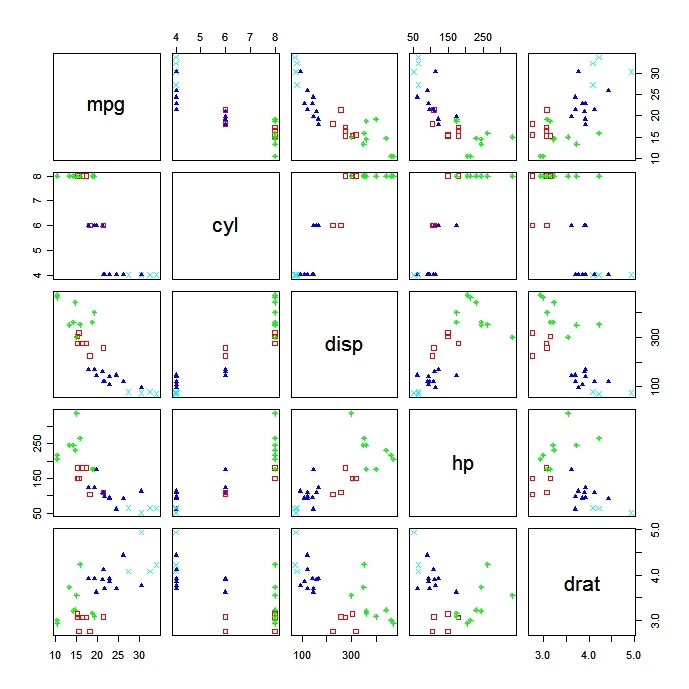
Plotagem de soluções de cluster
É sempre uma boa ideia você analisar os resultados do cluster.
# K-Means Clustering with 5 clusters
fit <- kmeans(mydata, 5)
# Cluster Plot against 1st 2 principal components
# vary parameters for most readable graph
library(cluster)
clusplot(mydata, fit$cluster, color=TRUE, shade=TRUE,
labels=2, lines=0)
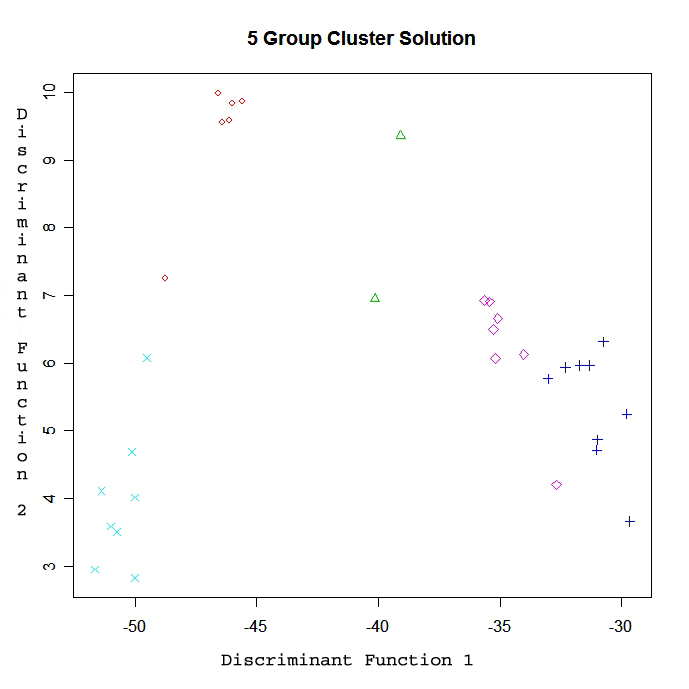
# Centroid Plot against 1st 2 discriminant functions
library(fpc)
plotcluster(mydata, fit$cluster)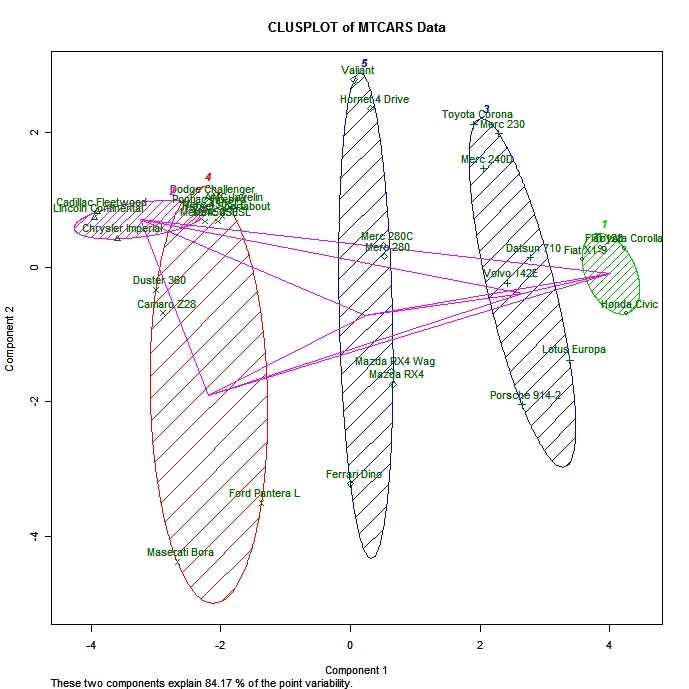
Validação de soluções de cluster
A função cluster.stats() do pacote fpc fornece um mecanismo para comparar a similaridade de duas soluções de cluster usando vários critérios de validação (coeficiente gama de Hubert, índice Dunn e índice rand corrigido)
# comparing 2 cluster solutions
library(fpc)
cluster.stats(d, fit1$cluster, fit2$cluster)em que d é uma matriz de distância entre objetos, e fit1$cluster e fit$cluste r são vetores inteiros que contêm resultados de classificação de dois agrupamentos diferentes dos mesmos dados.
Para praticar
Experimente o exercício de agrupamento neste curso de introdução ao aprendizado de máquina.</a>