Diskriminanzfunktionsanalyse in R
Das MASS-Paket enthält Funktionen zur Durchführung von linearen und quadratischen Diskriminanzfunktionen. Wenn keine Prioritätswahrscheinlichkeiten angegeben sind, wird jeweils von proportionalen Prioritätswahrscheinlichkeiten ausgegangen (d. h. die Prioritätswahrscheinlichkeiten basieren auf dem Stichprobenumfang). In den folgenden Beispielen sind die Kleinbuchstaben numerische Variablen und die Großbuchstaben kategorische Faktoren.
Lineare Diskriminanzfunktion
# Linear Discriminant Analysis with Jacknifed Prediction
library(MASS)
fit <- lda(G ~ x1 + x2 + x3, data=mydata,
na.action="na.omit", CV=TRUE)
fit # show resultsDer obige Code führt eine LDA durch, bei der fehlende Daten listenweise gelöscht werden. CV=TRUE generiert Jacknifed-Vorhersagen (d.h. eine Vorhersage wird ausgelassen). Der folgende Code prüft die Genauigkeit der Vorhersage.
# Assess the accuracy of the prediction
# percent correct for each category of G
ct <- table(mydata$G, fit$class)
diag(prop.table(ct, 1))
# total percent correct
sum(diag(prop.table(ct)))lda() gibt Diskriminanzfunktionen aus, die auf zentrierten (nicht standardisierten) Variablen basieren. Der "Anteil der Spur", der gedruckt wird, ist der Anteil der Varianz zwischen den Klassen, der durch aufeinanderfolgende Diskriminanzfunktionen erklärt wird. Es werden keine Signifikanztests durchgeführt. Siehe den Abschnitt über MANOVA für solche Tests.
Quadratische Diskriminanzfunktion
Um eine quadratische Diskriminanzfunktion zu erhalten, verwende qda( ) anstelle von lda( ). Die quadratische Diskriminanzfunktion setzt keine Homogenität der Varianz-Kovarianz-Matrizen voraus.
# Quadratic Discriminant Analysis with 3 groups applying
#
resubstitution prediction and equal prior probabilities.
library(MASS)
fit <- qda(G ~ x1 + x2 + x3 + x4, data=na.omit(mydata),
prior=c(1,1,1)/3))Beachte die alternative Möglichkeit, das listenweise Löschen fehlender Daten anzugeben. Die Re-Substitution (d.h. die Verwendung der gleichen Daten zur Ableitung der Funktionen und zur Bewertung ihrer Vorhersagegenauigkeit) ist die Standardmethode, es sei denn, CV=TRUE wird angegeben. Eine erneute Substitution wäre zu optimistisch.
Visualisierung der Ergebnisse
Mit dem folgenden Code kannst du jede Beobachtung im Raum der ersten beiden linearen Diskriminanzfunktionen darstellen. Punkte werden mit der Gruppen-ID gekennzeichnet.
# Scatter plot using the 1st two discriminant dimensions
plot(fit) # fit from lda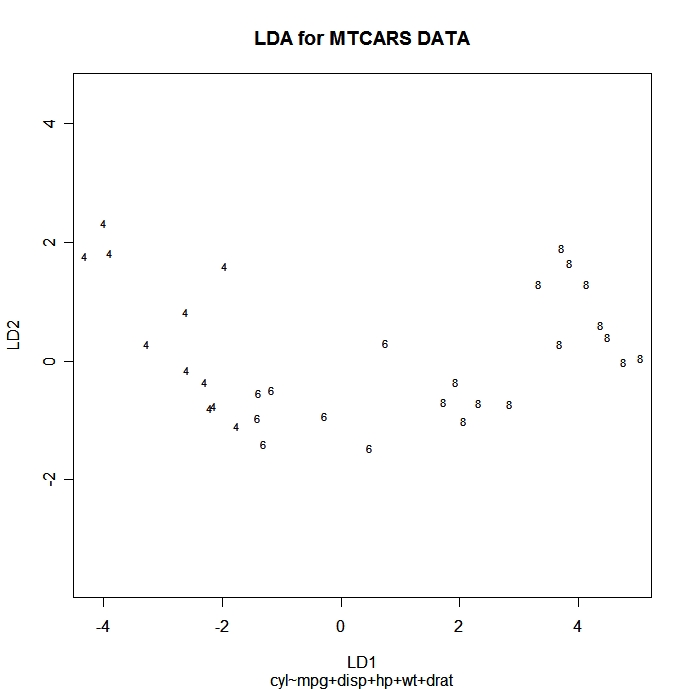
Der folgende Code zeigt Histogramme und Dichteplots für die Beobachtungen in jeder Gruppe auf der ersten linearen Diskriminanzdimension an. Für jede Gruppe gibt es ein Feld und sie erscheinen alle in einer Reihe auf demselben Diagramm.
# Panels of histograms and overlayed density plots
# for 1st discriminant function
plot(fit, dimen=1, type="both") # fit from lda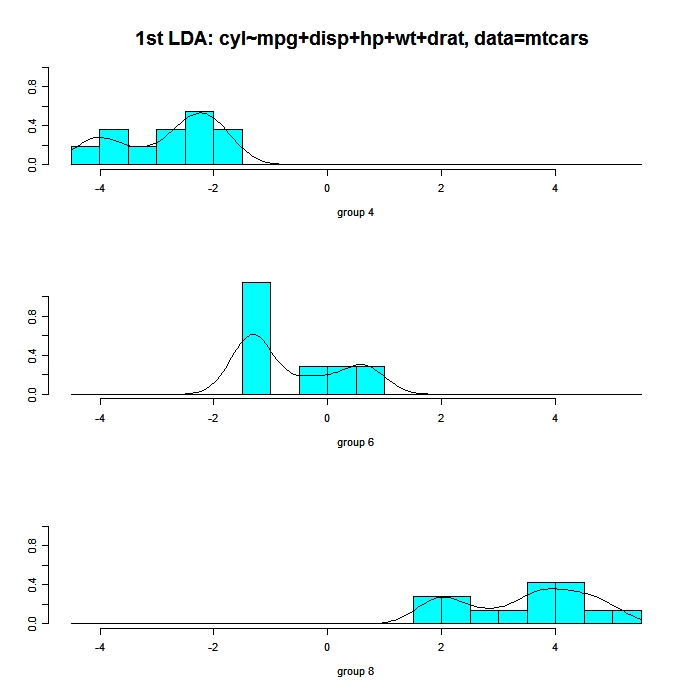
Die Funktion partimat( ) im klaR-Paket kann die Ergebnisse einer linearen oder quadratischen Klassifizierung 2 Variablen auf einmal anzeigen.
# Exploratory Graph for LDA or QDA
library(klaR)
partimat(G~x1+x2+x3,data=mydata,method="lda")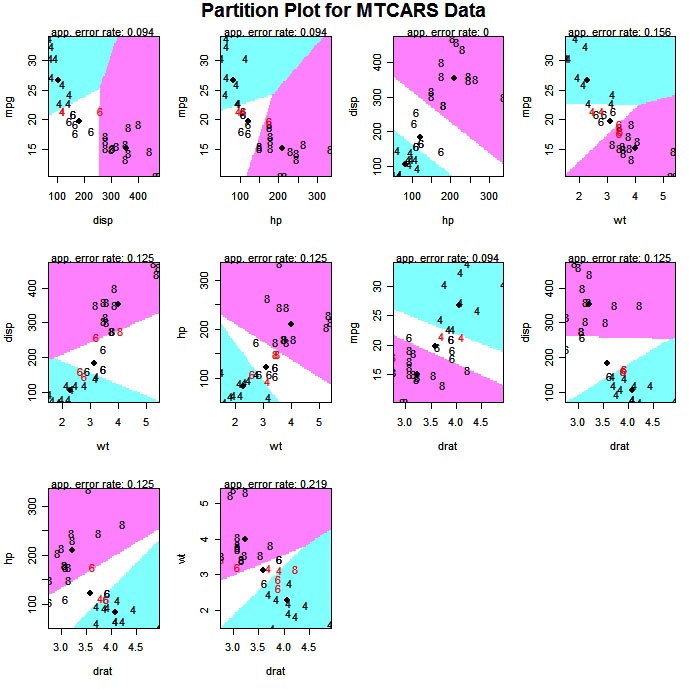
Du kannst auch eine Streudiagramm-Matrix mit Farbkodierung nach Gruppen erstellen.
# Scatterplot for 3 Group Problem
pairs(mydata[c("x1","x2","x3")], main="My Title ", pch=22,
bg=c("red", "yellow", "blue")[unclass(mydata$G)])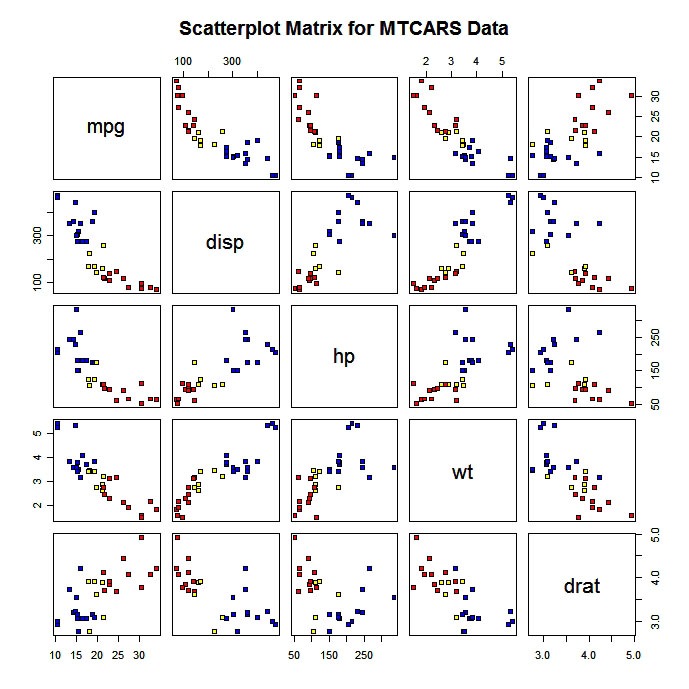
Test Annahmen
Siehe (M)ANOVA-Annahmen fürMethoden zur Bewertung der multivariaten Normalität und Homogenität von Kovarianzmatrizen.
Zum Üben
Um die Verbesserung von Vorhersagen zu üben, probiere den Kurs "Überwachtes Lernen in R" aus</a >