Análise de função discriminante no R
O pacote MASS contém funções para realizar análises de funções discriminantes lineares e quadráticas. A menos que as probabilidades anteriores sejam especificadas, cada uma assume probabilidades anteriores proporcionais (ou seja, as probabilidades anteriores são baseadas nos tamanhos das amostras). Nos exemplos abaixo, as letras minúsculas são variáveis numéricas e as letras maiúsculas são fatores categóricos.
Função discriminante linear
# Linear Discriminant Analysis with Jacknifed Prediction
library(MASS)
fit <- lda(G ~ x1 + x2 + x3, data=mydata,
na.action="na.omit", CV=TRUE)
fit # show resultsO código acima executa um LDA, usando a exclusão de dados ausentes em listas. CV=TRUE gera previsões jacknifed (ou seja, deixa um de fora). O código abaixo avalia a precisão da previsão.
# Assess the accuracy of the prediction
# percent correct for each category of G
ct <- table(mydata$G, fit$class)
diag(prop.table(ct, 1))
# total percent correct
sum(diag(prop.table(ct)))lda() imprime funções discriminantes com base em variáveis centralizadas (não padronizadas). A "proporção de traço" impressa é a proporção da variação entre classes que é explicada por funções discriminantes sucessivas. Não são produzidos testes de significância. Consulte a seção sobre MANOVA para obter informações sobre esses testes.
Função discriminante quadrática
Para obter uma função discriminante quadrática, use qda( ) em vez de lda( ). A função discriminante quadrática não pressupõe a homogeneidade das matrizes de variância-covariância.
# Quadratic Discriminant Analysis with 3 groups applying
#
resubstitution prediction and equal prior probabilities.
library(MASS)
fit <- qda(G ~ x1 + x2 + x3 + x4, data=na.omit(mydata),
prior=c(1,1,1)/3))Observe a maneira alternativa de especificar a exclusão em lista dos dados ausentes. A ressubstituição (usando os mesmos dados para derivar as funções e avaliar sua precisão de previsão) é o método padrão, a menos que CV=TRUE seja especificado. A re-substituição será excessivamente otimista.
Visualizando os resultados
Você pode plotar cada observação no espaço das duas primeiras funções discriminantes lineares usando o código a seguir. Os pontos são identificados com o ID do grupo.
# Scatter plot using the 1st two discriminant dimensions
plot(fit) # fit from lda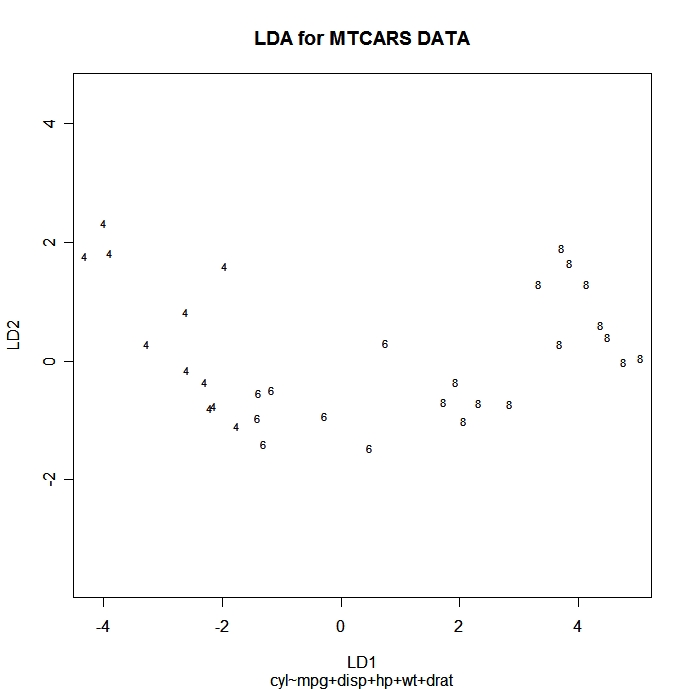
O código a seguir exibe histogramas e gráficos de densidade para as observações em cada grupo na primeira dimensão discriminante linear. Há um painel para cada grupo e todos eles aparecem alinhados no mesmo gráfico.
# Panels of histograms and overlayed density plots
# for 1st discriminant function
plot(fit, dimen=1, type="both") # fit from lda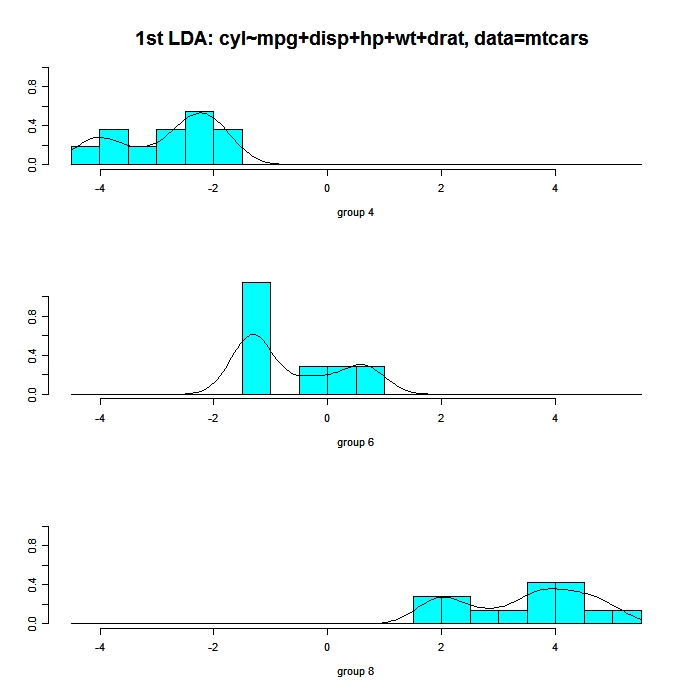
A função partimat( ) do pacote klaR pode exibir os resultados de uma classificação linear ou quadrática de 2 variáveis por vez.
# Exploratory Graph for LDA or QDA
library(klaR)
partimat(G~x1+x2+x3,data=mydata,method="lda")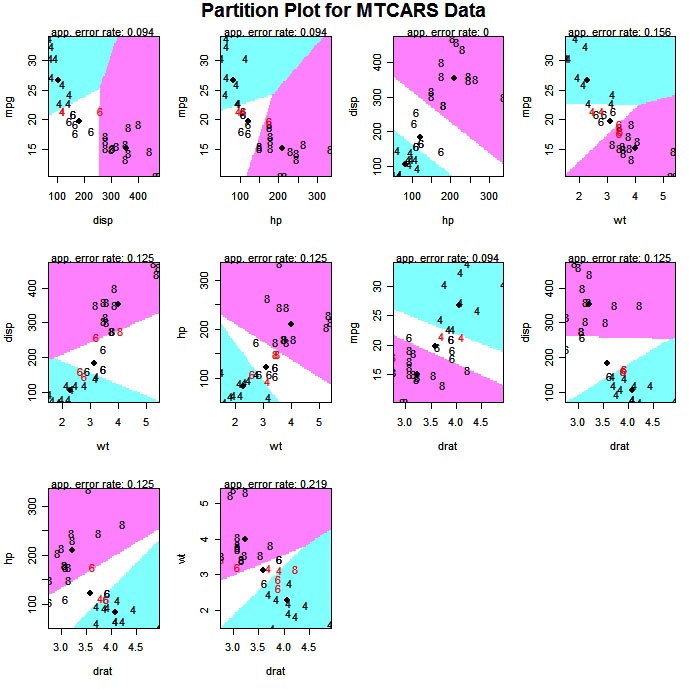
Você também pode produzir uma matriz de gráfico de dispersão com codificação de cores por grupo.
# Scatterplot for 3 Group Problem
pairs(mydata[c("x1","x2","x3")], main="My Title ", pch=22,
bg=c("red", "yellow", "blue")[unclass(mydata$G)])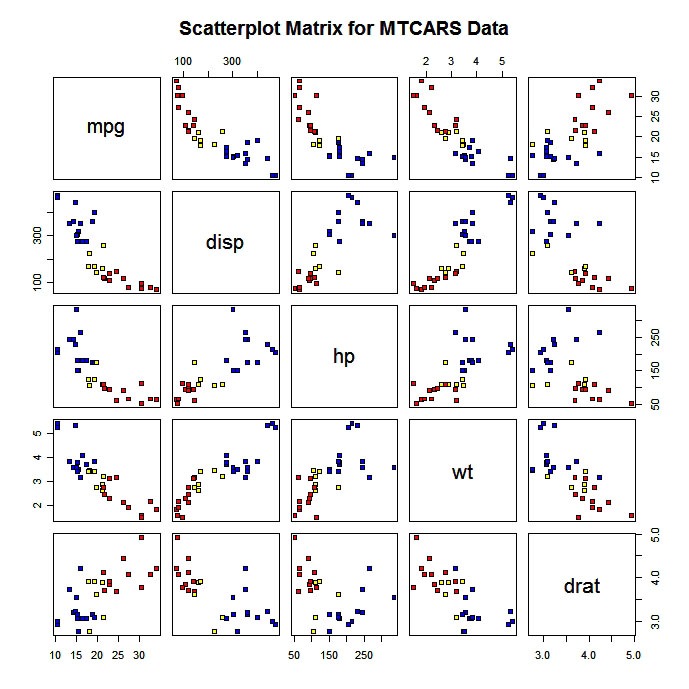
Premissas do teste
Consulte Pressupostos da (M)ANOVA para obtermétodos de avaliação da normalidade multivariada e da homogeneidade das matrizes de covariância.
Para praticar
Para praticar o aprimoramento das previsões, experimente o curso Aprendizado supervisionado em R</a >