Regressão múltipla (linear) no R
O R oferece suporte abrangente para regressão linear múltipla. Os tópicos abaixo são fornecidos em ordem crescente de complexidade.
Ajuste do modelo
# Multiple Linear Regression Example
fit <- lm(y ~ x1 + x2 + x3, data=mydata)
summary(fit) # show results# Other useful functions
coefficients(fit) # model coefficients
confint(fit, level=0.95) # CIs for model parameters
fitted(fit) # predicted values
residuals(fit) # residuals
anova(fit) # anova table
vcov(fit) # covariance matrix for model parameters
influence(fit) # regression diagnosticsGráficos de diagnóstico
Os gráficos de diagnóstico fornecem verificações de heterocedasticidade, normalidade e observações influentes.
# diagnostic plots
layout(matrix(c(1,2,3,4),2,2)) # optional 4 graphs/page
plot(fit)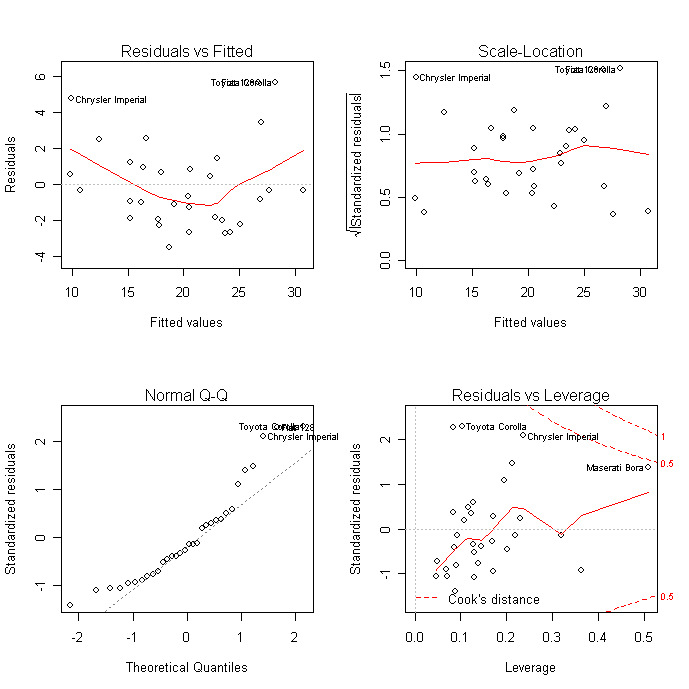
Para uma avaliação mais abrangente do ajuste do modelo, consulte os diagnósticos de regressão ou os exercícios em neste curso interativo</a > sobre Aprendizado Supervisionado em R: Regressão.
Comparação de modelos
Você pode comparar modelos aninhados com a função anova( ). O código a seguir fornece um teste simultâneo de que x3 e x4 são adicionados à previsão linear acima e além de x1 e x2.
# compare models
fit1 <- lm(y ~ x1 + x2 + x3 + x4, data=mydata)
fit2 <- lm(y ~ x1 + x2)
anova(fit1, fit2)Validação cruzada
Você pode fazer a validação cruzada K-Fold</a >usando a função cv.lm( ) no pacote DAAG.
# K-fold cross-validation
library(DAAG)
cv.lm(df=mydata, fit, m=3) # 3 fold cross-validationSome o MSE para cada dobra, divida pelo número de observações e tire a raiz quadrada para obter o erro padrão de estimativa com validação cruzada.
Você pode avaliar a redução de R2 por meio da validação cruzada K-fold. Usando a função crossval() do pacote bootstrap</a ></strong >, faça o seguinte:
# Assessing R2 shrinkage using 10-Fold Cross-Validation
fit <- lm(y~x1+x2+x3,data=mydata)
library(bootstrap)
# define functions
theta.fit <- function(x,y){lsfit(x,y)}
theta.predict <- function(fit,x){cbind(1,x)%*%fit$coef}
# matrix of predictors
X
<- as.matrix(mydata[c("x1","x2","x3")])
# vector of predicted values
y <- as.matrix(mydata[c("y")])
results <- crossval(X,y,theta.fit,theta.predict,ngroup=10)
cor(y, fit$fitted.values)**2 # raw R2
cor(y,results$cv.fit)**2 # cross-validated R2Seleção de variáveis
A seleção de um subconjunto de variáveis preditoras de um conjunto maior (por exemplo, seleção por etapas) é um tópico controverso. Você pode executar a seleção por etapas (para frente, para trás, ambas) usando a função stepAIC( ) do pacote MASS. stepAIC( ) executa a seleção do modelo por etapas pelo AIC exato.
# Stepwise Regression
library(MASS)
fit <- lm(y~x1+x2+x3,data=mydata)
step <- stepAIC(fit, direction="both")
step$anova # display resultsComo alternativa, você pode executar a regressão de todos os subconjuntos usando a função leaps( ) do pacote leaps. No código a seguir, nbest indica o número de subconjuntos de cada tamanho a ser relatado. Aqui, os dez melhores modelos serão relatados para cada tamanho de subconjunto (1 preditor, 2 preditores, etc.).
# All Subsets Regression
library(leaps)
attach(mydata)
leaps<-regsubsets(y~x1+x2+x3+x4,data=mydata,nbest=10)
# view results
summary(leaps)
# plot a table of models showing variables in each model.
#
models are ordered by the selection statistic.
plot(leaps,scale="r2")
# plot statistic by subset size
library(car)
subsets(leaps, statistic="rsq")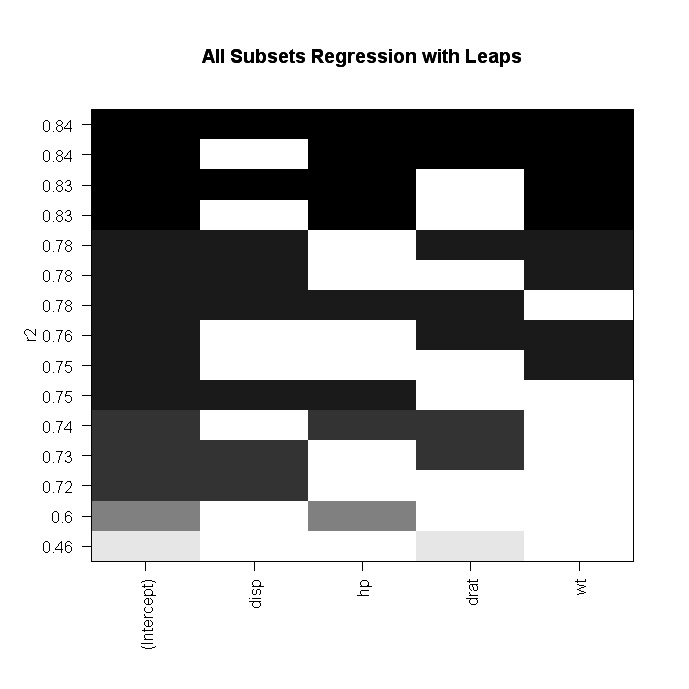
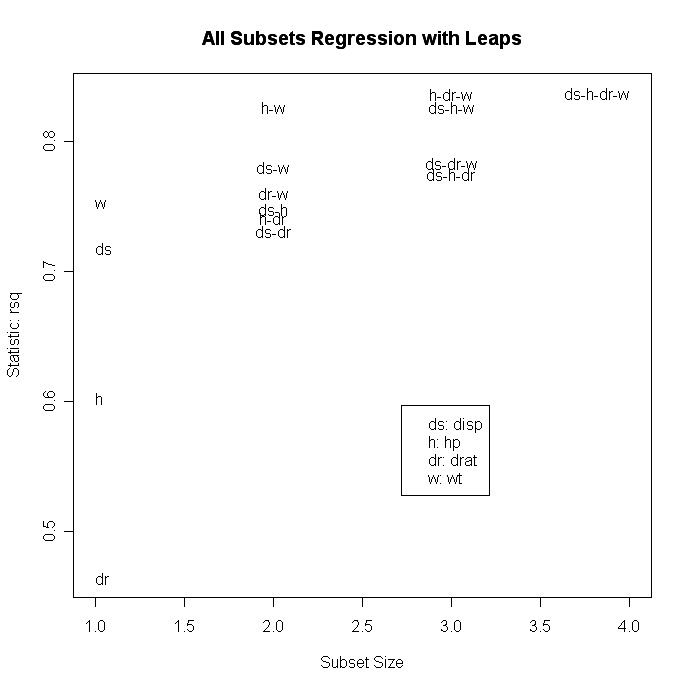
Outras opções para plot( ) são bic, Cp e adjr2. Outras opções para plotagem comsubset( ) são bic, cp, adjr2 e rss.
Importância relativa
# Calculate Relative Importance for Each Predictor
library(relaimpo)
calc.relimp(fit,type=c("lmg","last","first","pratt"),
rela=TRUE)
# Bootstrap Measures of Relative Importance (1000 samples)
boot <- boot.relimp(fit, b = 1000, type = c("lmg",
"last", "first", "pratt"), rank = TRUE,
diff = TRUE, rela = TRUE)
booteval.relimp(boot) # print result
plot(booteval.relimp(boot,sort=TRUE)) # plot result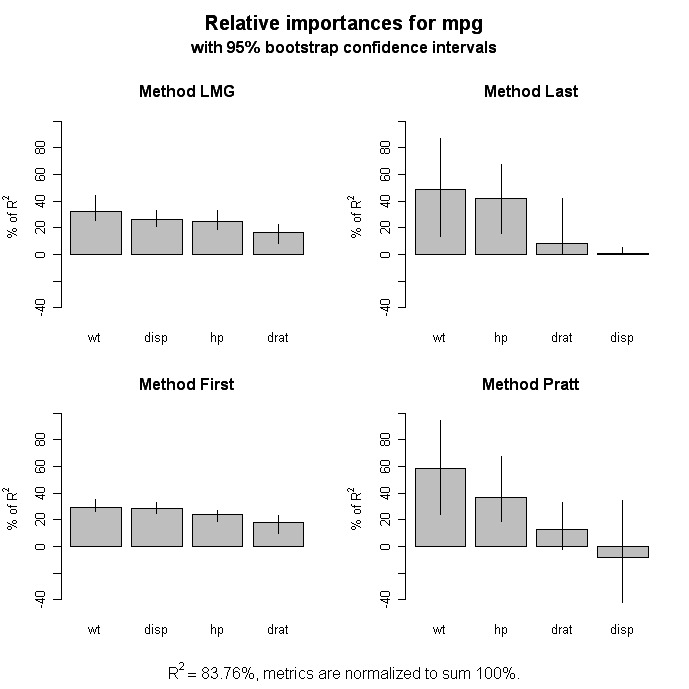
Aprimoramentos gráficos
O pacote car oferece uma ampla variedade de gráficos para regressão, incluindo gráficos de variáveis adicionadas, diagnóstico aprimorado e gráficos de dispersão.
Indo além
Regressão não linear
O pacote nls fornece funções para regressão não linear. Consulte o site de John Fox Nonlinear Regression and Nonlinear Least Squares</a > para obter uma visão geral. Huet e colegas Statistical Tools for Nonlinear Regression (Ferramentas estatísticas para regressão não linear): A Practical Guide with S-PLUS and R Examples</a > é um valioso livro de referência.
Regressão robusta
Há muitas funções no R para ajudar na regressão robusta. Por exemplo, você pode realizar uma regressão robusta com a função rlm( ) do pacote MASS. De John Fox (quem mais?) Robust Regression</a > fornece uma boa visão geral inicial. O site da UCLA Statistical Computing tem Exemplos de regressão robusta</a > .
O pacote robust fornece uma biblioteca abrangente de métodos robustos, incluindo regressão. O pacote robustbase</a > também fornece estatísticas robustas básicas, incluindo métodos de seleção de modelos. E David Olive forneceu uma análise on-line detalhada de Applied Robust Statistics</a > com um exemplo de código R .