Avaliando as suposições de testes clássicos no R
Nos procedimentos paramétricos clássicos, geralmente assumimos a normalidade e a variância constante do termo de erro do modelo. Os métodos para explorar essas premissas em uma estrutura ANOVA/ANCOVA/MANOVA são discutidos aqui. Os diagnósticos de regressão são abordados na regressão linear múltipla.
Excedentes
Como os valores discrepantes podem afetar gravemente a normalidade e a homogeneidade da variância, os métodos para detectar observações discrepantes são descritos primeiro.
# Detect Outliers in the MTCARS Data
library(mvoutlier)
outliers <-
aq.plot(mtcars[c("mpg","disp","hp","drat","wt","qsec")])
outliers # show list of outliers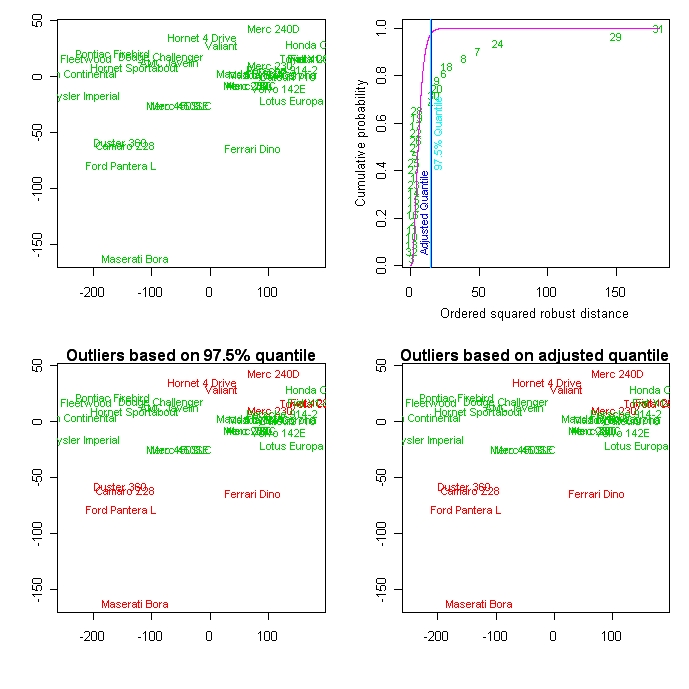
Normalidade univariada
Você pode avaliar a normalidade de uma variável usando um gráfico Q-Q.
# Q-Q Plot for variable MPG
attach(mtcars)
qqnorm(mpg)
qqline(mpg)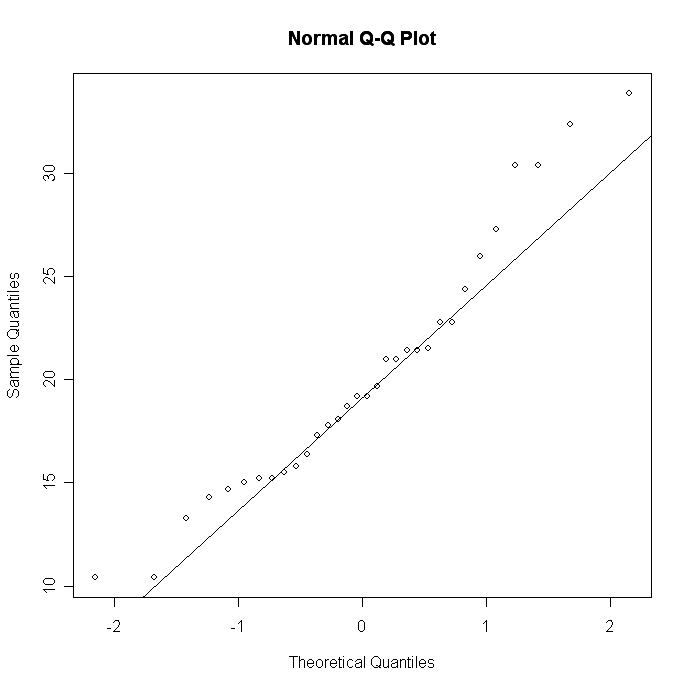
Desvios significativos da linha sugerem violações da normalidade.
Você também pode executar um teste de normalidade Shapiro-Wilk com a função shapiro.test(x), em que x é um vetor numérico. Funções adicionais para testar a normalidade estão disponíveis no pacote nortest</a > .
Normalidade multivariada
A MANOVA pressupõe normalidade multivariada. A função mshapiro.test( ) no pacote mvnormtest</a > produz o teste de Shapiro-Wilk para normalidade multivariada. A entrada deve ser uma matriz numérica.
# Test Multivariate Normality
mshapiro.test(M)Se tivermos um vetor aleatório normal multivariado p x 1, a distância de Mahalanobis ao quadrado entre x e μ será distribuída como qui-quadrado com p graus de liberdade. Podemos usar esse fato para construir um gráfico Q-Q para avaliar a normalidade multivariada.
# Graphical Assessment of Multivariate Normality
x <- as.matrix(mydata) # n x p numeric matrix
center <- colMeans(x) # centroid
n <- nrow(x); p <- ncol(x); cov <- cov(x);
d <-
mahalanobis(x,center,cov) # distances
qqplot(qchisq(ppoints(n),df=p),d,
main="QQ Plot Assessing Multivariate Normality",
ylab="Mahalanobis D2")
abline(a=0,b=1)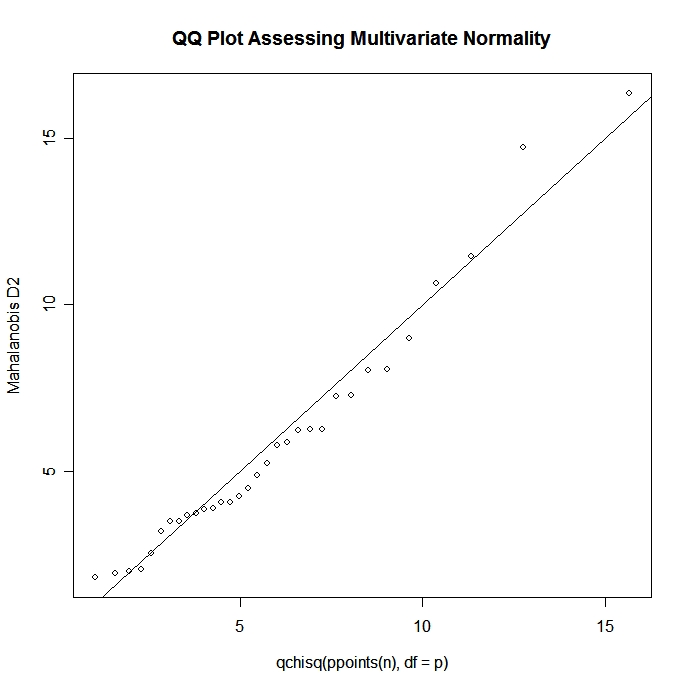
Homogeneidade de variâncias
A função bartlett.test( ) fornece um teste de amostra K paramétrico da igualdade de variâncias. A função fligner.test( ) fornece um teste não paramétrico do mesmo. Nos exemplos a seguir, y é uma variável numérica e G é a variável de agrupamento.
# Bartlett Test of Homogeneity of Variances
bartlett.test(y~G, data=mydata)
# Figner-Killeen Test of Homogeneity of Variances
fligner.test(y~G, data=mydata)A função hovPlot( ) do pacote HH fornece um teste gráfico de homogeneidade de variâncias com base em Brown-Forsyth. No exemplo a seguir, y é numérico e G é um fator de agrupamento. Observe que G deve ser do tipo fator.
# Homogeneity of Variance Plot
library(HH)
hov(y~G, data=mydata)
hovPlot(y~G,data=mydata)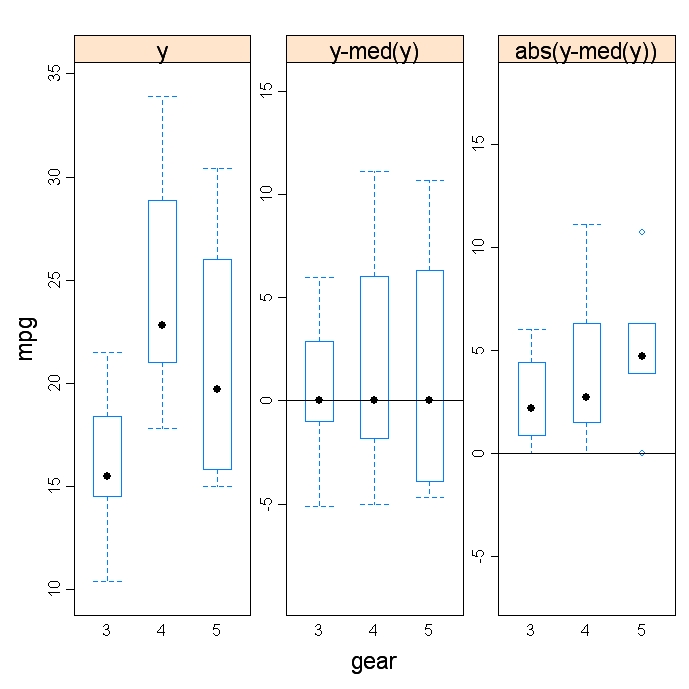
Homogeneidade das matrizes de covariância
A MANOVA e a LDF pressupõem a homogeneidade das matrizes de variância-covariância. Normalmente, a suposição é testada com o Box's M. Infelizmente, o teste é muito sensível a violações da normalidade, levando à rejeição na maioria dos casos típicos. O M de Box está disponível por meio da função boxM no pacote biotools</a > .
Para praticar
Faça o curso Introduction to Statistics in R</a > para aprimorar ainda mais suas habilidades estatísticas .