Componentes principais e análise de fatores no R
Esta seção aborda os componentes principais e a análise de fatores. O último inclui métodos exploratórios e confirmatórios.
Componentes principais
A função princomp( ) produz uma análise de componente principal não rotacionada.
# Pricipal Components Analysis
# entering raw data and extracting PCs
#
from the correlation matrix
fit <- princomp(mydata, cor=TRUE)
summary(fit) # print variance accounted for
loadings(fit) # pc loadings
plot(fit,type="lines") # scree plot
fit$scores # the principal components
biplot(fit)Use cor=FALSE para basear os componentes principais na matriz de covariância. Use a opção covmat= para inserir uma matriz de correlação ou covariância diretamente. Se você inserir uma matriz de covariância, inclua a opção n.obs=.
A função principal( ) do pacote psych pode ser usada para extrair e girar os componentes principais.
# Varimax Rotated Principal Components
# retaining 5 components
library(psych)
fit <- principal(mydata, nfactors=5, rotate="varimax")
fit # print resultsmydata pode ser uma matriz de dados brutos ou uma matriz de covariância. A exclusão par a par dos dados ausentes é usada. A opção de rotação pode ser "none", "varimax", "quatimax", "promax", "oblimin", "simplimax" ou "cluster"
Análise fatorial exploratória
A função factanal( ) produz uma análise de fator de máxima verossimilhança.
# Maximum Likelihood Factor Analysis
# entering raw data and extracting 3 factors,
#
with varimax rotation
fit <- factanal(mydata, 3, rotation="varimax")
print(fit, digits=2, cutoff=.3, sort=TRUE)
# plot factor 1 by factor 2
load <- fit$loadings[,1:2]
plot(load,type="n") # set up plot
text(load,labels=names(mydata),cex=.7) # add variable namesAs opções de rotation= incluem "varimax", "promax" e "none". Adicione a opção scores="regression" ou "Bartlett" para produzir pontuações de fator. Use a opção covmat= para inserir uma matriz de correlação ou covariância diretamente. Se você inserir uma matriz de covariância, inclua a opção n.obs=.
A função factor.pa( ) do pacote psych oferece várias funções relacionadas à análise de fatores, incluindo a fatoração do eixo principal.
# Principal Axis Factor Analysis
library(psych)
fit <- factor.pa(mydata, nfactors=3, rotation="varimax")
fit # print resultsmydata pode ser uma matriz de dados brutos ou uma matriz de covariância. É usada a exclusão par a par dos dados ausentes. A rotação pode ser "varimax" ou "promax".
Determinação do número de fatores a serem extraídos
Uma decisão crucial na análise exploratória de fatores é quantos fatores devem ser extraídos. O pacoteThenFactors oferece um conjunto de funções para ajudar nessa decisão. Detalhes sobre essa metodologia podem ser encontrados em uma apresentação em PowerPoint feita por Raiche, Riopel e Blais. É claro que, para ser útil, qualquer solução de fator deve ser interpretável.
# Determine Number of Factors to Extract
library(nFactors)
ev <- eigen(cor(mydata)) # get eigenvalues
ap <- parallel(subject=nrow(mydata),var=ncol(mydata),
rep=100,cent=.05)
nS <- nScree(x=ev$values, aparallel=ap$eigen$qevpea)
plotnScree(nS)Indo além
O pacote FactoMineR oferece um grande número de funções adicionais para a análise exploratória de fatores. Isso inclui o uso de variáveis quantitativas e qualitativas, bem como a inclusão de variáveis e observações complementares. Aqui está um exemplo dos tipos de gráficos que você pode criar com esse pacote.
# PCA Variable Factor Map
library(FactoMineR)
result <- PCA(mydata) # graphs generated automaticallyO pacote GPARotation oferece uma grande variedade de opções de rotação além de varimax e promax.
Modelagem de equações estruturais
A análise fatorial confirmatória (CFA) é um subconjunto da metodologia de modelagem de equações estruturais (SEM), que é muito mais ampla. O SEM é fornecido no R por meio do pacote sem. Os modelos são inseridos por meio da especificação RAM (semelhante ao PROC CALIS no SAS). Embora o Sem seja um pacote abrangente, minha recomendação é que, se você estiver fazendo um trabalho significativo de SEM, adquira uma cópia do AMOS. Ele pode ser muito mais fácil de usar e criar resultados mais atraentes e prontos para publicação. Dito isso, aqui está um exemplo de CFA usando sem.
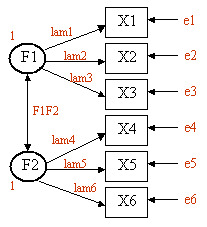
Suponha que tenhamos seis variáveis observadas (X1, X2, ..., X6). Nossa hipótese é que há dois fatores latentes não observados (F1, F2) que sustentam as variáveis observadas, conforme descrito neste diagrama. X1, X2 e X3 carregam em F1 (com cargas lam1, lam2 e lam3). X4, X5 e X6 carregam em F2 (com cargas lam4, lam5 e lam6). A seta de duas pontas indica a covariância entre os dois fatores latentes (F1F2). e1 a e6 representam as variâncias residuais (variância nas variáveis observadas não consideradas pelos dois fatores latentes). Definimos as variações de F1 e F2 iguais a um para que os parâmetros tenham uma escala. Isso resultará em F1F2, que representa a correlação entre os dois fatores latentes.
Para sem, precisamos da matriz de covariância das variáveis observadas - portanto, a instrução cov( ) no código abaixo. O modelo CFA é especificado usando a função specify.model( ). O formato é especificação de seta, nome do parâmetro, valor inicial. A escolha de um valor inicial de NA diz ao programa para escolher um valor inicial em vez de você mesmo fornecer um. Observe que a variância de F1 e F2 é fixada em 1 (NA na segunda coluna). A linha em branco é necessária para encerrar a especificação da RAM.
# Simple CFA Model
library(sem)
mydata.cov <- cov(mydata)
model.mydata <- specify.model()
F1 -> X1, lam1, NA
F1 -> X2, lam2, NA
F1 -> X3, lam3, NA
F2 -> X4, lam4, NA
F2 -> X5, lam5, NA
F2 -> X6, lam6, NA
X1 <-> X1, e1, NA
X2 <-> X2, e2, NA
X3 <-> X3, e3, NA
X4 <-> X4, e4, NA
X5 <-> X5, e5, NA
X6 <-> X6, e6, NA
F1 <-> F1, NA, 1
F2 <-> F2, NA, 1
F1 <-> F2, F1F2, NA
mydata.sem <- sem(model.mydata, mydata.cov, nrow(mydata))
# print results (fit indices, paramters, hypothesis tests)
summary(mydata.sem)
# print standardized coefficients (loadings)
std.coef(mydata.sem)Você pode usar a função boot.sem( ) para fazer o bootstrap do modelo de equação estrutural. Consulte help(boot.sem) para obter detalhes. Além disso, a função mod.indices( ) produzirá índices de modificação. O uso de índices de modificação para melhorar o ajuste do modelo por meio da reespecificação dos parâmetros faz com que você passe de uma análise confirmatória para uma exploratória.
Para obter mais informações sobre o sem, consulte Structural Equation Modeling with the sem Package in R, de John Fox.
Para praticar
Para praticar o aprimoramento das previsões, experimente o curso Aprendizado supervisionado em R





